本文内容速览:

作物驯化是人类文明起始的里程碑事件。作物驯化的本质是人类对具有潜力的野生植物进行选择,从大量的自然变异中保留优异的等位基因,使得植物性状发生改变以适合农业生产,满足人类需求。野生物种在被驯化的过程中会逐步发生不同程度、多样化的表型变异,转变成介于野生和完全驯化之间的半驯化物种和完全驯化的物种。
完全驯化的物种可以理解为人类社会现阶段的主要农作物栽培种,它们适应了人工栽培的环境,相比于其野生祖先种具有理想的株型结构、高产、高品质等表型。然而,栽培种也并非十全十美,其中最大的一个缺陷就是遗传多样性丢失,这对于栽培作物产量和品质的进一步改良十分不利。
针对这类现象,培育新型作物品种成为了育种学家的研究方向。野生物种相较于栽培品种普遍具有较强的环境适应力,而且次生代谢较旺盛,能够产生更多的风味和营养物质。以未驯化或半驯化的野生物种为底盘材料进行作物品种创新,是增加作物多样性的有效途径。
接下来跟着小远一起去看看,那些通过驯化野生物种培育新型作物的科研工作吧!
科研工作者将番茄作为研究野生物种快速驯化的首要选择,主要由于番茄有16个野生种或近缘野生种,在耐逆境、果实品质、风味和营养等方面具备优良的种质特性,于是大家不约而同地将眼光聚焦于这一物种。
2018年10月,中国科学院遗传与发育生物学研究所许操课题组联合高彩霞课题组在
Nature Biotechnology
杂志上发表了一篇题为“Domestication of wild tomato is accelerated by genome editing”的研究论文。该研究选用天然耐盐碱和抗细菌疮痂病的野生醋栗番茄(
Solanum pimpinellifolium
)为底盘材料,利用基因编辑技术精准靶向多个产量和品质性状控制基因的编码区及调控区,在不牺牲其对盐碱和疮痂病天然抗性的前提下,将产量和品质性状精准地导入了野生番茄,加速了野生番茄的人工驯化。
根据不同基因的功能结构特点,研究团队使用多靶点CRISPR/Cas9载体系统,精准靶向开花光周期敏感性、株型和果实同步成熟控制基因
SP
和
SP5G
的编码区、果实大小控制基因
SlCLV3
、
SlWUS
的顺式调控元件和维生素C合成酶基因
SlGGP1
的上游开放阅读框,获得了140个独立的基因编辑株系(图1)。

图1 通过CRISPR/Cas9技术加速野生番茄驯化的示意图(Li et al., 2018)。
后代群体的基因型和表型鉴定表明,基因编辑消除了野生番茄开花的光周期敏感性,突破了栽培种的地理范围限制,实现了野生植物驯化的第一步。同时成功将株型转变为紧凑型并使果实变大(图2、图3)。
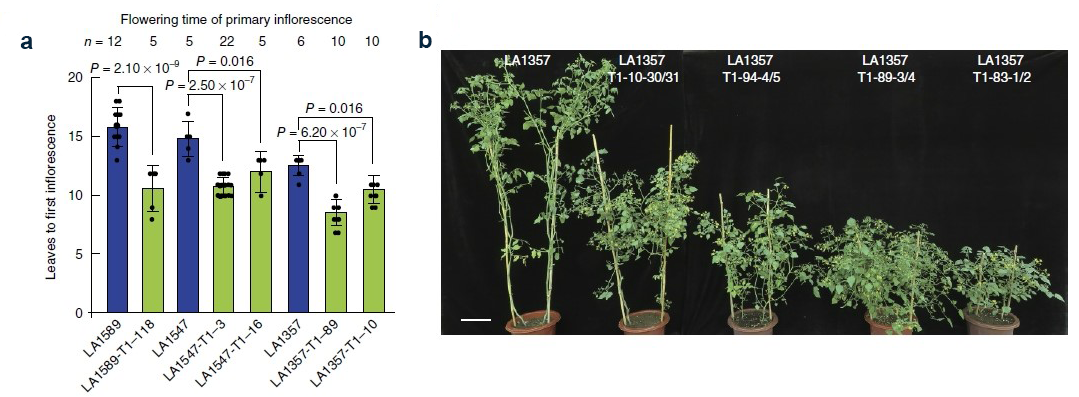
图2 基因编辑株系T1代开花时间及株型情况(Li et al., 2018)。(a)野生型LA1589、LA1547及LA1357和基因编辑株系T1代植物开花时间比较;(b)野生型LA1357和基因编辑株系T1代株型比较。
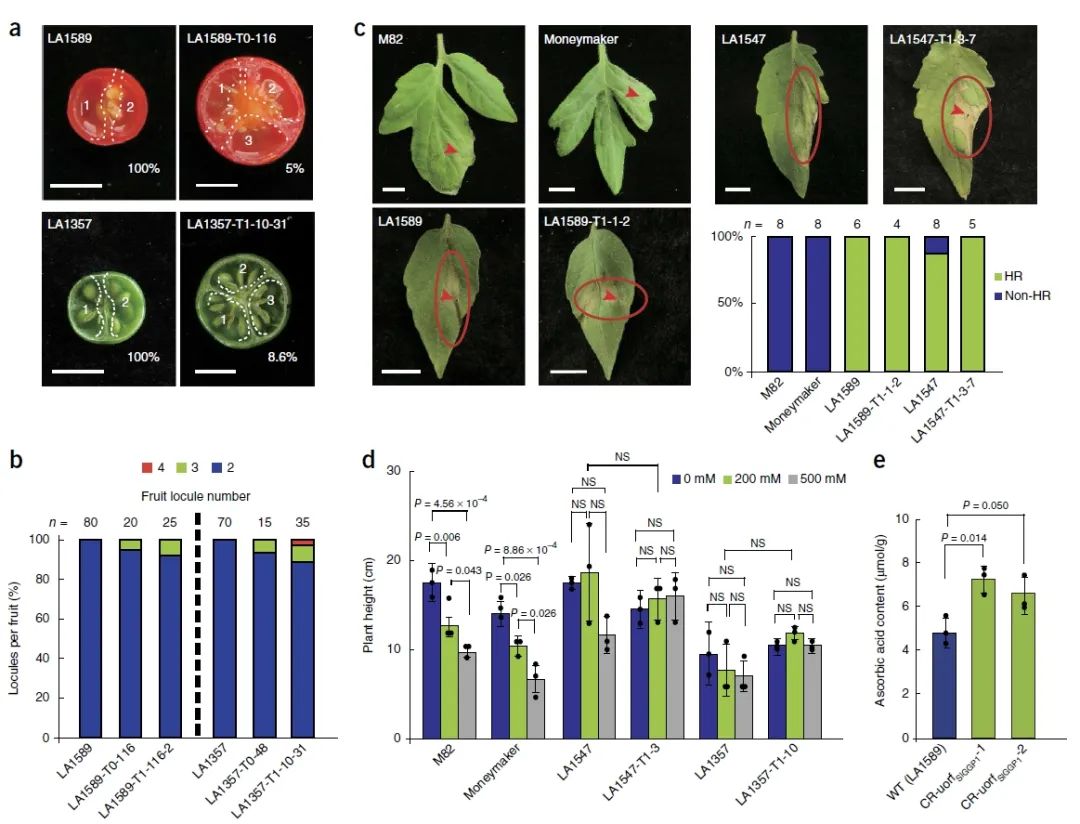
图3 基因编辑株系T1代果实大小、胁迫耐受性及营养成分情况(Li et al., 2018)。(a、b)野生型LA1589和LA1357与基因编辑植株T0代及T1代果实子房数量比较;(c、d)野生型与基因编辑株系抗细菌疮痂病性(c)及耐盐性(d)比较;(e)野生型LA1589与基因编辑植株T0代叶面抗坏血酸含量比较。
2018年10月,巴西圣保罗大学科研团队联合德国明斯特大学科研团队在
Nature Biotechnology
杂志上发表了一篇题为“
De novo
domestication of wild tomato using genome editing”的研究论文。同样以野生醋栗番茄(
Solanum pimpinellifolium
)为底盘材料,通过编辑对产量和生产率有重要影响的6个基因,实现了对野生醋栗番茄的从头驯化。
在基因编辑策略和基因的选择上,与前面的科研团队有所不同。该团队利用基因编辑技术对番茄株型相关基因
SELF PRUNING
、果实形状相关基因
OVATE
、果实大小相关基因
FASCIATED
和
FRUNIT WEIGHT
、果实数量相关基因
MULTIFLORA
和营养品质相关基因
LYCOPENE BETA CYCLASE
进行编辑。与野生型相比,突变体果实大小增加了3倍,数量增加了10倍,其中番茄红素含量比野生番茄增加了1倍,而比现在广泛栽培的番茄品种增加了5倍。

图4 从头驯化的野生醋栗番茄的形态和果实形状(Zsögön et al., 2018)。(a)基因编辑植株中
SELF PRUNING
的突变形式;(b-g)野生型与基因编辑植株在株型(b、c)、营养枝的生长情况(d、e)、株高(f)以及合轴分枝指数(g)上的比较;(h)基因编辑植株中
OVATE
的突变形式;(i-m)野生型与基因编辑植株在果实形状(i、j、k、l)及果实长宽比(m)上的比较。
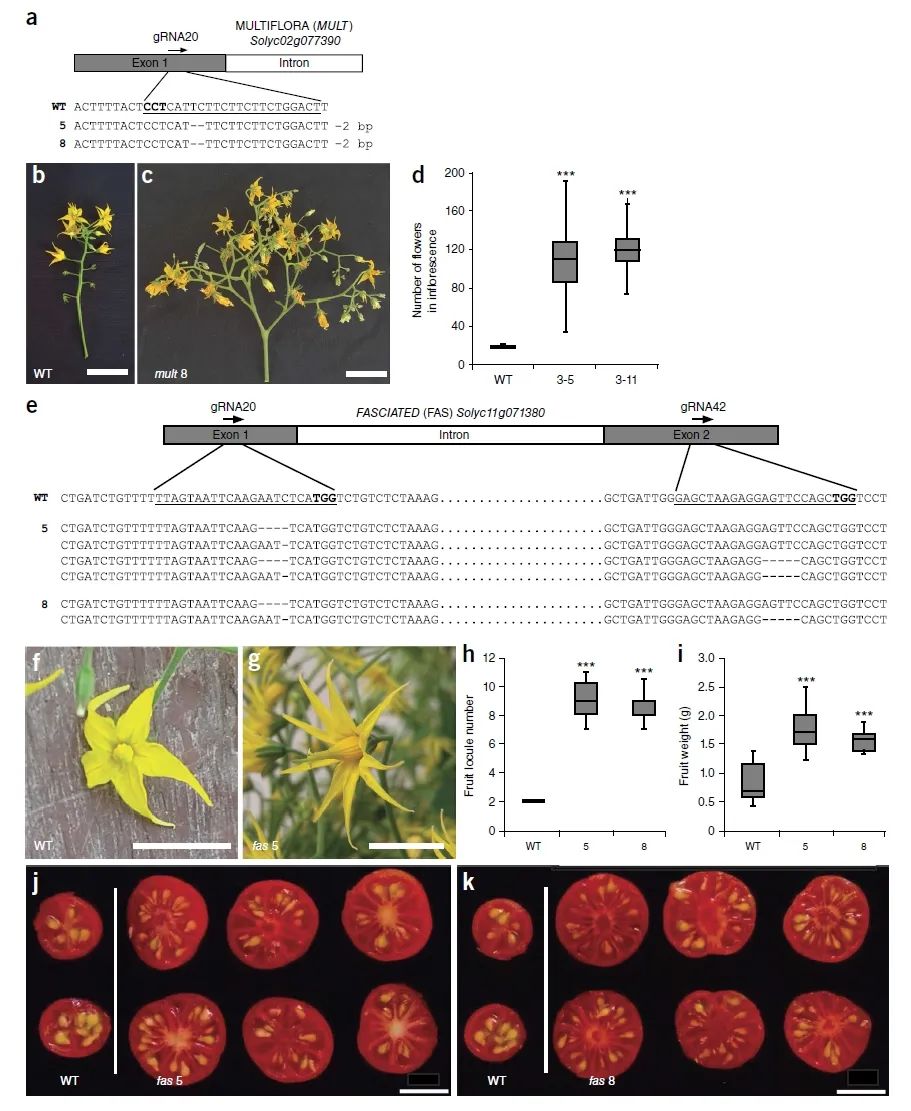
图5 从头驯化的野生醋栗番茄果实的数量和大小(Zsögön et al., 2018)。(a)基因编辑植株中
MULTIFLORA
的突变形式;(b-d)野生型与基因编辑植株在花序数量上的比较;(e)基因编辑植株中
FASCIATED
的突变形式;(f-k)野生型与基因编辑植株在花瓣数量(f、g)、果实子房数量(h、j、k)及果实重量(i)上的比较。
图6 从头驯化的野生醋栗番茄果实的营养含量(Zsögön et al., 2018)。(a)基因编辑植株中
LYCOPENE BETA-CYCLASE
的突变形式;(b-g)野生型与基因编辑植株因为番茄红素积累的不同在花药颜色(b、c)、花药番茄红素浓度(d)、果实颜色(e、f)及成熟果实番茄红素浓度(g)上的比较。
孤生作物指相对于水稻、小麦、玉米等常见的经济作物,目前在全世界范围内没有广泛种植和贸易的一类作物。灯笼果就是一种孤生作物,为茄科洋酸浆属植物,与番茄具有较远的亲缘关系。
2018年10月,纽约冷泉港实验室联合康奈尔大学科研团队在
Nature Plants
杂志上发表了一篇题为“Rapid improvement of domestication traits in an orphan crop by genome editing”的文章。该研究选用了灯笼果(
Physalis pruinosa
)为底盘材料,通过编辑番茄驯化相关基因的直系同源基因,改善了野生种的落粒性及果实品质,提高了产量,完成了灯笼果的快速驯化。
研究团队选择了株型相关基因
SELFPRUNING
、果实落粒性相关基因
SELF-PRUNING 5G
(图7)
以及果实大小相关基因
CLV1
(图8)
这三个基因进行驯化和改良,与野生型相比,基因编辑植株株型更加紧凑,果实数量增多,果实增重达24%同时果实落粒性降低。
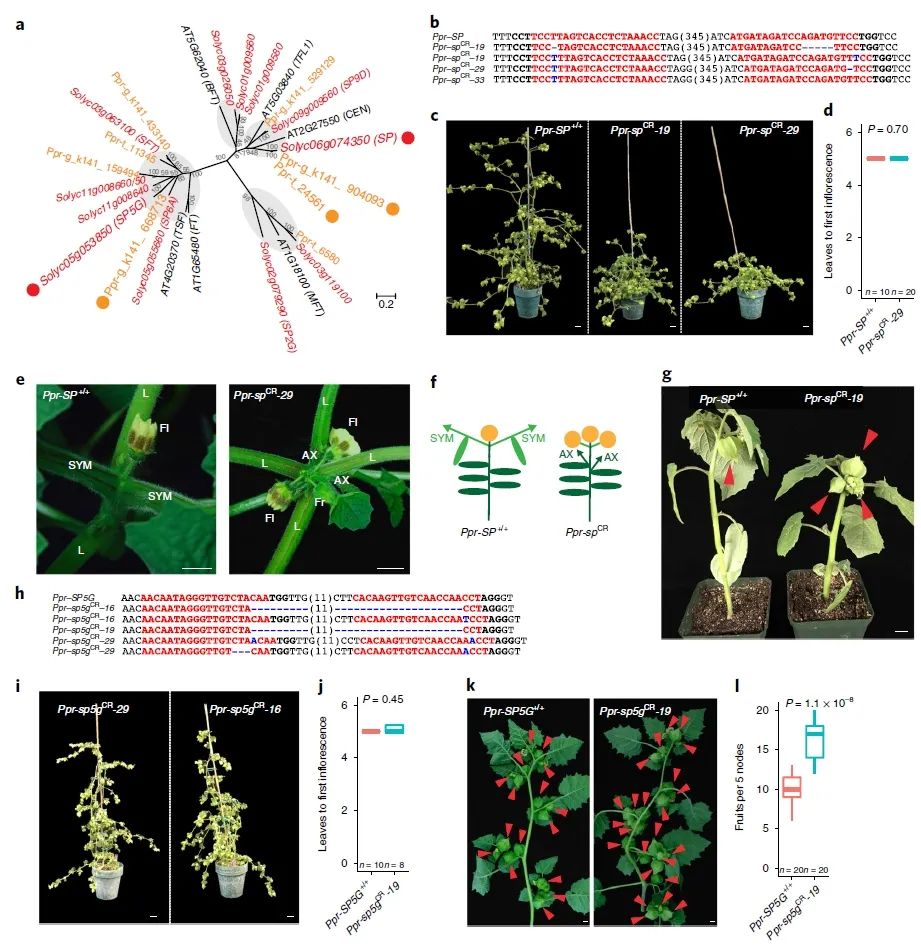
图7 CRISPR-Cas9靶向
P. pruinosa
驯化和改良相关基因(Lemmon et al., 2018)。(a)番茄、拟南芥及灯笼果
SELF-PRUNIN
G
(
CETS
)
基因家族系统发育树;(b)基因编辑植株中
SP
的突变形式;(c-g)基因编辑植株与野生型植株在株型(c)、初梢开花时间(d)、坐果数量(e、f、g)的比较;(h)基因编辑植株中
SP5G
的突变形式;(i)T1代纯合基因编辑植株;(j)基因编辑植株与野生型植株在初梢开花时间的比较;(k)基因编辑植株比野生型开花及果实发育时间更早;(l)基因编辑植株与野生型植株5个连续共生枝条节点上果实数量比较。
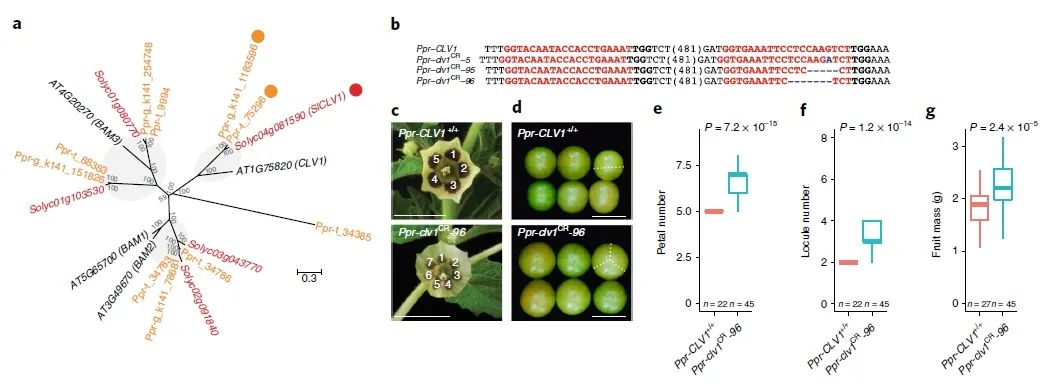
图8 CRISPR-Cas9靶向
P. pruinosa
驯化和改良相关基因
CLV1
(Lemmon et al., 2018)。(a)番茄、拟南芥及灯笼果
CLV1
基因家族系统发育树;(b)基因编辑植株中
CLV1
的突变形式;(c-g)野生型与基因编辑植株在花瓣数(c、e)、果实子房数(d、f)、果实重量(g)的比较。
以上三个案例让我们了解到,野生物种从头驯化对于培育抗逆境胁迫的作物来说是一种可行的技术手段,理论上讲,通过从头驯化,研究人员可以通过调整少量基因来快速驯化野生植物。在其他物种中尝试的案例,也进一步印证了这一观点。
水稻栽培品种为二倍体,是由其祖先野生二倍体水稻
Oryza rufipogon
驯化而来。但由于全球气候变化,导致的全球变暖、极端高温和低温、干旱和洪水增多、病虫害的出现,培育开发新型水稻品种迫在眉睫。多倍体主要由全基因组复制或种间杂交产生,是开花植物的常见进化模式,多倍体植物在生物量、活力和对环境变化的强大适应能力方面往往具有显著的优势,异源四倍体野生稻(
Oryza alta
)具有生物量大、自带杂种、环境适应能力强等优势,但也具有非驯化特征,无法进行农业生产,所以研究团队将目光瞄准了水稻异源四倍体。
2021年2月,中国科学院遗传与发育生物学研究所李家洋课题组联合梁承志、高彩霞课题组在
Cell
杂志上发表了一篇题为“A route to
de novo
domestication of wild allotetraploid rice”的研究论文。该研究首次提出了异源四倍体野生稻快速从头驯化的新策略,旨在培育出新型多倍体水稻作物,从而大幅提升粮食产量并增加环境变化适应性。
研究团队筛选出一份高秆野生稻资源-多倍体水稻1号(PolyPloid Rice 1,PPR1),PPR1的生物量极大,株高可达2.7米,穗长可达48厘米,叶宽可达5厘米,但它也具备典型的未经过驯化特征,如稀穗、粒小(栽培稻的1/3)、芒长(大于4厘米)等。
研究注释了驯化基因及农艺性状基因,系统分析其同源性,并进一步对高秆野生稻PPR1中控制落粒性、芒长、株高、粒长、茎秆粗度及抽穗期的同源基因进行了基因组编辑,创制出落粒性降低、芒长变短、株高降低、粒长变长、茎秆变粗、抽穗时间不同程度缩短的各种基因编辑材料。
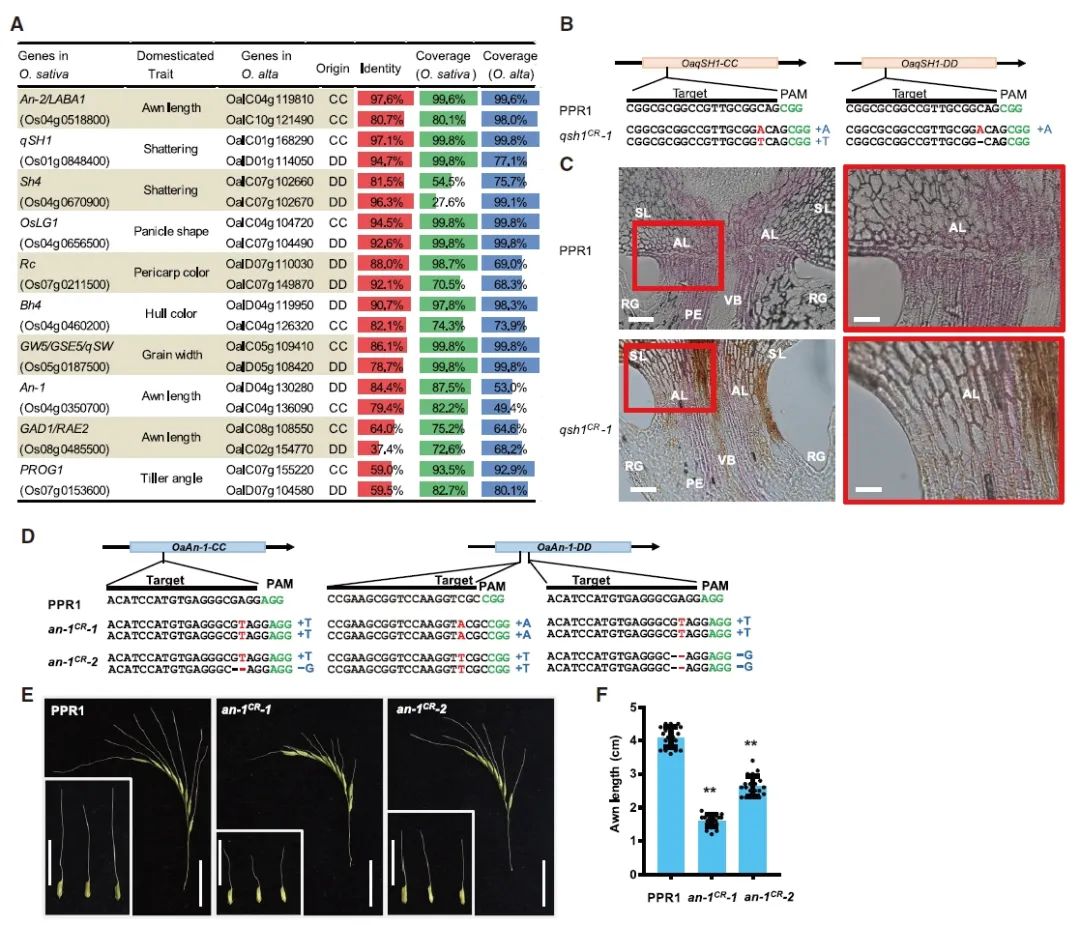
图9 通过基因组编辑快速驯化PPR1(Yu et al., 2021)。(A)10个
O. sativa
驯化相关基因的
O. alta
同源物;(B)基因编辑植株中落粒性相关基因
q
sh
1
的突变形式;(C)野生型和基因编辑植株的木质素含量比较;(D)基因编辑植株中芒长相关基因
An-1
的突变形式;(E-F)野生型和基因编辑植株芒长度比较。

图10 农艺学上重要的水稻
O. sativa
基因与野生稻
O. alta
同源基因的序列相似性和基因组编辑(Yu et al., 2021)。(A)水稻
O. sativa
中113个农艺学上重要的基因与
O. alta
中的同源基因的相似性;(B)基因编辑植株中株高相关基因
SD1
的突变形式;(C-E)野生型和基因编辑植株株高比较;(F)基因编辑植株中粒长相关基因
GS3
的突变形式;(G、H)野生型和基因编辑植株粒长比较。
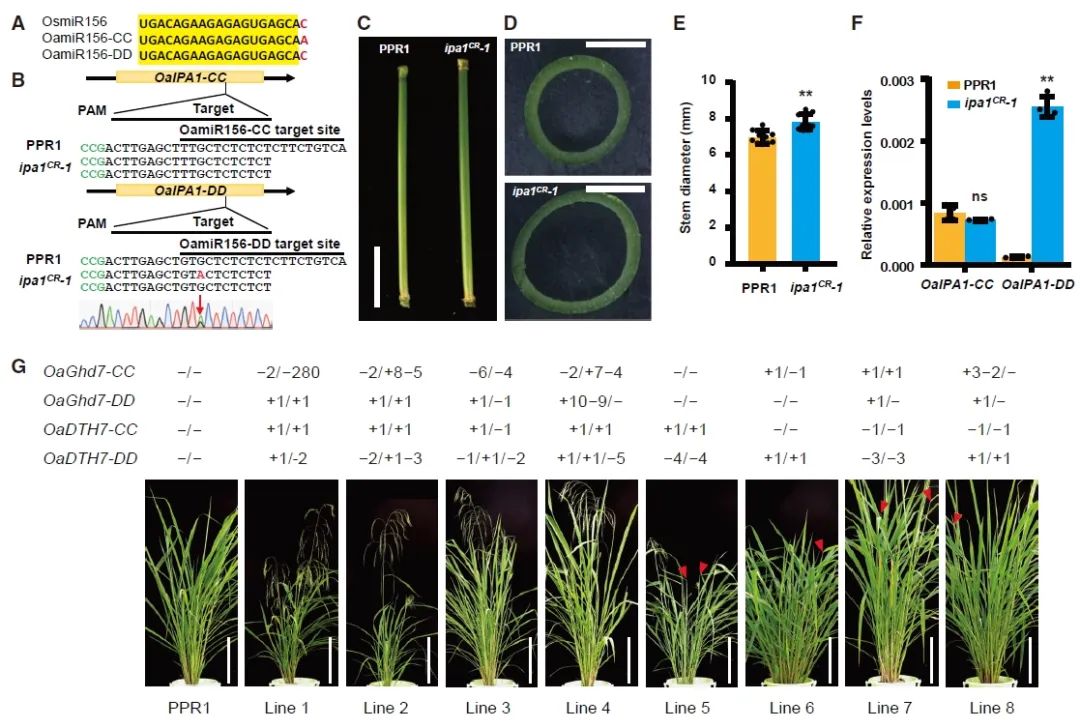
图11 通过碱基编辑器和多重编辑系统改进 PPR1(Yu et al., 2021)。(A)
OsmiRNA156
、
OamiRNA156-CC
和
OamiRNA156-DD
的序列比对;(B)突变体植株中碱基编辑器诱导的点突变形式;(C-F)野生型和突变体植株在茎基部第三节间长度(C)、茎横截面(D)、茎粗度(E)及基部茎基因表达水平(F)比较;(G)野生型和突变体植株抽穗期比较,红色三角形代表新生的圆锥花序。
四倍体马铃薯是生产及研究中经常被使用的对象,但其存在遗传背景复杂、基因组高度杂合、育种周期长及繁殖系数低、储存成本高、易携带病虫害等缺陷,导致四倍体马铃薯的育种工作进展缓慢。为了解决这一困境,科研工作者开始尝试对自然种质资源中广泛存在的二倍体马铃薯进行重新驯化来创制新的品种。
2018年8月,
中国农业科学院深圳农业基因组研究所黄三文课题组联合张春芝课题组在
Nature Plants
杂志在线发表了一篇题为“Generation of self-compatible diploid potato by knockout of
S-RNase
”的研究论文。该研究通过使用CRISPR-Cas9系统敲除马铃薯中自交不亲和基因
S-RNase
创造了自交亲和的二倍体马铃薯。
研究团队从头组装了二倍体马铃薯RNA-seq数据,获得了2个完整的
S-RNase
等位基因。通过设计sgRNA靶向S-RNase蛋白的相对保守结构域并同时靶向
S-RNase
的两个等位基因,最终获得了5个二倍体马铃薯基因编辑植株。基因编辑植株在温室生长、自花授粉后,均产生果实,并且植物形态和长势与野生型没有差异(图12)。T0代自交、T1代分离外源基因后,突变仍然保持稳定,仍是自交亲和,说明诱导
S-RNase
基因功能丧失可以导致二倍体马铃薯变成自交亲和性植株,继而构建“二倍体杂交育种-种子繁育”体系,可实现马铃薯的快速驯化。
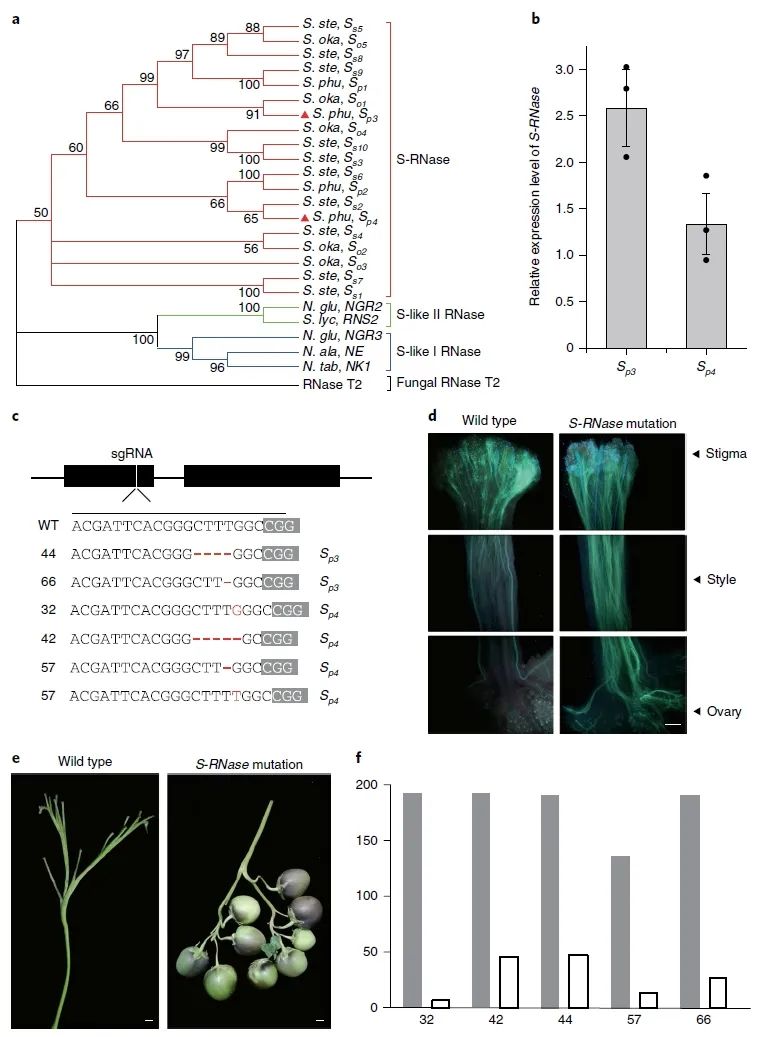
图12
S-RNase
的敲除克服了马铃薯的自交不亲和(Ye et al., 2018)。(a)S-RNases及其类S-RNases的系统发育树;(b)两个
S-RNase
等位基因在花柱中的相对表达量;(c)基因编辑植株中
S-RNase
的突变形式;(d)授粉花柱中花粉管的生长;(e)野生型与基因编辑植株的坐果数量比较;(f)T1代无Cas9幼苗的分离。
多年生作物具有一次种植,多次收获的特性,能
够显著节约人工和机械成本。
此外多年生作物发达的根系能保证高的水肥利用,减少土壤流失。
培育多年生作物是我国推动农业可持续发展和应对未来气候变化的重要战略储备。
2024年5月,中国科学院分子植物科学卓越创新中心王佳伟课题组在
Cell
杂志上在线发表了一篇题为“Reciprocal conversion between annual and polycarpic perennial flowering behavior in the Brassicaceae”的研究论文。该研究揭示了多年生到一年生过渡的进化机制,为研究植物生命历史策略的遗传基础提供了重要模型。
研究团队根据前期一系列实验将喜马拉雅须弥芥(
Crucihimalaya himalaica
)与内华达糖芥(
Erysimum nevadense
)确定为多次结实多年生模式植物,将小花糖芥(
Erysimum cheiranthoides
)和卵叶须弥芥(
Crucihimalaya wallichii
)确定为一年生模式植物。
Crucihimalaya wallichii
与
Crucihimalaya himalaica
杂交
、Erysimum nevadense
与
Erysimum cheiranthoides
杂交得到的F1代也表现出相似的多年生特性,表明多年生特性在这些模型物种中是显性的,支持了多年生性状是由基因突变从一年生祖先演化而来。

图13 两种多年生模式的特征(Zhai et al., 2024)。(A-C)亲本
C. wallichi
的生长习性;(D-F)亲本
C. himalaica
的生长习性;(G-I)两亲本
C. wallichi、C. himalaica
杂交F1代生长习性;(J-K)亲本
E. cheiranthoides
的生长习性;(L-N)亲本
E. nevadense
的生长习性;(O-Q)两亲本
E. cheiranthoides、E. nevadense
杂交F1代生长习性。AV代表“春化后”,W代表“周龄”。
在这两个跨物种的遗传群体中,通过正向遗传学定位到了三个决定植物多年生的MADS-box基因。对照模式植物拟南芥(
Arabidopsis thaliana
)的基因功能注释,发现在这三个遗传区间内存在一类亲缘关系密切的MADS-box转录因子编码基因,即
FLOWERING LOCUS C
(
FLC
)、
FLOWERING LOCUS M
(
FLM
)和
MADS AFFECTING FLOWERING
(
MAF
)。
通过基因编辑突变体、基因的表达模式及表观遗传学分析验证了基因的功能及调控机制。研究发现,植物的生活史策略演化是由这三个基因剂量叠加所决定的连续过程,其中
FLC
基因在多年生特性中起主导作用。尤为重要的是,只需在一年生植物中转入三个基因中的一个,就足以使其生活史策略转向多次结实多年生。
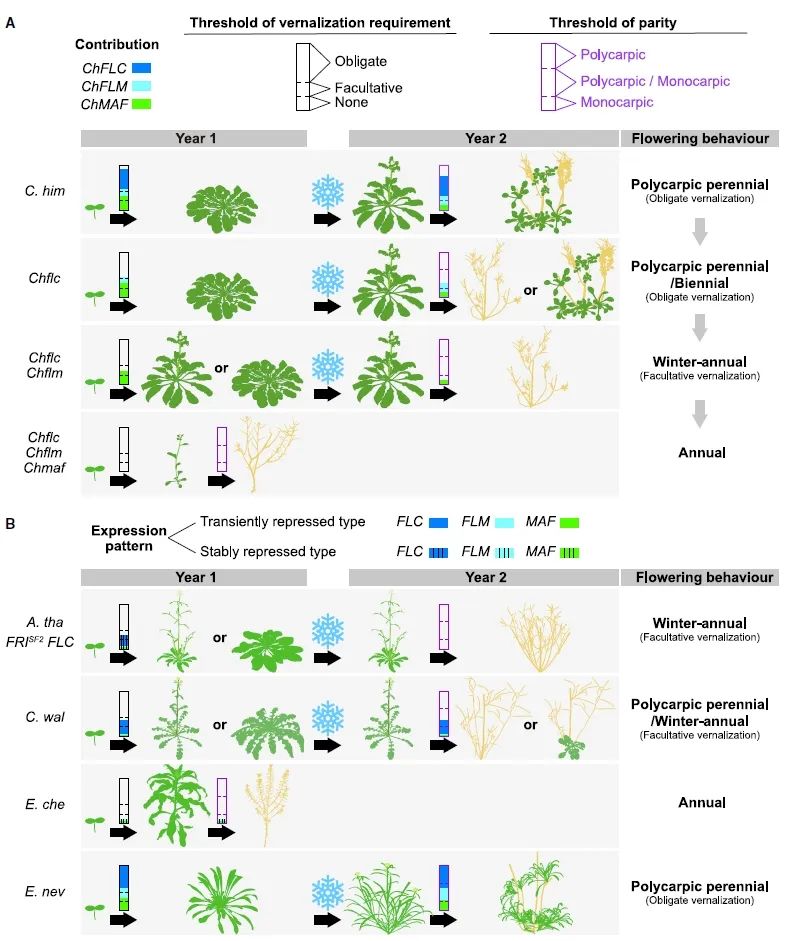
图14
FLC
类基因的剂量叠加效应决定十字花科植物生活史策略的多样化(Zhai et al., 2024)。(A)
C. himalaica
生活史相关开花行为的调控。尽管
ChFLC
、
ChFLM
和
ChMAF
都受到春化的短暂抑制,它们对春化要求和胎次(即多果、多果/单果或单果)校验做出了不同的贡献(用不同的颜色表示;没有颜色表示零贡献)。组合这三个基因的影响根据春化要求的假设阈值(黑色虚线)以及胎次的阈值(紫红色虚线)来定义开花行为(即专性、兼性,或无);(B)拟南芥、
Crucihimalaya
以及
Erysimum
生活史相关开花行为的调控。除了对春化需求和胎次(A)做出不同的贡献外,这些基因在表达模式上也表现出差异(即它们是稳定地还是短暂地受到春化的抑制)。
鉴于MADS-box基因功能在十字花科作物中的保守性,在将来很可能会成为改变十字花科作物生活史策略和开展物种驯化的首选基因。
作物快速驯化能够在短短几年的时间内完成需要花几千年进行的育种工作,但是快速驯化的技术挑战也是巨大的,大多数野生植物没有得到充分的研究,而且一些野生种质资源存在于土著人群的生活环境中,对野生资源的挖掘可能会破坏这一群体内部的和谐。未来这一方向的发展充满了挑战与可能性,尽管现在看来仍存在一些问题,但是没准儿随着时间的推移此时的问题将不再是问题,我们拭目以待!
References
:
Li T, Yang
X, Yu Y
,
et al. Domestication of wild tomato is accelerated by genome editing.
Nat Biotechno
l
.
2018;
36
:
1160–1163.
Zsögön A, Čermák T, Naves E, et al. De novo domestication of wild tomato using genome editing.
Nat Biotechnol
. 2018;36:1211–1216.
Lemmon ZH, Reem NT, Dalrymple J, et al. Rapid improvement of domestication traits in an orphan crop by genome editing.
Nat Plants
. 2018;4:766–770.
Yu H, Lin T, Meng X, et al. A route to de novo domestication of wild allotetraploid rice.
Cell
. 2021;184(5):1156-1170.e14.
Ye M, Peng Z, Tang D, et al. Generation of self-compatible diploid potato by knockout of S-RNase.
Nat Plants
. 2018;4(9):651-654.
Zhai D, Zhang LY, Li LZ, et al. Reciprocal conversion between annual and polycarpic perennial flowering behavior in the Brassicaceae.
Cell
. Published online May 21, 2024.
Michael Marshall. Creating new crops with CRISPR.
Nature
. 2024; 625:230-232.



















