
今天给大家介绍一个miRNA相关的数据库mirDIP(http://ophid.utoronto.ca/mirDIP/)。

作为小张的铁粉,在公众号里应该已经看过N多的miRNA数据库(本文末有miRNA数据库整理),那么今天介绍的这个数据库有何特别之处呢?下面就给大家划重点~
1、支持多miRNAs查询靶基因
据本宫所知,其它miRNA数据库预测miRNA靶基因的时候,只能输入一个miRNA的名称进行预测,比如StarBase,你输入两个基因名称是跑不出来的。
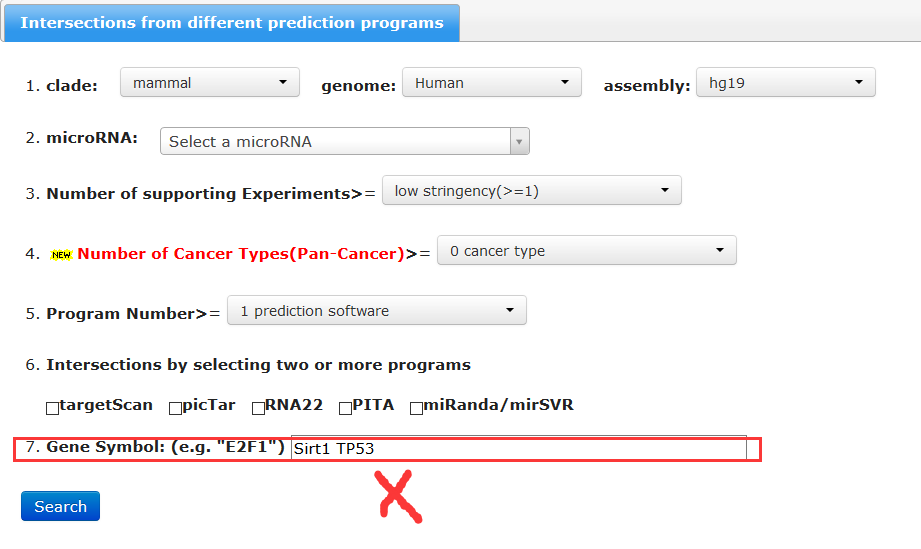
如果你做miRNA芯片检测出一大票的差异miRNAs,而你发现这些个数据库极其不人道的只能一个个的检索靶基因时,想必你的内心一定是奔溃的~~~
所以本宫今天极力推荐mirDIP,它既支持多miRNAs预测靶基因,也支持多mRNAs预测miRNA。
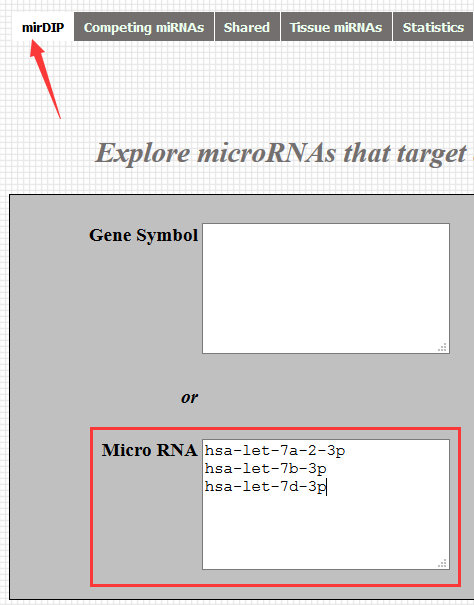
本宫其实不太理解其它数据库为何没有这项功能,个人觉得实现起来应该并不复杂。难道是因为怕大家输入不对miRNA的名称?所以限定只能输入一个,这样系统可以自动匹配标准名称?或者直接像StarBasev那样做成下拉菜单~
mirDIP是需要输入miRNA标准名称的,对miRNA命名还不太熟悉的小伙伴可以看下这篇文章miRNA的名字,到底是什么意思?
2、ceRNA查询
如果你还不太了解ceRNA机制,那你可以看一下X湿兄是如何一本正经地玩坏ceRNA机制的,又或者看看大牛的经典综述ceRNA综述、数据库及经典文章。
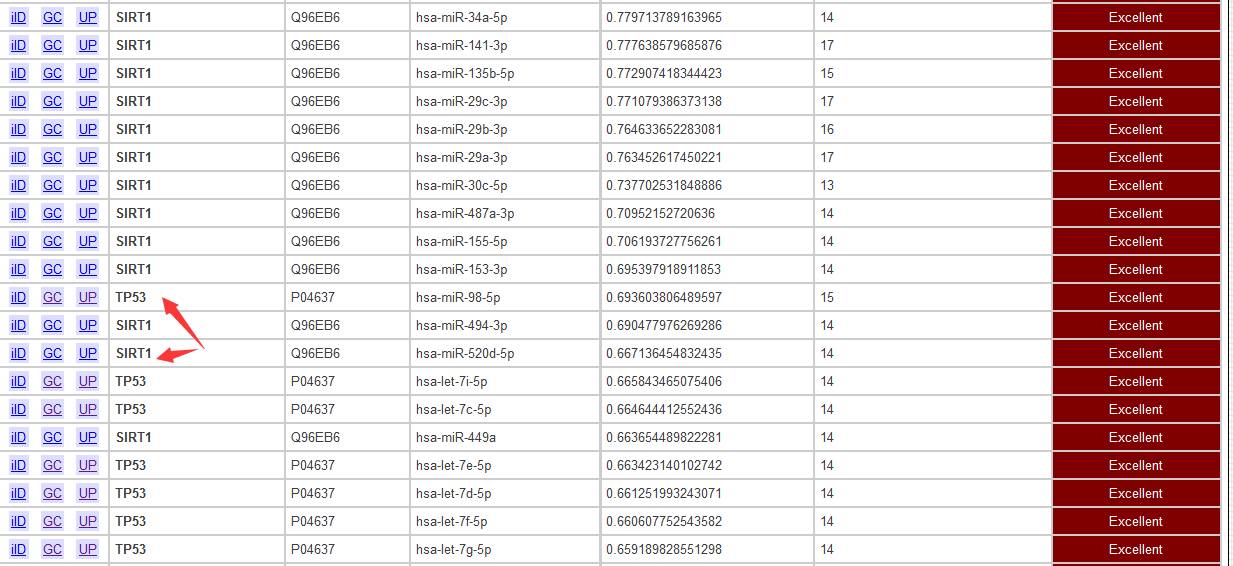
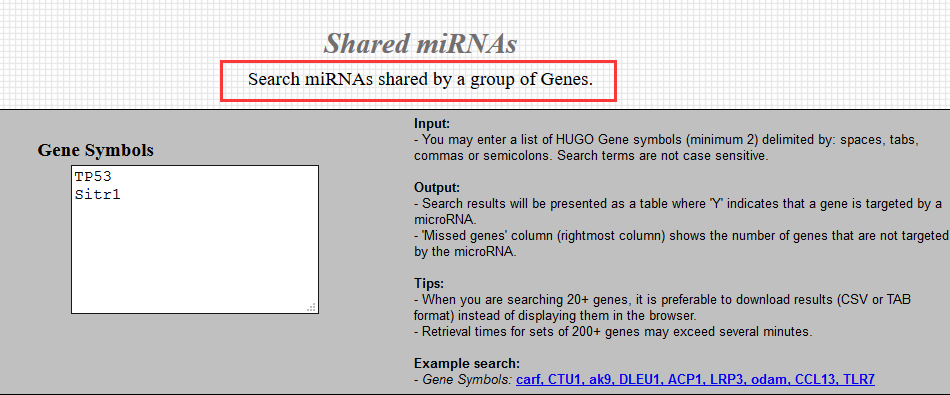
我们可以直接用mirDIP查询两个基因或多个基因之间是否存在相同的miRNA。如果查到两个基因之间确实有相同的miRNA,那就又能玩ceRNA啦~
3、剔除Globally Targeted Genes
有些基因呐,它特别招miRNA喜欢,什么miRNA都和它有一腿(这类mRNA一般是存在某些特殊序列,miRNA预测工具都是用一定的算法预测的,算法不能排除这样的特殊序列导致的假阳性),这样的基因就是Globally Targeted Genes。mirDIP可以剔除这样的基因,降低预测结果的假阳性率。
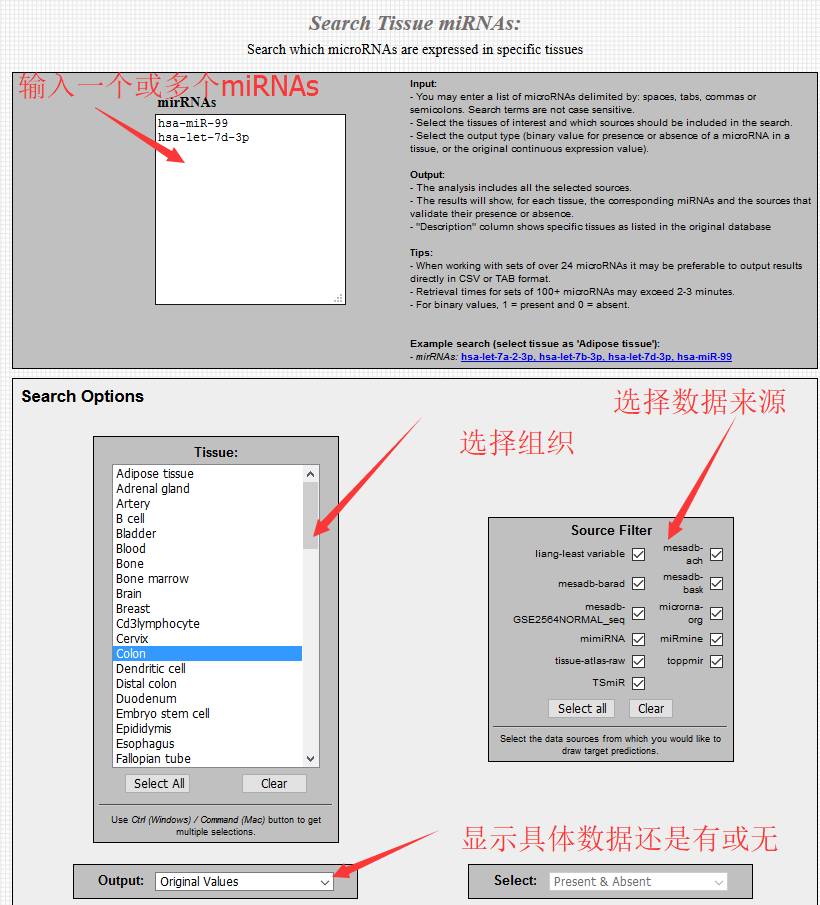
4、miRNA在不同组织中的表达情况
mirDIP可以查询多个miRNAs在不同组织中的表达情况,方便大家进行比对和展示。
文章中是如何运用mirDIP的呢?
Computational identification of microRNAs associated to both
epithelial to mesenchymal transition and NGAL/MMP-9 pathways in bladder
cancer (Oncotarget)
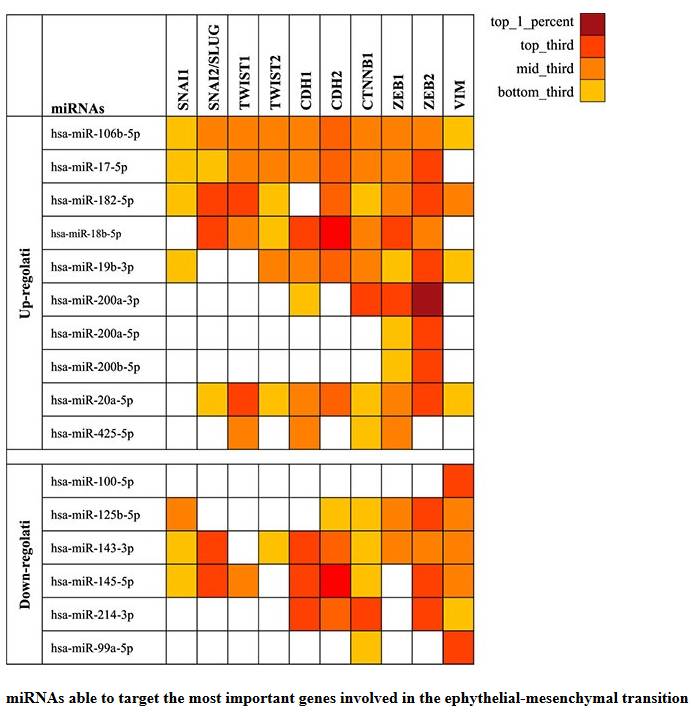
图片展示的是miRNA能够靶向某一生物学过程涉及的多数基因。
今天有mirDIP就介绍到这里了。
特别提醒:mirDIP物种仅限于人类!
更多的miRNA数据库介绍(StarBase,TargetScan,miRTarBase,DIANA等),可以参看下列文章:
我好像发现了一个miRNA各种软件的大本营……
如果miRNA神器只能推荐一个,就是这个了!
一个全新的MicroRNA-Pathway数据库
如何从信号通路、疾病查询microRNA和靶基因?
长按二维码识别关注“小张聊科研”

关注后获取《科研修炼手册》1、2、3、4、5,基金篇、生信特辑















