微生物扩增子测序指通过检测目的区域(高变区)序列的变异度和丰度,以研究环境微生物多样性及群落组成差异。低廉的价格是其备受青睐的关键,其主要从物种组成及分布上进行差异研究。质控拼接是微生物测序的关键前提,其直接决定后续分析结果的优劣,主要包括过滤低质量reads和去除接头、barcode、及嵌合体几个步骤。
Illumine 下机PE reads:sample1_raw_1.fq.gz和sample1_raw_2.fq.gz
常用嵌合体数据库:silva,rdp,glod,这里使用比较小的并方便使用的glod.fa
安装软件Fqcheck,flash,Qiime, Usearch
软件fqcheck,安装过程这里就不详细介绍了,感兴趣的可以自行搜索相关资源。
fqcheck -q 33 -r sample1_raw_1.fq.gz -c sample1_raw_1.fqchek
fqcheck –q 33 -r sample1_raw_2.fq.gz -c sample1_raw_2.fqchek
参数说明:
-q 过滤的最低质量值;
-r 输入文件,原始reads的fastq文件;
-c 输出的质量统计文件
以fqcheck为后缀名的质量统计文件(sample1_raw_1.fqchek,sample1_raw_2.fqchek,见图1)会统计每条reads前100个位点每个位置的ACGTN含量,以及0-40质量值的个数,最终会得到整体的错误率,GC,Q20,Q30(位于文件结尾,见下图)
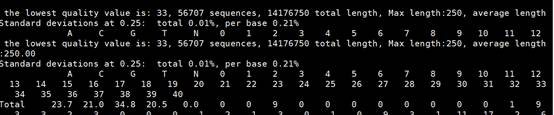
图1 fqchek质量统计文件
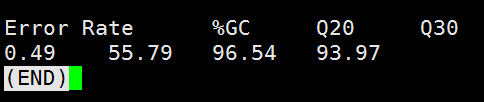
图2 fqcheck质量统计文件最后两行
由于扩增子测序目前多以双末端测序为主,测序得到的PE reads需要经过拼接才能得到目标扩增片段的序列,拼接过程和参数如下:
flash sample1.raw_1.fq.gz sample1.raw_2.fq.gz -o sample1 > sample1.flash.log
#将左右两条reads进行拼接获得扩增区域序列,其结果文件以sample1为前缀名(-o参数设置)
参数说明:
-m 拼接时overlap区的最小长度,默认10bp;
-x overlap区的最大碱基错配数目/overlap区长度,默认0.25;
-p 质量值类型,64或者33,默认33;
-t 线程数,默认1。
共生成6个结果文件,如图3所示
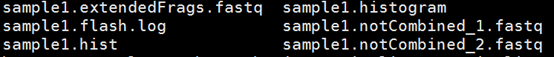
图3 flash拼接的结果文件
其中sample1extendeFrags.fastq文件为扩增片段序列文件,sample1.notCombined_1/2.fastq文件为拼接不上的reads;sample1.flash.log文件详细记录了拼接过程的参数和拼接统计的数据。
拼接得到的tags需要进一步进行质控和序列截取,此部分分析过程和参数如下:
Python split_libraries_fastq.py -q 19 --barcode_type not-barcoded --store_demultiplexed_fastq -o ./ --sample_ids sample1 -i sample1.extendedFrags.fastq –m sample1.mf
#split_libraries_fastq.py为Qiime的python包
参数说明:
-q 测序质量阈值,默认为3,程序建议设为19
-r 需要截断的连续低质量碱基数目,默认为3
-p 一条reads中连续高质量碱基率阈值,reads中连续高质量碱基数目/该条reads长度,默认0.75
--barcode_type 设置barcode类型,默认为golay_12,并支持错配;通常设置为整数,对应barcode的长度总和,(此处设置为not-barcoded,我们的样本已经在下机时去掉barcode);
-m 实验设计,依赖样品barcode列表将序列按样品重新命名,见图4;
-o 输出文件夹
-I 输入文件,此处为上一步拼接的文件
--
store_demultiplexed_fastq 是否产生对应的fastq文件,默认为false
--sample_ids 以逗号隔开的样本名列表,当store_demultiplexed_fastq为真时使用
Sample.mf文件为实验配置文件,需要提前自行配置,主要由sampleID,BarcodeSequence,LinkerPrimeSequence,Description四列组成,其中sampleID和Description为必须配置列(见图4);

图4 sample.mf文件















