首先,研究者分析了拟南芥数据库中可能被PRC2调控的基因序列,并找到170个候选PREs(按照600bp长度)(Fig.S1)。这其中选择3个基因上的5个候选PREs进行验证,分别是:
AGAMOUS (AG):AG_1,AG_2
SEPALLATA3 (SEP3):SEP3_1,SEP3_2
At5g61120 (At5g):At5g
如图Figure1 A-B,PRC2组成蛋白FIE、和H3K27me3都能够结合包含有PREs的片段。进一步利用PRE连接GFP和GUS报告系统,也发现以上PREs都能够抑制报告基因的表达(Fig.1C-D)。
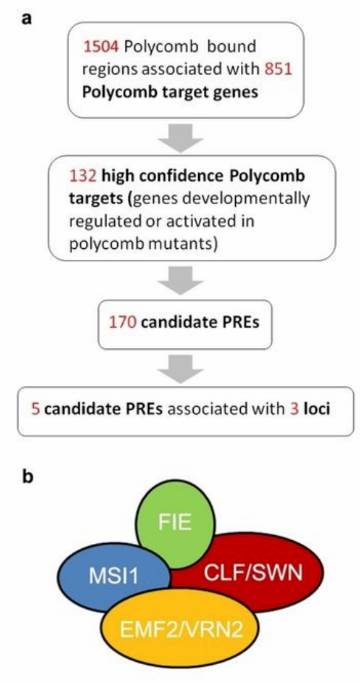
Figure S1 PRC2靶点筛选:靶基因和PREs
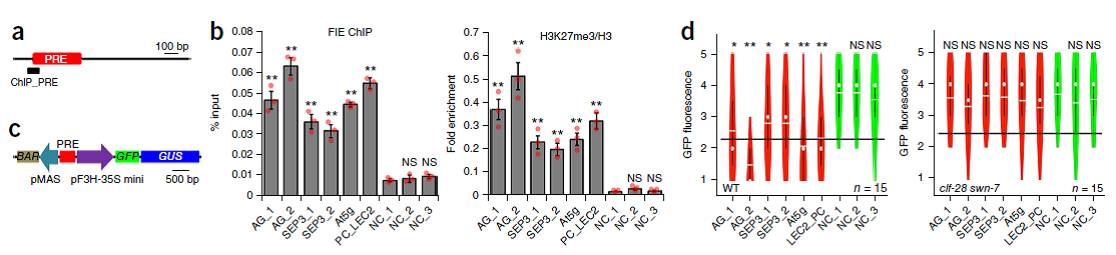
Figure. 1 验证PREs片段功能:介导PRC2调控
接下来,研究者利用上述5个PREs,连接酵母单杂交系统后,用于筛选拟南芥共计1956个转录因子的库,并找到了233个转录因子能够结合。通过进一步加权分析等,进一步缩减到20个亚家族的55个转录因子。在这些亚家族中,排名最前的三个分别为(Fig.2A):
C2H2 zinc-finger (ZnF) family(C1-2iD subfamily; 4 of 6 members identified)
植物特异的APETALA2-like family(AP2 subfamily; 2 of 6 members identified)
BASIC PENTACYSTEINE (BPC) family(class I subfamily; 2 of 3 members identified)
利用酵母双杂交分别验证了筛选获得的转录因子和PRC2各个组成蛋白之间的相互作用(Fig.2B-C);并利用BiFC和Co-IP等手段,进一步验证了部分蛋白之间相互作用(Fig.2D-F)。
验证成功的转录因子举例:TOE1,AZF1,和BPC1等。

Figure. 2 转录因子筛选:能与PREs、和PRC2结合
通过针对上述170个PREs的DNA序列进一步分析,研究者找到了6个motif,分别能够与不同种类的转录因子相结合,包括:CTCC,CCG,G-box,GA repeat,AC-rich和Telobox(Fig.3A-B)。这其中有的motif(如GA repeat和Telobox)已经有报道与拟南芥的Polycomb的分布相关联,再次证实了本文方法的可重复性。继续利用Figure 1A中的报告系统,发现将AG_2和At5g中的Telobox和GA repeat motif分别突变,都会影响PRC2的抑制作用(Fig.3C)。更有趣的是,这些GA repeat在整个十字花科植物中都是保守的(Fig.3D)。
接下来研究者利用凝胶阻滞(EMSA)实验,在体外证明了这些转录因子和相应DNA motif的结合(Fig.3E):
1)GA repeat和BPC家族的BPC1
2)Telobox和C1-2iD ZnF家族的AZF1
以上研究将PRC2组成蛋白—转录因子—结合的序列即PREs片段直接联系起来。
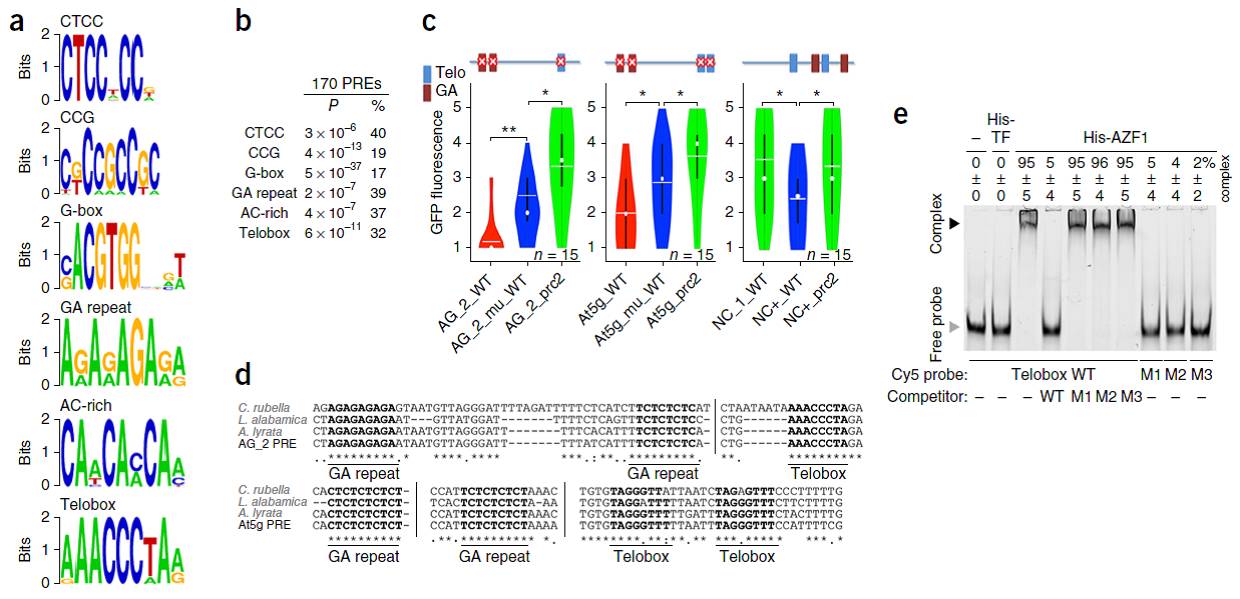
Figure. 3 PREs的功能发挥需要其上保守的顺势作用元件














