PROPKA3
是一个基于经验的
pKa
预测工具,在3.1版本中还加入了
蛋白配体复合物
的
pKa
预测。其是计算蛋白电荷的知名服务
PDB2PQR
中使用的
pKa
预测工具,其包全为Python所写。
其
Github
地址如下: https://github.com/jensengroup/propka-3.1
首先从
G
ithub
中下载安装包:
然后进行常规的安装,要求
python2.7
或者
python3.1
及以上
cd propka-3.1python setup.py install --user
使用的方法也非常简单,例如:
propka31 1hpx.pdb
其计算方法主要是增加了库仑力与氢键的作用,例如:
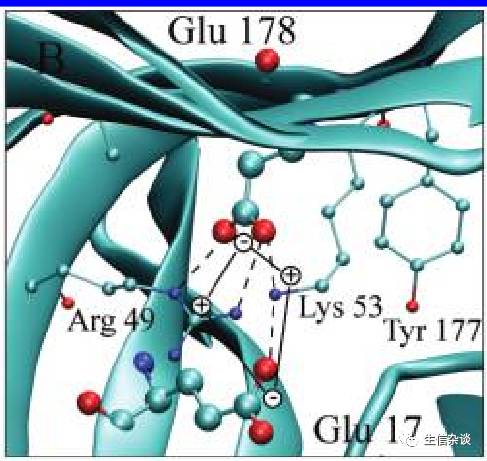
Glu 17 and Glu 178 in Bacillus agaradhaerens xylanase
因为Arg 49与Glu 17库伦联系使得其大约降低1.7pH的pKa值,与Lys53的联系使得降低了1.9pH(0.9的氢键联系和1.0的库伦作用)
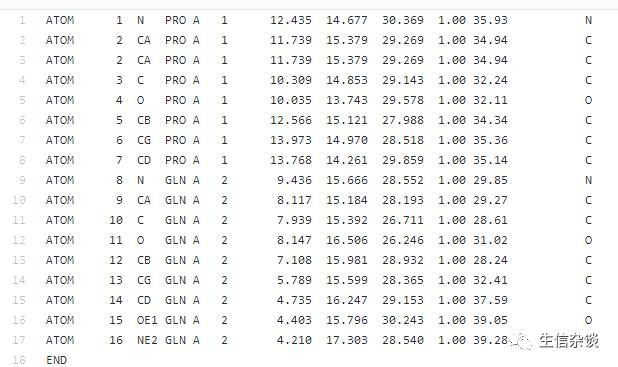
值得注意的是其好像只计算极性氨基酸残基的pKa值,例如
1HPX-warn.pdb
而其
result
为:
7.95
只有一个数据
参考文献:
-
Sondergaard, Chresten R., Mats HM Olsson, Michal Rostkowski, and Jan H. Jensen. "Improved Treatment of Ligands and Coupling Effects in Empirical Calculation and Rationalization of pKa Values." Journal of Chemical Theory and Computation 7, no. 7 (2011): 2284-2295.
-
Olsson, Mats HM, Chresten R. Sondergaard, Michal Rostkowski, and Jan H. Jensen. "PROPKA3: consistent treatment of internal and surface residues in empirical pKa predictions." Journal of Chemical Theory and Computation 7, no. 2 (2011): 525-537.
更多原创精彩视频敬请关注
生信杂谈:






