组蛋白乙酰化是表观遗传调控的主要方式之一,在植物生长发育和响应逆境胁迫过程中发挥重要作用(Kumar et al., 2021)。组蛋白乙酰化主要由组蛋白乙酰转移酶(HATs)和组蛋白去乙酰化酶(HDACs)共同催化调控,是一种动态可逆的修饰状态(Grunstein, 1997)。土壤盐胁迫作为植物最主要的非生物胁迫之一,严重影响植物的生长发育,更会导致农作物严重减产。解析HATs和HDACs介导的组蛋白乙酰化动态变化在植物响应盐胁迫中的作用,不仅可以阐明植物对盐胁迫响应表观遗传的调控机制,同时也为培育农作物耐盐品种提供理论依据。
JIPB
近日在线发表了扬州大学龚志云课题组题为“Histone deacetylase OsHDA706 increases salt
tolerance via H4K5/K8 deacetylation of
OsPP2C49
in rice”
(
https://doi.org/10.1111/jipb.13470
)
的研究论文。
该研究发现水稻中一个
RPD3/HDA1
家族的组蛋白去乙酰化酶
OsHDA706
能够特异去除组蛋白
H4
乙酰基,并通过调控
H4K5ac
和
H4K8ac
修饰降低
2C
类蛋白磷酸酶基因
OsPP2C49
的表达,从而提高水稻的耐盐性。
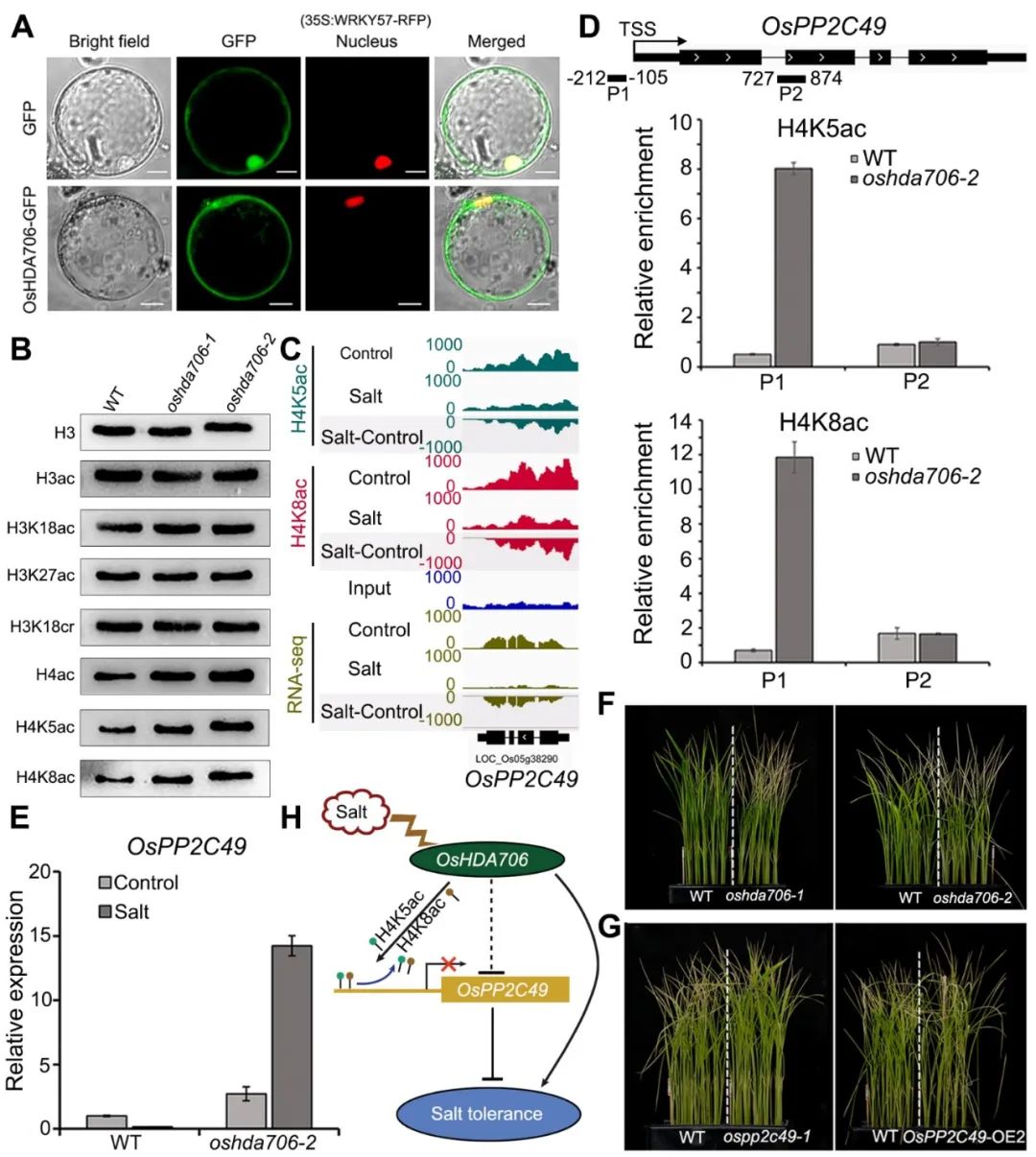
图. OsHDA706通过H4K5ac和H4K8ac影响
OsPP2C49
的表达正调控水稻耐盐性
龚志云
课题组研究发现OsHDA706在叶片中高表达,并且受盐胁迫诱导,其蛋白定位于细胞核和细胞质中,能够特异性去除组蛋白H4上的赖氨酸乙酰化(H4K5ac和H4K8ac)。通过基因编辑技术获得了
OsHDA706
的两个敲除突变体(
oshda706-1
和
oshda706-2
),发现该基因功能缺失后,导致植株对盐胁迫高度敏感。RNA-seq和荧光定量分析发现,盐胁迫处理后2C类蛋白磷酸酶基因
OsPP2C49
表达显著降低,但在
oshda706-2
中的表达水平显著高于野生型。ChIP-seq分析表明盐胁迫下
OsPP2C49
基因上H4K5ac和H4K8ac修饰富集水平明显降低;进一步ChIP-qPCR分析发现在
oshda706
-2突变体中
OsPP2C49
启动子区域的H4K5ac和H4K8ac出现显著富集,暗示
OsPP2C49
可能是OsHDA706响应盐胁迫的下游靶基因。构建了
OsPP2C49
的敲除和过表达株系,证明
OsPP2C49
负调控植株耐盐性。
以上研究结果为解析组蛋白乙酰化调控植物响应盐胁迫的表观遗传机制提供了理论基础
。
龚志云课题组
近年来在水稻组蛋白酰化修饰研究中取得了一系列进展,揭示了水稻中多种组蛋白酰化在基因表达调控和逆境响应中的作用
(Liu et al., 2018;
Xue et al., 2018; Liu et al., 2019; Xue et al., 2020)
。这篇论文进一步揭示了组蛋白乙酰化动态调控在水稻响应盐胁迫中的重要意义。博士生
刘凯
和硕士生
陈积金
为该论文的第一作者,
龚志云
教授、
周勇
教授和
薛超
博士为共同通讯作者。该研究得到了国家自然科学基金和江苏省种业振兴揭榜挂帅项目等资助。
参考文献:
Grunstein, M.J.N.
(1997). Histone acetylation
in chromatin structure and transcription. Nature
389:
349-352.
Kumar, V., Thakur, J.K., and
Prasad, M.
(2021). Histone acetylation dynamics regulating plant development and stress
responses. Cell. Mol. Life Sci.
78:
4467-4486.
Liu, S., Liu, G., Cheng, P.,
Xue, C., Zhou, Y., Chen, X., Ye, L., Qiao, Z., Zhang, T., and Gong, Z.
(2019). Genome-wide
Profiling of Histone Lysine Butyrylation Reveals its Role in the Positive
Regulation of Gene Transcription in Rice. Rice
12:
86.
Liu, S., Xue, C., Fang, Y.,
Chen, G., Peng, X., Zhou, Y., Chen, C., Liu, G., Gu, M., Wang, K., Zhang, W.,
Wu, Y., and Gong, Z.
(2018). Global Involvement of Lysine Crotonylation in Protein Modification and
Transcription Regulation in Rice. Mol. Cell. Proteomics
17:
1922-1936.
Xue, C., Liu, S., Chen, C.,
Zhu, J., Yang, X., Zhou, Y., Guo, R., Liu, X., and Gong, Z.
(2018). Global Proteome
Analysis Links Lysine Acetylation to Diverse Functions in Oryza Sativa.
Proteomics
18:
1700036.
Xue, C., Qiao, Z., Chen, X.,
Cao, P., Liu, K., Liu, S., Ye, L., and Gong, Z.
(2020). Proteome-Wide Analyses Reveal
the Diverse Functions of Lysine 2-Hydroxyisobutyrylation in Oryza sativa. Rice
13:
34.
为了不让您最关心的内容被湮没
防止我们一不小心失散
快把“iPlants”设置为星标吧★
只需三步↓↓
文章顶部点击「iPlants」名称进入公众号主页,点击右上角「三个小点」,点击「设为星标」,iPlants名称旁边出现一个黄色的五角星,就设置成功啦~














