
最近几篇推送会简单介绍几个常见的弱相互作用测试集,欢迎大家批评指正。标题中的测试集在文献中可能对应好多种英文说法,如data set、
benchmark database
、
benchmark set
、
training set
、
validation set
、
assessment set
、
test set
等等,在不同的用途中应该选用恰当的说法。
JSCH2005
、
S22
文献:
Petr Jurečka, Jiří Šponer, Jiří Černý, Pavel Hobza,
Phys. Chem. Chem. Phys.
, 2006,
8
, 1985–1993
1. JSCH2005
JSCH
为作者四人的姓的缩写,
2005
为提出年份。
该论文发表于
2006
年,但文章的收稿日期为
2006
年
1
月
4
日,因此文章应该是
2005
年完成的。
JSCH
中共有
143
个结构,包含了三种摆放方式的
DNA
、
RNA
碱基对和一些氨基酸对,原子数最少
24
,最多
54
。
几个代表性的结构如下图所示:
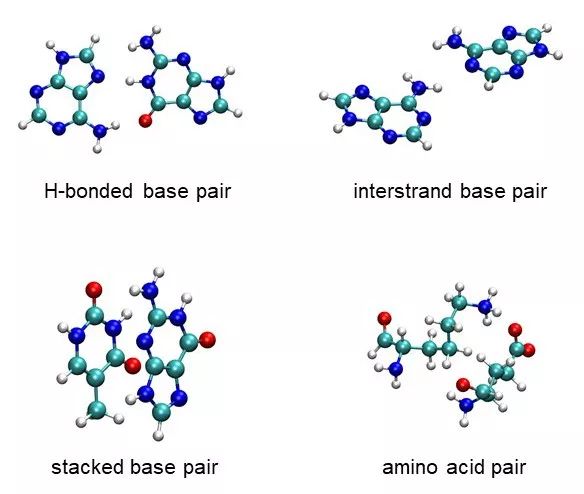
在这类化合中,主要存在氢键和色散两种弱相互作用。文章主要对这些化合进行了
MP2
和
CCSD(T)
水平的结合能的计算。
在这篇文章中,作者对我们常说的结合能作了两种形式的区分。结合能的笼统定义为
Δ
E
=
E
(AB)−
E
(A)−
E
(B)
即复合物的能量减去两个单体的能量,而单体的结构是直接取自复合物的结构还是经过优化的结构,便对应着两种定义,作者在文中分别称之为
interaction energy
和
stabilization energy
,两者之差实际就是单体在复合物中结构和经过优化的结构的能量差,称为
deformation energy
。
这也提醒我们在自己的研究中,要注意当谈及结合能时,最好说清楚单体的结构是否经过优化。
在计算方法上,作者在
MP2
水平下,使用了
aug-cc-pVDZ
、
aug-cc-pVTZ
基组,部分较小的体系还使用了
aug-cc-pVQZ
基组,并使用了以下两种外推方式,将能量外推到基组极限:
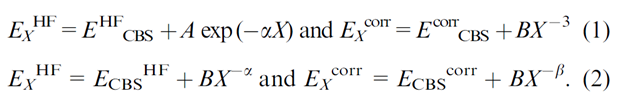
在
CCSD(T)
方法下,由于当时的计算条件远不如现在,最多只能用到
aug-cc-pVDZ
基组,因此
CCSD(T)
在基组极限下的能量使用了以下方法得到:
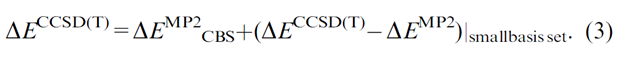
以上外推和近似在《
电子能量的基组外推以及ORCA中的自动实现
》一文中均提到过。
对比以氢键结合和堆叠方式结合的碱基对的结合能,文章中得到结论,
MP2
方法通常会略微低估氢键作用,而比较多地高估色散相互作用。
事实上,这已经是目前计算化学领域的常识了,
MP2
方法对氢键往往表现不错,而对
π-π
类型的弱相互作用往往会严重高估。
因此在处理色散相互作用主导的体系时,
CCSD(T)
是非常有必要的。
此外,作者还得到一个有趣的结论。长期以来人们认为
DNA
双螺旋结构的主要稳定因素来自于两条链之间的氢键相互作用,而文中作者发现碱基对之间的氢键相互作用大概在
10
到
30 kcal/mol
,而色散相互作用往往也会有
10
几
kcal/mol
,而在实际的
DNA
双螺旋结构中是存在碱基之间的堆叠结构的,因此色散相互作用也是很关键的。
2. S22
S22
为
Set 22
的缩写,包含了
22
个化合物,是一些较小的模型体系。
作者将其分成了三类,如下图所示:
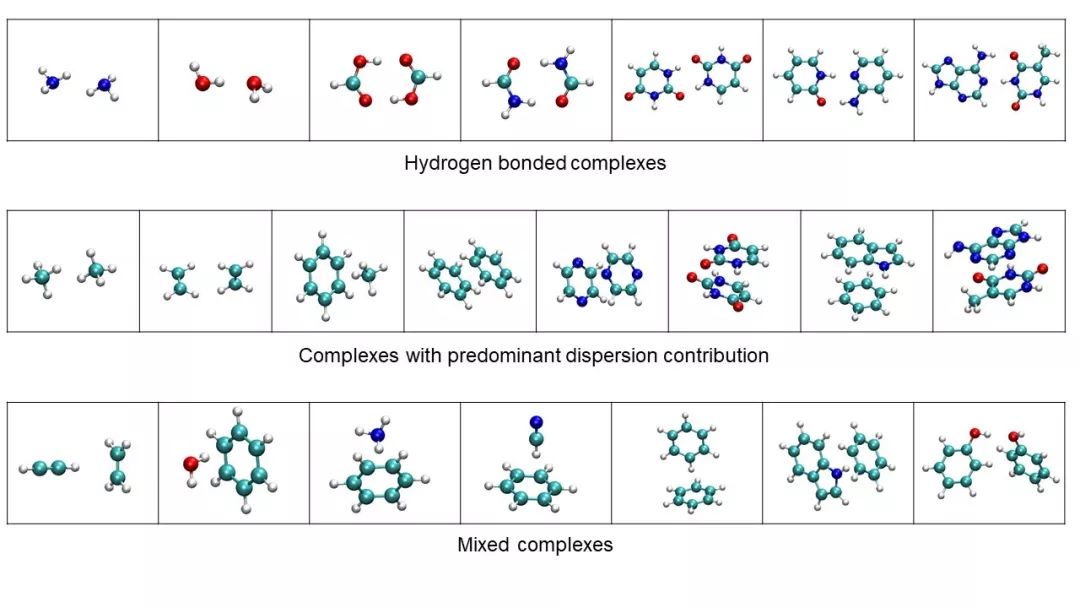
关于相互作用能的计算,所用的方法和所得结论与
JSCH2005
类似。
文中作者提到,在当时,一个新的方法通常只用少量含氢键的体系和极少的(有时只有一个)色散作用体系进行参数拟合和测试。而将方法应用到实际体系中之后才会发现其不足之处。因此他们提出一个比较稳定的数据集,可供新方法进行测试。作者认为他们提出的
S22
数据集中包含了各种弱相互作用类型,可以较好地用来对量子化学方法(如
DFT
方法)进行评价。
受限于当时的计算能力,
S22
数据集中的结合能的精度还不是太高。
而且作者根据体系的大小,对不同的体系使用了不同大小的基组。对公式(













