下面我们仔细了解一下谷歌号称的重构人类基因组的深度学习技术到底是什么。首先,重构人类基因组是个具有巨大计算挑战的特殊领域,而DeepVariant其实和人类基因组重构并没有太多的关系。它所做的和业界采用的思路并没有太大的区别,都是通过与已知的人类基因组的比较来寻找基因变异,和传统方法的区别仅仅在于进行比较的实现方法。
一般来说,传统方法 利用严格的统计数学模型来检测基因突变,而DeepVariant则另辟蹊径,采用视觉深度神经元网络为核心,用监督学习(见下文描述 )出来的模型去发现个体和已知基因组的差异(变异基因)。 坦白地说,DeepVariant所采用的也不是一种完全崭新的策略,因为只要对基因组方面的学习方法应用有所了解的同学就知道,从2000年前后开始流行的机器学习方法,到当下正热的深度学习方法都已经在基因组数据分析领域做了不少尝试,比如GATK里的VQSR功能(Variant Quality Score Recalibration, 变异位点质量值重新校正,另外一种基于机器学习以提高寻找基因变异精度的方法)。但是很遗憾,由于基因数据的特殊性,这些学习方法还没有达到影像数据分析那样,特别是医学影像数据分析那样的成功。因为基于学习的人工智能,简单来讲,是数据驱动的数学建模方法。因此数据本身的某些性质决定了学习方法的有效性,而同时学习方法所构建的模型核心在很多情况下是难以解释的或者不明确的,也就是常说的黑盒子。因而在大多数情况下,人们很难明确地知道到底数据里面哪些特性决定学习方法的有效性。DeepVariant的聪明之处在于从设计思路上,它不再聚焦于高通量测序数据本身,而是把已经在影像分析领域取得巨大成功的深度学习方法应用在高通量数据的展示方面, 通过模仿人类观察高通量测序数据的方式来寻找基因变异,因此它才在这个领域有所建树。同时在仔细研究DeepVariant的训练方法后,我们注意到它所用的训练样本也是来源于DeepVariant号称要完胜的统计数学模型。
在2016年面向全球的美国FDA主导的精准FDA真相比赛中(precisionFDA Truth Challenge),DeepVariant的早期版本(以Verily Life Sciences的名义提交)拿到了六个奖项中的最佳单核苷酸多态性(SNP)综合分数奖,这也是所谓在准确性上有“突破性意义”的主要证据之一。但是如果看表中的结果,它并没有所宣称地那样在精度上有突破性地进展,而只是在SNP F-score(一个用来综合衡量SNP性能的指标)一项上有极其微弱的优势,在SNP其它维度上表现平平,而在INDEL上的所有维度更是距领先者有着比较明显的差距(SNP即单核苷酸多态性,指基因组上的单个核苷酸的变异;INDEL即插入缺失标记,指少量的核苷酸插入或者缺失的变异)[3][4]。

图1. precisionFDA Truth Challenge 的六个奖项[3]
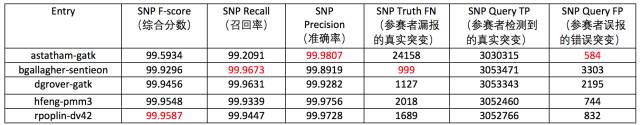
表1. 所有获奖团队的单核苷酸多态性(SNP)表现(DeepVariant以rpoplin-dv42的名字出现)
其中标红的为最佳表现[4]
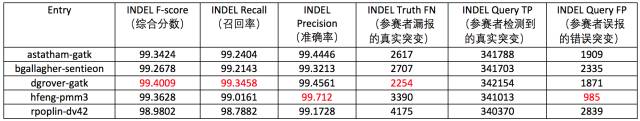
表2. 所有获奖团队的插入缺失标记(INDEL)表现
(DeepVariant以rpoplin-dv42的名字出现)
其中标红的为最佳表现[4]














