BioArt按
:
近年来赖氨酸修饰研究的一大重要进展是发现赖氨酸不仅存在乙酰化修饰,还存在一系列脂酰化修饰如巴豆酰化、丙酰化、丁酰化、琥珀酰化、丙二酰化等,其中组蛋白巴豆酰化的修饰因其广泛发生于组蛋白并与基因表达活化相关越来越受到研究者的重视。过去几年里,尽管有一些研究报道了调控组蛋白巴豆酰化修饰的重要因子(主要是系列催化巴豆酰化产生以及去除的酶类),但是是否还有其它重要的调控因子参与调节组蛋白巴豆酰化修饰的水平目前并不清楚。8月10日,
Molecular Cell
杂志发表了来自北京大学
尚永丰
院士课题组题为“Chromodomain Protein CDYL Acts as a Crotonyl-CoA Hydratase to Regulate Histone Crotonylation and Spermatogenesis”的研究论文,
报了Chromodomain蛋白CDYL作为一种巴豆酰辅酶A水合酶参与调控组蛋白巴豆酰化修饰水平及其在精子发生中的作用。该研究为进一步弄清楚包括组蛋白巴豆酰化在内的一些新修饰提供了新的视角
。尚永丰和梁静为本文的共同通讯作者。

论文解读:
2011年,芝加哥大学赵英明教授实验室在
Cell
杂志上首次报道了组蛋白上的巴豆酰化修饰修饰【1】,此后围绕巴豆酰化修饰的相关研究渐渐展开,目前的相关研究已经比较能够系统的了解巴豆酰化修饰是如何产生、如何被去除、如何被识别【2-9】,此外有关非组蛋白巴豆酰化修饰的相关研究也有一些研究报道【10,11】。
尚永丰实验室长期从事表观遗传学相关研究,近几年来也开始重视并研究一些组蛋白上的新修饰,去年该实验室还报道了SIRT7调控去组蛋白琥珀酰化修饰的相关研究【12】。
在这篇文章中,研究人员主要聚焦在了一个名为CDYL的蛋白身上。
CDYL
(Chromodomain Y-like)是人类Y染色体上含有Chromodomain的一个蛋白,1999年被克隆【13】。过去的研究表明,CDYL能够结合组蛋白H3K9甲基化修饰以及结合组蛋白去乙酰化酶HDAC1/2,被认为是一个转录共抑制因子参与基因转录调控
【14,15】
。早前,尚永丰课题组还曾报道CDYL能够协助PRC2介导的H3K27me3抑制基因的转录【16】,最近该课题组还在
JMCB
杂志上报了CDYL在抑制性组蛋白修饰传递过程中的作用【17】,所以说该课题组在研究CDYL方面有很好的基础。
事实上,多年以前研究人员就发现CDYL蛋白的C端包含一个
烯酰辅酶A水合酶结构域(enoyl-coenzyme A hydratase),具有辅酶A结合口袋(CoA-binding pocket)【15】,还有报道显示CDYL在体外有乙酰转移酶活性【18】。给予过去的这些研究以及近几年的研究,研究人员最先想到的是CDYL是否具有催化组蛋白巴豆酰化形成的能力,通过一些列体外实验发现,CDYL并不能催化巴豆酰化修饰形成。尽管如此,研究人员进一步想知道CDYL蛋白的变化是否与细胞内组蛋白巴豆酰化修饰水平相关。通过一些列的基因敲低以及基因敲除实验表明,CDYL能够负控
组蛋白巴豆酰化修饰的变化(
下图
)。
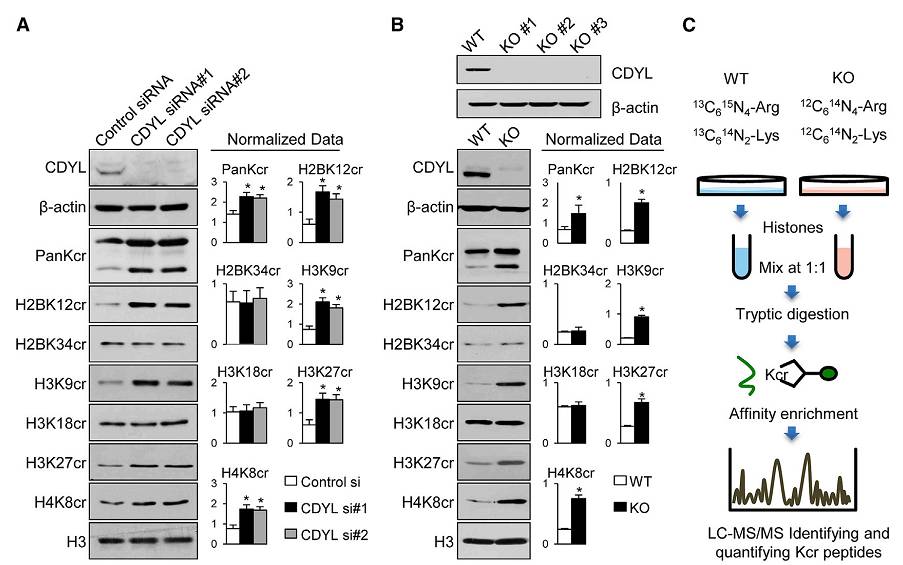
在获得了上面的研究结果之后,很显然需要进行一些常规实验,比如CDYL的哪个结构域是调控
组蛋白巴豆酰化修饰所必须的,关于这一点通过在KO细胞中用不同突变体做回复实验即可证明,结果是
Chromodomain 和CoAP Domain都是必需的。
那到底CDYL是如何调控巴豆酰化修饰的呢?首选最容易想到的是CDYL是否有类似于HDAC家族蛋白的功能能够直接去组蛋白除巴豆酰化基团。答案显然是否定的。这就需要考虑其它可能的作用方式了,几番周折之后研究人员发现CDYL事实上作用的对象是巴豆酰辅酶A,这一点大概可以从2009年的一篇不起眼的论文中找出一些端倪【19】。这篇文章从结构生物学的角度分析CDYL可能作为一个水合酶能够直接作用巴豆酰辅酶A。后面的实验结果显然证实了这一点,即CDYL通过酶活作用将巴豆酰辅酶A转变成β-羟丁酰辅酶A。
故事讲到这就差不多了,再往后就是常规动作了。因为过去很早就知道CDYL能够抑制基因的转录并且在精子发生中具有重要的调控作用。所以,这篇文章从CDYL调控巴豆酰化修饰水平这一点出发从另一个角度阐明了其调控基因转录以及精子发生过程中的作用。
总的来说,这是一项非常有意思的研究工作,为研究细胞体内巴豆酰化修饰的水平是如何调控的提供了新的视角。当然,也有一些问题还需要进一步阐明。比如,CDYL这个水合酶的活性如何更好的更直接的被证实;CDYL调控巴豆酰化的水平如何同过去参与调控HDAC1/2的功能区分开,因为HADC1/2能够去除巴豆酰化;CDYL调控精子发生直接通过巴豆酰化起作用的程度有多少,如何排除影响组蛋白乙酰化或甲基化修饰所造成的影响,再者,似乎没有直接看到敲除CDYL之后组蛋白乙酰化、β-羟丁酰化等修饰水平。当然,一篇文章不能解决所有的问题,希望围绕CDYL的新功能将来会有更深入的研究。
参考文献:
1、
Tan M, Luo H, Lee S, et al. Identification of 67 histone marks and histone lysine crotonylation as a new type of histone modification.
Cell
2011; 146:1016–1028.
2、Sabari BR, Tang Z, Huang H, et al. Intracellular crotonyl-CoA stimulates transcription through p300-catalyzed histone crotonylation.
Mol Cell
2015; 58:203–215.
3、
Wei, W., Liu, X., Chen, J., Gao, S., Lu, L., Zhang, H., ... & Li, J. (2017). Class I histone deacetylases are major histone decrotonylases: evidence for critical and broad function of histone crotonylation in transcription.
Cell Research
.
4、Bao X, Wang Y, Li X, et al. Identification of 'erasers' for lysine crotonylated histone marks using a chemical proteomics approach.
eLife
2014; 3. doi: 10.7554/eLife.02999
5、Liu, X., Wei, W., Liu, Y., Yang, X., Wu, J., Zhang, Y., ... & Lei, M. (2017). MOF as an evolutionarily conserved histone crotonyltransferase and transcriptional activation by histone acetyltransferase-deficient and crotonyltransferase-competent CBP/p300.
Cell Discovery
, 3, 17016.
6、Li Y, Sabari BR, Panchenko T, et al. Molecular coupling of histone crotonylation and active transcription by AF9 YEATS domain.
Mol Cell
2016; 62:181–193.
7、Xiong X, Panchenko T, Yang S, et al. Selective recognition of histone crotonylation by double PHD fingers of MOZ and DPF2.
Nat Chem Biol
2016; 12:1111–1118.
8、Andrews FH, Shinsky SA, Shanle EK, et al. The Taf14 YEATS domain is a reader of histone crotonylation.
Nat Chem Biol
2016; 12:396–398.
9、Zhao D, Guan H, Zhao S, et al. YEATS2 is a selective histone crotonylation reader.
Cell Res
2016; 26:629–632.
10、Wei, W., Mao, A., Tang, B., Zeng, Q., Gao, S., Liu, X., ... & Wong, J. (2017). Large-scale identification of protein crotonylation reveals its role in multiple cellular functions.
Journal of Proteome Research
, 16(4), 1743-1752.
11、Xu, W., Wan, J., Zhan, J., Li, X., He, H., Shi, Z., & Zhang, H. (2017). Global profiling of crotonylation on non-histone proteins.
Cell research,
27(7), 946.
12、Li, L., Shi, L., Yang, S., Yan, R., Zhang, D., Yang, J., ... & Liang, J. (2016). SIRT7 is a histone desuccinylase that functionally links to chromatin compaction and genome stability.
Nature communications
, 7.
13、Lahn, B. T., & Page, D. C. (1999). Retroposition of autosomal mRNA yielded testis-specific gene family on human Y chromosome.
Nature genetics
, 21(4), 429-433.
14、Fischle, W., Franz, H., Jacobs, S. A., Allis, C. D., & Khorasanizadeh, S. (2008). Specificity of the chromodomain Y chromosome family of chromodomains for lysine-methylated ARK (S/T) motifs.
Journal of Biological Chemistry
, 283(28), 19626-19635.
15、Caron, C., Pivot‐Pajot, C., van Grunsven, L. A., Col, E., Lestrat, C., Rousseaux, S., & Khochbin, S. (2003). Cdyl: a new transcriptional co‐repressor.
EMBO reports
, 4(9), 877-882.
16、
Zhang, Y., Yang, X., Gui, B., Xie, G., Zhang, D., Shang, Y., & Liang, J. (2011). Corepressor protein CDYL functions as a molecular bridge between polycomb repressor complex 2 and repressive chromatin mark trimethylated histone lysine 27.
Journal of Biological Chemistry
, 286(49), 42414-42425.
17、Liu, Y., Liu, S., Yuan, S., Yu, H., Zhang, Y., Yang, X., ... & Sun, L. (2017). Chromodomain protein CDYL is required for transmission/restoration of repressive histone marks.
Journal of molecular cell biology,
1-17.
18、Lahn, B. T., Tang, Z. L., Zhou, J., Barndt, R. J., Parvinen, M., Allis, C. D., & Page, D. C. (2002). Previously uncharacterized histone acetyltransferases implicated in mammalian spermatogenesis.
Proceedings of the National Academy of Sciences
, 99(13), 8707-8712.
19、
Wu, H., Min, J., Antoshenko, T., & Plotnikov, A. N. (2009). Crystal structures of human CDY proteins reveal a crotonase‐like fold.
Proteins: Structure, Function, and Bioinformatics,
76(4), 1054-1061.















