RNA剪接是高等真核生物基因表达所必须的一步,由剪接体催化完成。虽然剪接反应的化学本质仅为两步转酯反应,但是由于需要保证翻译时可读框的准确,剪接产物必须保证单碱基的精确性。为了完成这一任务,从剪接位点的识别开始,剪接体要经历一系列中间状态的转换,目前已鉴定出了E、A、pre-B、B、Bact、B*、C、C*、P和ILS complex十个中间状态(下图)。在这一系列状态转换过程中,剪接体的蛋白和snRNA组成及结构都发生了巨大变化,转酯反应的催化活性中心也在这一过程中逐步形成【1-3】。
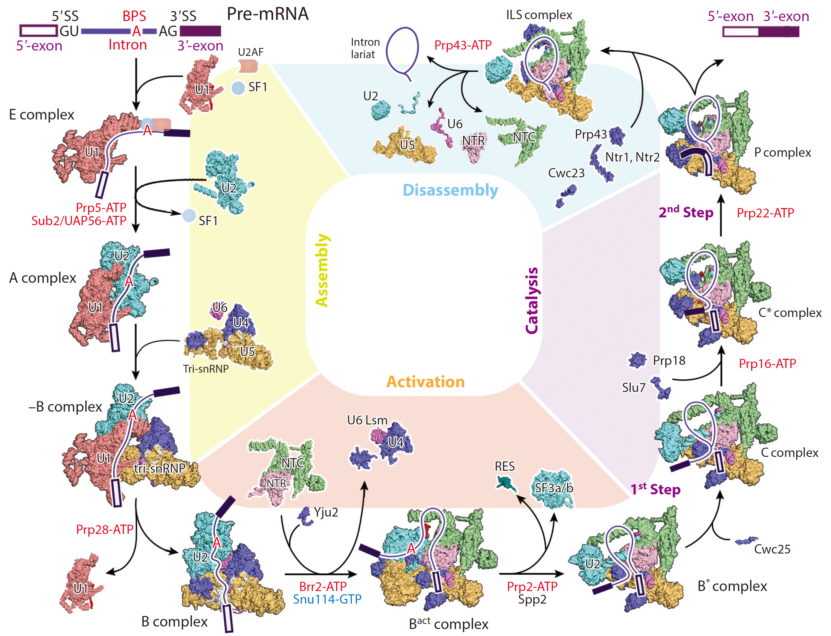
图片来源:Wan R, et al, Annu Rev Biochem, 2020
剪接体是一种核酶,特定折叠的U6 snRNA催化转酯反应【4】。虽然剪接体Bact complex的电镜结构揭示了活性中心U6 snRNA的折叠结构【5-7】,但它是如何正确折叠形成的、以及后续状态转换的动力机制细节,目前并不清楚。
2020年11月27日,德国马普研究所Reinhard Lührmann教授团队和西湖大学施一公教授团队发表在Science上的两项工作,分别解析了剪接体pre-Bact complex和包含Prp2和Spp2的Bact complex的电镜结构,从而回答了这两个问题。


Reinhard Lührmann教授团队的工作Mechanism of protein-guided folding of the active site U2/U6 RNA during spliceosome activation,通过使用一种新的抑制剂将人剪接体阻滞到了B complex和Bact complex间的一个新的状态,pre-Bact complex。电镜结构解析得到了两个pre-Bact complex的结构,为B complex转变到Bact complex间的两个连续中间体,从而能够从蛋白组成、新加入蛋白的结合部位以及snRNA结构的变化,直观推断出剪接体的蛋白组分如何帮助U6 snRNA正确、有序的折叠成剪接活性中心(下图)。
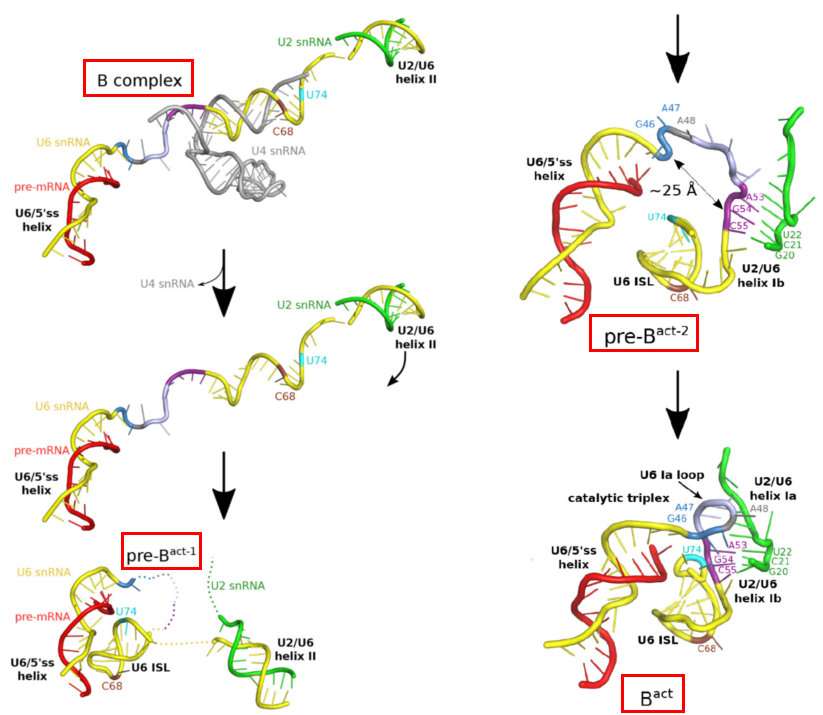
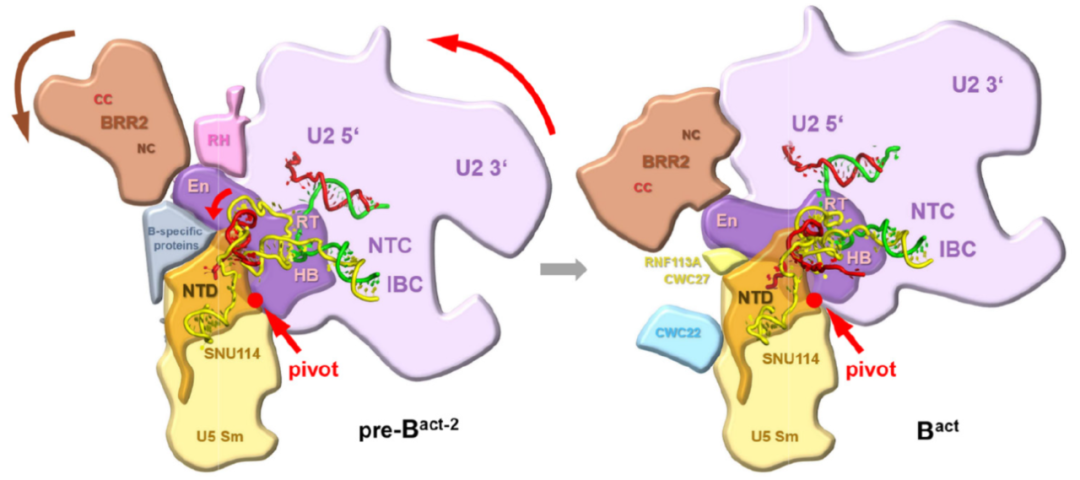
在这一过程中,RNA解旋酶Brr2剥离U4 snRNA提供早期动力,Prp8起核心支撑锚定作用,Prp8构型的变化伴随着其它蛋白的有序加入和离开,帮助U6 snRNA折叠成正确的活性中心结构(下图)。
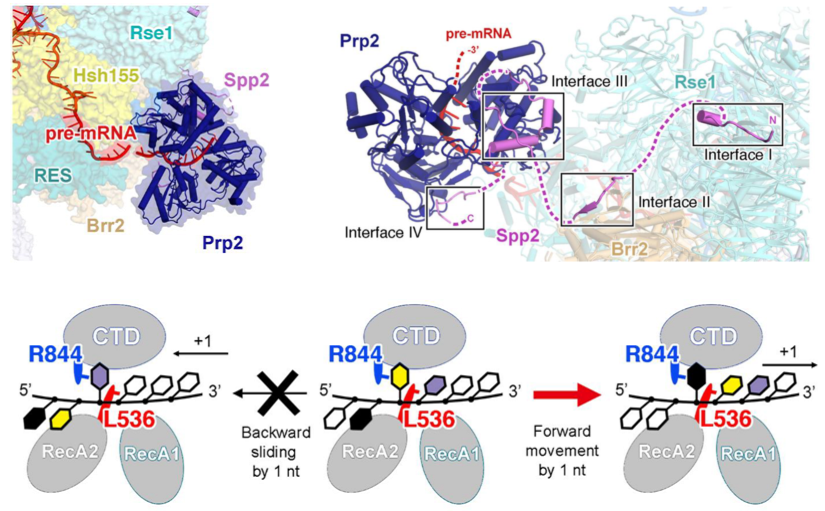
施一公教授团队的工作Mechanism of spliceosome remodeling by the ATPase/helicase Prp2 and its coactivator Spp2,通过使用新的阻滞方法得到了大量Bact complex,从而获得了高分辨率的酿酒酵母剪接体Bact complex的电镜结构。在这一新的结构中观察到了以前未在结构研究中观察到的RNA解旋酶/ATP酶Prp2、其激活因子G-patch蛋白Spp2及与Prp2结合的底物pre-mRNA。从而结合生化实验,阐明了剪接体Bactcomplex重塑的发动机Prp2如何被募集到剪接体上、Spp2如何稳定Prp2以及Prp2为何具有使底物pre-mRNA向其3’端单向移动的方向性等关键科学问题(下图)。
近些年结构生物学的进步极大推动了剪接体反应机制的研究,从早年依靠遗传、生化实验进行推断到现在能够直观的从结构上进行观察阐释。但是由于剪接体高度的动态性以及剪接位点较低的一致性,尤其在哺乳动物中,还有许多机制细节有待阐明,需要更多高分辨率的结构研究来进一步完善。
原文链接:
1. https://science.sciencemag.org/lookup/doi/10.1126/science.abc3753
2. https://science.sciencemag.org/lookup/doi/10.1126/science.abe8863
1. Wan, R., Bai, R., Zhan, X. & Shi, Y. How Is Precursor Messenger RNA Spliced by the Spliceosome?Annual review of biochemistry 89, 333-358, doi:10.1146/annurev-biochem-013118-111024 (2020).
2. Wilkinson, M. E., Charenton, C. & Nagai, K. RNA Splicing by the Spliceosome. Annual review of biochemistry 89, 359-388, doi:10.1146/annurev-biochem-091719-064225 (2020).
3. Kastner, B., Will, C. L., Stark, H. & Lührmann, R. Structural Insights into Nuclear pre-mRNA Splicing in Higher Eukaryotes. Cold Spring Harbor perspectives in biology 11, doi:10.1101/cshperspect.a032417 (2019).
4. Fica, S. M. et al. RNA catalyses nuclear pre-mRNA splicing. Nature 503, 229-234, doi:10.1038/nature12734 (2013).
5. Yan, C., Wan, R., Bai, R., Huang, G. & Shi, Y. Structure of a yeast activated spliceosome at 3.5 Å resolution. Science (New York, N.Y.) 353, 904-911, doi:10.1126/science.aag0291 (2016).
6. Zhang, X. et al. Structure of the human activated spliceosome in three conformational states. Cell research 28, 307-322, doi:10.1038/cr.2018.14 (2018).
7. Haselbach, D. et al. Structure and Conformational Dynamics of the Human Spliceosomal B(act) Complex. Cell 172, 454-464.e411, doi:10.1016/j.cell.2018.01.010 (2018).
















