四轴散点图就是利用坐标系的四个轴,填充两组信息进去,比如左下两个轴映射一组散点图,右上两个轴映射一组散点图。这个图在桌面绘制工具如Origin中是很好绘制的。但是如果要使用ggplot2绘制,无法直接得到。
gtable包
是可以个性化处理ggplot2对象的包,本文的四轴散点图也是通过gtable来组合的两个散点图。
为了更好的组合两个散点图,需要使用cowplot包对grob对象进行对齐操作。
原始散点图
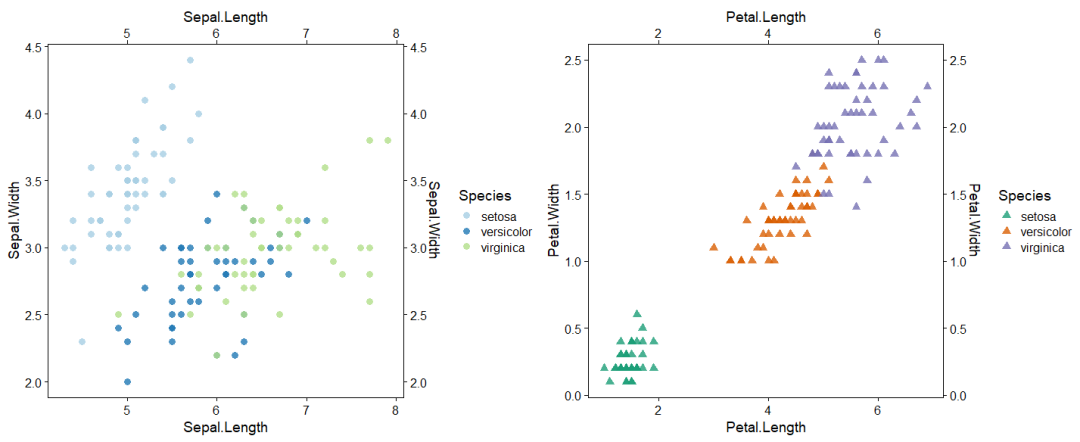
使用内建数据集iris绘制两张散点图,如下图。
不同于普通的散点图,每个图在右、上添加了次坐标轴(使用scale系列函数的sec.axis参数)。
library(gtable)
library(cowplot)# 绘图:添加两个次坐标轴
iris %>% ggplot(aes(x = Sepal.Length, y = Sepal.Width, color = Species)) +
geom_point(shape = 16, size = 3, alpha = 0.8) + theme_cowplot() +
scale_x_continuous(sec.axis = dup_axis()) +
scale_y_continuous(sec.axis = dup_axis()) +
scale_color_brewer(palette = "Paired") -> p1
iris %>% ggplot(aes(x = Petal.Length, y = Petal.Width, color = Species)) +
geom_point(shape = 17, size = 3, alpha = 0.8) + theme_cowplot() +
scale_color_brewer(palette = "Dark2") +
scale_x_continuous(sec.axis = dup_axis()) +
scale_y_continuous(sec.axis = dup_axis()) -> p2
cowplot::plot_grid(p1, p2)
对齐拼接并完成绘图
对齐图片
对ggplot2图片进行合并之前,要先让两图的坐标轴可以相互对齐。
这个工作可以使用cowplot包的align_plots来完成。
去除无用坐标轴和legend
第一张图的右、上坐标轴去除,第二张图的左、下坐标轴去除。同时去除legend。
# 1. 使用cowplot对齐绘图的部件
aln_plot
# 2. 第一张图删除它的右、上坐标轴及legend
# 第二张图删除它的左、下坐标轴及legend
g1 1]], c("axis-r", "axis-t", "xlab-t", "ylab-r", "guide-box"))
g2 2]], c("axis-l", "axis-b", "xlab-b", "ylab-l", "guide-box"))
拼接legend
如上图中显示的,两张图的legend的信息并不一致,因此需要拼接在一起后,再覆盖到第一张图的legend位置。
获取legend可以使用get_legend函数,拼接的话就要用到gtable(也可以使用cowplot)。
拼接legend后,再将legend放回第一张图的legend位置(已去除原来的legend)。
# 3. 获取legend
l1 1]])
l2 2]])
# 4. 将legend上下排列, 组合成一个gtable对象
g_legend 1,1), "null"),
widths = unit(1, "null"))
g_legend 1, l=1)
g_legend 2, l=1)
# 5. 将第一张图的legend替换为合并的legend
para_list 1]]$layout %>% filter(name == "guide-box") %>% c()
para_list[["x"]] para_list[["grobs"]] g1
绘图
最后使用cowplot绘图即可,结果如下:
# 6. 绘图














