目前人类应用的基因编辑工具中主要有三种:ZFN,TALEN和CRISPR-Cas。
20世纪90年代,科学家发现,一种广泛存在的蛋白结构模块锌指(ZF)能够介导蛋白质与核酸间的相互作用,将这些模块化的锌指串联起来,所形成的蛋白能够特异性识别并结合DNA,这种蛋白就是锌指蛋白(ZFP)。研究发现,锌指蛋白可以实现对DNA的识别,由核酸酶进行精准切割。
这也是第一个基因组编辑技术—锌指蛋白核酸酶技术(ZFNs)。
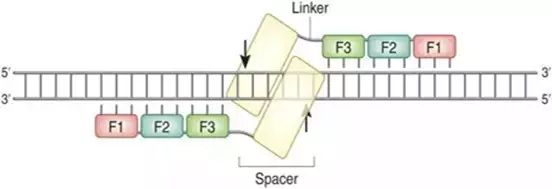
ZFN特异性识别DNA并与DNA结合示意图。每个DNA识别域包含三个锌指,锌指从N端开始命名,在图中标示为F1、F2、F3。每个锌指结构分别与3个碱基发生直接接触,由此产生特异性。单独的FokI切割域不具有特异性识别能力,但当与锌指结构相连,并与另一个FokI切割域形成二聚体后,便能够对DNA双链进行切割。
TALE是一种从植物病原体—单胞杆菌中分离出来的蛋白,这种蛋白能够通过特异性结合宿主DNA,调控其表达来提高病原体的致病性。
TALE蛋白与锌指蛋白最大的不同体现在,TALE蛋白的重复单元与DNA的碱基是按照一对一的识别方式特异性识别的。而且相比使用锌指蛋白靶向DNA序列更加稳定,特异性也更高且不受序列的限制。
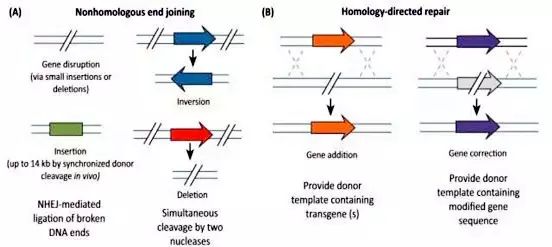
TALEN进行基因组编辑的原理。利用位点特异性核酸酶可以进行基因组编辑,而核酸酶诱导的DNA双链断裂(DSB)可由同源定向修复(HDR)或非同源末端连接途径(NHEJ)来修复。
20世纪80年代末,研究人员在观测大肠杆菌时发现,在细菌基因的尾端,有一些看上去很奇怪的重复序列。这些序列随后被命名为
成簇的规律间隔的短回文重复序列(CRISPR)
。病毒性感染就像定时炸弹,在它“爆炸”之前,细菌只有很短的时间来处理,而CRISPR就是一个“拆弹专家”。
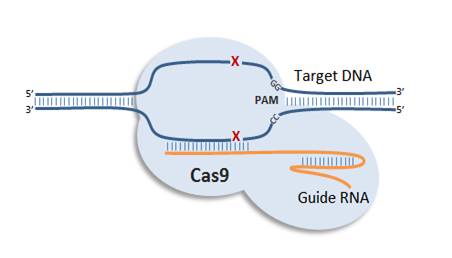
CRISPR-Cas9
那CRISPR是如何“拆弹”的?
研究人员注意到,这些重复的序列之间总是由一些很古怪的间隔区隔开,这些间隔区之所以看上去很古怪,是因为它们根本就不是细菌自身所有的东西,而是从噬菌体病毒的DNA上“剪”下来的小片段。
简言之,细菌细胞产生CRISPR相关蛋白(Cas蛋白),在病毒入侵之后,Cas蛋白便会结合到病毒DNA上,从上面“剪”下一块病毒DNA,然后将其转运到细菌细胞的基因组,插入其中,使之成为一处“间隔区”。
从此以后,细菌细胞便会利用这一间隔区来识别与之相对应的病毒,实现对病毒再次入侵的免疫应答。更神奇的是,CRISPR系统还可以将获得的部分DNA片段整合进基因组,形成记忆并遗传,从而可以保护后代的细胞免受病毒的攻击,就像随身带了一张基因的“疫苗接种卡”。
CRISPR的登场,把基因治疗的武器库升级到一种前所未有的高度。更便宜、更便捷、靶向更加准确。
5月30日,《NatureMethods》期刊上的一篇题为 Unexpected mutations after CRISPR-Cas9 editing in vivo的一篇通信文章显示,来自美国哥伦比亚大学等研究机构的科学家确认通过CRISPR基因编辑技术成功修复了导致两只小鼠失明的基因后,经全基因组测序发现小鼠体内有超过1500个单核苷酸发生突变,并有百个以上的位点发生发生了大片段的缺失和插入。
如何规避“脱靶”风险?
近日,《Nature Methods》同时刊登的两篇研究中,发表了两种分CRISPR/Cas9基因组编辑脱靶效应的新方法。其中一种方法可以鉴定细胞类型特异性SNP相关的脱靶突变,另一种则可以检测潜在脱靶切割位点。
第一项研究:来自麻省总医院(MGH)和哈佛大学的研究人员开发了一种新的
体外筛查方法CIRCLE-seq
,该方法是一种高度灵敏的体外筛查法,比现有识别CRISPR/Cas9全基因组脱靶突变的细胞学或生化方法更有效,表现更佳。
为了检测新方法的灵敏性,研究人员将靶向HBB基因的sgRNA的脱靶结果与利用另一种方法Digenome-seq识别的脱靶结果进行比较。结果发现CIRCLE-seq 鉴定出Digenome-seq已检测到的29个位点中的26个,并且还检测到156个其他方法无法检测的新位点。
研究人员表示,与Digenome-seq检测到的结果相比,CIRCLE-seq具有更高的信噪比,而且使用的测序reads减少了一百倍,这可能是由于该技术在识别全基因组范围脱靶位点方面具有更高的灵敏度。

Caribou Biosciences 公司 CEO Rachel Haurwitz
第二项研究:在同一期的杂志上,CaribouBiosciences和DuPont Pioneer的研究人员介绍了另一种
名为SITE-Seq的生化方法
,它利用通过向导RNA编程的Cas9来鉴定基因组内的切割位点。该技术使用了sgRNA 编程的Cas9对基因组DNA中的剪切位点序列进行识别。
研究人员表示:“我们使用 SITE-Seq检测靶向人类基因组的sgRNA与Cas9的特异性。识别的位点数量依赖于sgRNA序列和核苷酸浓度。低浓度条件下鉴定到的位点更有可能是细胞中的脱靶突变。细胞中具有活性的脱靶位点会受到sgRNP传递、细胞类型和在核苷酸环境接触的持续时间影响。
总而言之,我们的结果强调了在评估核苷酸活性和特异性时,将全面的脱靶位点生化识别方法与基于细胞的活性检测方法结合的有效性。”
其实在CRISPR-Cas9问世之初,大家就认识到存在脱靶效应的存在,如果克服了这一难题,那么CRISPR技术用于人类治疗的安全性就能得到很大的提高。由此我们可以知道,在保持CRISPR技术优势的基础上,努力突破其限制壁垒将是未来几年基因编辑技术发展的大趋势。
对此,CaribouBiosciences 公司 CEO Rachel Haurwitz 表示:“通过向研究人员提供一种精确定位CRISPR-Cas 活性的基因组区域,我们可以选择活性和特性行最高的靶点来减少脱靶效应的发生,从而使CRISPR-Cas9成为一种安全且有效的基因编辑工具。”
根据美国国家卫生研究院(NIH)的说法,美国重组DNA顾问委员会(Recombinant DNA Advisory Committee,RAC)下周将审查宾夕法尼亚大学申请首次利用革命性的基因编辑技术CRISPR治疗人类癌症的临床试验。利用CRISPR技术,科学家们能够准确地切割靶DNA。
这项临床研究将从癌症患者体内提取出免疫系统的T细胞。接着,研究人员将利用CRISPR对T细胞进行基因修饰,并将基因修饰后的T细胞灌注回病人体内,这样它们将靶向摧毁肿瘤细胞。

由于在许多生物医学和生物技术领域均有着广泛的应用,“基因魔剪”CRISPR/Cas9或将完全打开癌症研究领域的大门;日前一项刊登在国际杂志Journal of the National Cancer Institute上的研究报告中,来自德国德累斯顿工业大学 (Dresden University of Technology)等机构的研究人员通过研究发现,扮演癌症驱动子的突变或许能够被靶向作用并且修复,而且这些相关的突变也可以被快速诊断,并被用来改善个体化疗法。
作为生物技术研究领域的革命性工具,CRISPR/Cas9在生物医学研究上有着其广泛的用途,其可以实现对细胞基因组中特定位点的DNA进行切割,如今研究人员就发现了一种方法,能够利用该技术诊断并且使得癌症突变失活,从而加速癌症领域的研究。研究者Frank Buchholz说道,通过新一代测序技术我们就能够快速鉴别出癌细胞中的突变,但很多时候我们并不知道到底是哪些突变能够驱动疾病的发生,而且哪些突变是相对良性的。
这项研究中,研究者首先对超过50万个报道的癌症突变进行分析,这些突变从理论上来讲能够被靶向作用,并且超过80%的突变都可以被CRISPR/Cas9系统进行切割修饰;随后研究者人员发现CRISPR/Cas9可以在不明显靶向作用健康野生型等位基因的同时,对一系列常见的癌症突变进行特异性靶向作用;此外,携带癌症特异性引导RNAs的Cas9酶类的表达还能够揭开引发癌细胞生长和变异的突变。