文章主要描述了在知道蛋白质ID号的情况下,如何通过Uniport网站和其他资源找到对应的基因ID。重点介绍了在R操作中如何利用Uniport提供的ID mapping文件,针对拟南芥的基因组ID进行转换。
作者尝试使用Uniport网站的ID转换服务,但遇到了两个问题:一是需要转换的蛋白质ID较多,不便手动复制粘贴;二是网站不提供转换成Araport的服务。
作者通过搜索找到了Uniport提供的ID mapping文件下载地址,并成功下载完整的文件。导入到R语言后,通过筛选得到了所需的Araport对应的基因ID。
介绍了使用read.table函数读取ID mapping文件,然后使用subset函数筛选得到Araport对应的基因ID。最后展示了转换结果的格式。
最近有个需求,就是给定一个蛋白质的ID号,就知道它是什么基因。
比如说
O49397
。
但有个问题是,我并不知道这是个什么东西,于是我就去搜了下。
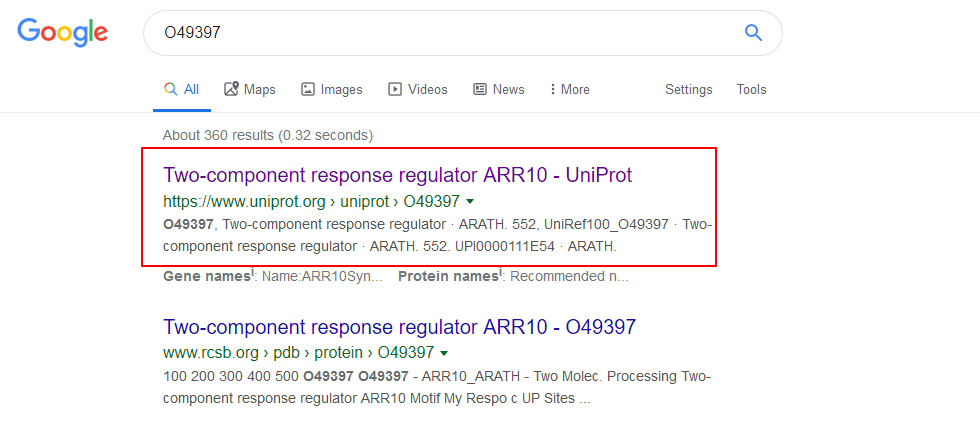
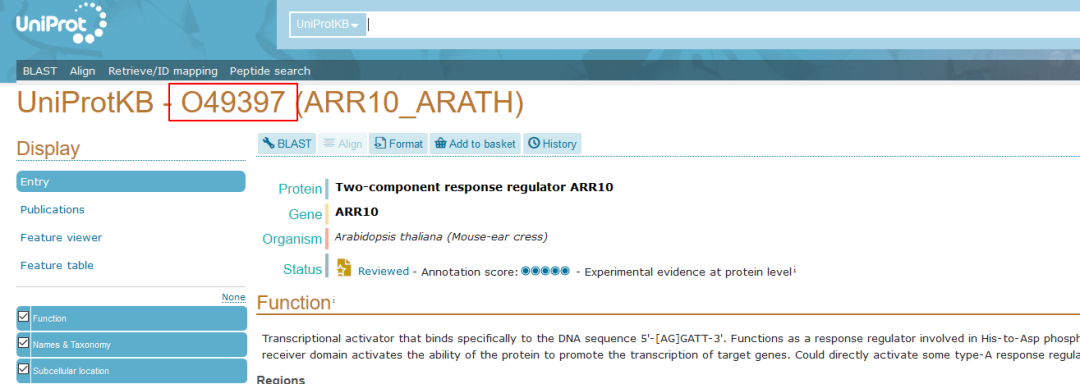
发现这其实是Uniport网站的蛋白质ID。
然后发现网站还提供了ID转换的服务(https://www.uniprot.org/uploadlists/),但发现了几个问题
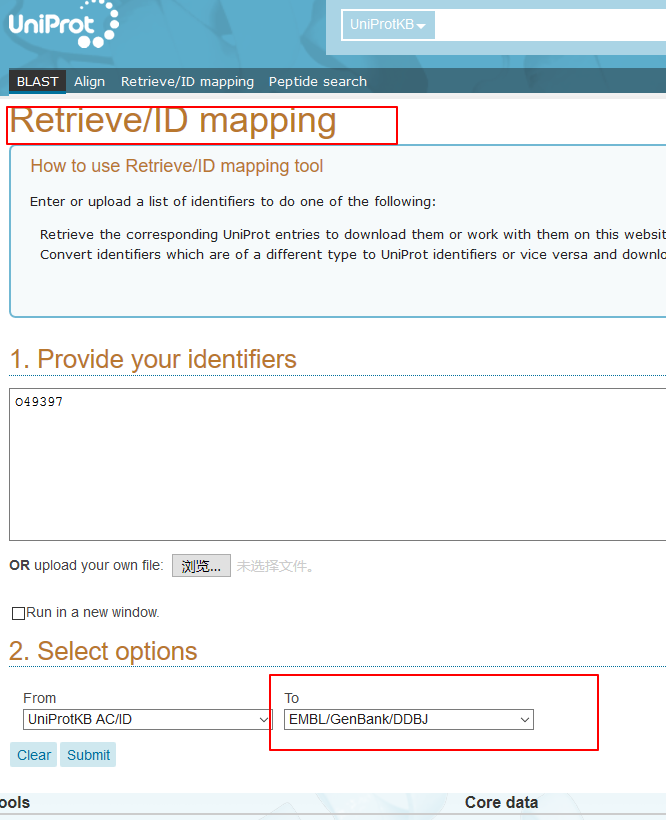
一个问题是我的蛋白质ID有很多且来自于R操作中的中间步骤,所以不想复制粘贴到那个框里面,其次是To那里实际上不提供转换成Araport的服务(植物没人权╮(╯_╰)╭)。所以不得不去寻找其他方法。
我想到Uniport应该是提供了ID mapping的文件下载的,就去搜索了下,果然找到了。
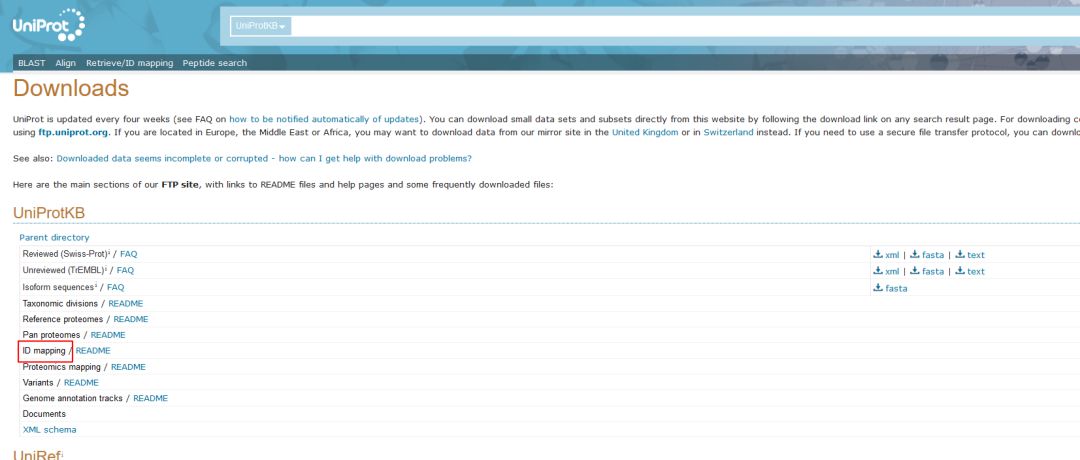
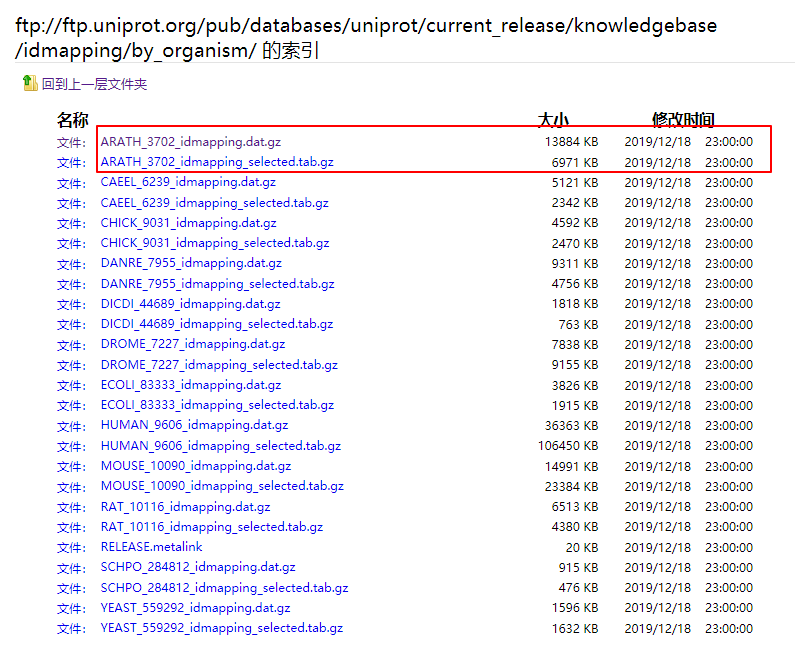
3702其实就是拟南芥的基因组ID。下载地址(ftp://ftp.uniprot.org/pub/databases/uniprot/current_release/knowledgebase/idmapping/by_organism/)
这里可以选select也可以选没有完整的。我这里选了完整的。下完了之后就可以导入到R里面进行操作了。
# 因为我们只想转Araport对应的基因ID,所以只要选择Araport那里就行
uniport_map read.table("~/reference/annoation/Athaliana/uniport/ARATH_3702_idmapping.dat",
header = F,
fill = T,
stringsAsFactors = F)
uniport_map subset(uniport_map,V2 == "Araport")
# 出来的格式就是
> head(uniport_map)
V1 V2 V3
50 P48347 Araport AT1G22300
106 Q9S9Z8 Araport AT1G34760
149 Q9C5W6 Araport AT1G26480
205 P42643 Araport AT4G09000
256 Q01525 Araport AT1G78300
324 P42644 Araport AT5G38480
这样我们就可以根据蛋白ID批量提取拟南芥基因ID了。





