⌈ 医见生信 ⌋ 是中南大学
“
医学遗传与生物信息课题组
”
运营的公众号,课题组利用生物信息、深度学习等方法开发精准解读基因变异、鉴定候选基因的新工具、新平台及数据库,并应用到
孤独症、帕金森病为代表的神经精神类疾病,
探究疾病发生发展的本质规律及发病机制,为其精准防控和诊疗提供科学依据。在医见生信,
我们每周都会分享
组会论文
、
生信技术
或生信心得,您可以订阅任何您感兴趣的合集。此外,
您还可以订阅我们团队已发表的
代表性论文
,以及我们开发的整合疾病表型数据与遗传信息的
分析工具和数据库
。本课题组长期
招聘
生物信息、计算机、医学遗传、流统等方向的研究助理、博士后、技术员和全职科研人员,也欢迎相关背景的优秀本科生
攻读课题组研究生学位
,期待您的加入~
PubTator 3.0是一个生物医学文献资源挖掘网站工具,使用最先进的人工智能技术为蛋白质、遗传变异、疾病和化学品等关键概念提供语义和关系搜索。
它目前提供了超过10亿个实体和关系注释,涵盖了来自PMC开放获取子集的约3600万篇PubMed摘要和600万篇全文文章,每周更新。PubTator 3.0检索的文章数量比PubMed或Google Scholar更多。文章进一步表明,将ChatGPT(GPT-4)与PubTator API集成可以显着提高其响应的真实性和可验证性。
图1 摘要图
总而言之,PubTator 3.0提供了一套全面的功能和工具,使研究人员能够浏览不断丰富的生物医学文献,加快研究速度并为科学发现解锁有价值的知识。主要贡献:
(1)问答
:将ChatGPT(GPT-4)与PubTator API集成可以显着提高其响应的真实性和可验证性;
(2)知识发现
:发现文献中信息的潜在关系(例如蛋白质功能);
(3)支持其他文本挖掘任务
:通过PubTator API支持程序化访问;
(4)目标优先级
:优先提供包含候选基因、基因-表型关联以及疾病共病的相关文献;
(5)生物信息整理辅助
:生物信息整理工作通常涉及大量文献、数据库和实验数据的整合与分析,而NLP技术可以帮助研究者从这些海量数据中提取有用的信息,提高数据处理的效率和准确性。
Key issues and research motivation
(1)文献数量庞大,手动分析效率低
:生物医学文献是满足生物和临床科学信息需求的主要资源,命名实体识别模型和关系抽取模型等可加速文献中知识的挖掘过程,节省人力资源;
(2)传统的关键词搜索具有局限性
:传统的基于关键词的搜索方法长期以来是生物医学文献搜索的基础,但有很大的局限性,例如由于术语不同而丢失相关文章,或者由于表面级术语匹配无法充分表示查询术语之间所需的关联而包括不相关的文章;
(3)人工智能技术赋能知识挖掘
:自然语言处理(NLP)方法为创建生物信息学资源提供了巨大的价值,并可以通过启用语义和关系搜索来改进文献搜索。
3.1 PubTator3.0 平台工具主要功能
(A)检索功能:可对文献中六种生物医学实体并进行自动注释:基因,疾病,化学物质,遗传变异,物种和细胞系,以及根据文献PMID或实体名称快速检索相关文献资源;
(B)关系挖掘:识别并可搜索实体之间的12种(化学-化学、化学-疾病、化学-基因、化学-变异、疾病-基因、疾病-变异、基因-基因和变异-变异等)常见关系,增强了其在实体关系的针对性和探索性搜索的实用性。
图2 平台文献阅读功能截图
平台的数据流程如下图所示,具体流程在下文逐一介绍:
图3A 平台处理流程图
3.2 数据来源
PubTator 3.0每周从BioC的PubMed API
https://www.ncbi.nlm.nih.gov/research/bionlp/APIs/BioC-PubMed/
和BioC PMC API
https://www.ncbi.nlm.nih.gov/research/bionlp/APIs/BioC-PMC/
下载新的文章,这些文章采用BioC_XML格式获取。
平台现已包含超过 3600 万 PubMed 摘要和 600 万全文文章,每周更新。
3.3 命名实体识别—AIONER模型
PubTator 3.0使用AIONER 模型,来快速识别定位文献中以下类型的实体信息:基因、蛋白质、化学物质、疾病、物种、遗传变异和细胞系;
3.4 实体名称标准化
(1)GNorm 2系统:将基因名称归一化为NCBI基因标识符,并将物种分类归一化为NCBI分类体系。;
(2)tmVar 3工具:将遗传变异信息正常化,对于dbSNP标识符和HGNV格式列出的变异,使用dbSNP标识符进行归一化;
(3)NLM-Chem标记器:将化学物质通过NLM-Chem标记器归一化为MeSH(医学主题词表)标识符;
(4)TaggerOne:使用一种新的仅归一化模式将疾病正常化为MeSH,将细胞系正常化为Cellosaurus数据库的名称。
通过将每一个识别到的实体的名称映射到每个词典名称来增强训练数据,从而大大提高了词典中存在但未在带注释的训练数据中的名称的性能,显著提高了实体规范化性能。
3.5 实体间关系抽取
使用BioREx模型进行关系提取:通过8个实体类型对提取12种关系:化学-化学、化学-疾病、化学-基因、化学-变异、疾病-基因、疾病-变异、基因-基因和变异-变异等关系。
3.6 数据存储与平台搭建
文章正文与提取的数据均使用MongoDB进行内部存储,并通过Solr进行索引以便于搜索,从而确保了稳健且可扩展的访问性,且不受NCBI eUtils API等外部依赖的限制。
在以上数据的基础上搭建用户平台。
图4 PubTator 3.0系统构建及搜索结果页面:(1)查询自动补全功能提升了搜索精确度和同义词匹配度。(2)基于自然语言处理(NLP)的相关性增强:搜索结果根据查询实体间关系的紧密程度进行优先级排序。(3)用户可通过分类筛选器(包括部分、期刊和类型)进一步精炼搜索结果。(4)搜索结果中包含高亮显示的实体片段,用以解释其相关性。(5)直方图以出版年份为维度,直观展示了结果的数量分布。(6)用户可根据个人偏好开启或关闭实体高亮功能。
4.1 实体识别与关系抽取可靠性实验
图3 B&C 实体识别与关系抽取F-score值
BioRED语料库上的PubTator2(也称为PubTator中心)相比,每个实体类型的(B)实体识别性能实体注释通过专门的映射器与数据库标识符相关联;关系提取性能与SemRep等之前显著的最佳系统相比较on the BC5CDR数据集。
4.2 文献检索质量对比
对比了PubTator 3.0、PubMed和Google Scholar的检索质量。
(1)准备用于查询测试的12个实体对(包括疾病/基因、化学物/疾病、化学物/基因、化学物/化学物、基因/基因和疾病/变异) ,例如“GLPG0634 + ulcerative colitis”(GLPG0634与溃疡性结肠炎)等。
(2)手动评估了每个系统和每个查询的前20个结果:
如果文章提及了查询中的两个实体并支持它们之间的关系,则判断该文章为相关;
两位评估员独立评估每篇文章,并对分歧进行讨论直至达成一致
(3)实验评估了每种检索方法前20个结果的相关性
图3 D 检索文献数量以及正确率结果
比较PubTator 3.0、PubMed和谷歌Scholal对实体对查询的信息检索,包括文章总数和前20篇文章精度。
在12个信息检索案例研究中,PubTator 3.0对前20篇文章的整体精确度达到了90.0%(240篇中的216篇),显著高于PubMed的81.6%(103篇中的84篇)和Google Scholar的48.5%(202篇中的98篇)。
这些差异可归因于以下因素:
(1)PubTator 3.0的搜索范围包括PMC-OA中的全文,因此文章覆盖范围显著更广;
(2)实体归一化提高了召回率,例如,将“paraoxonase 1”(对氧磷酶1)与“PON1”相匹配;
(3)PubTator 3.0优先返回包含查询实体之间关系的文章;
(4)PubTator 3.0优先返回实体出现在附近段落而非远距离段落中的文章。
4.3 对ChatGPT-4进行检索增强生成
PubTator增强的GPT-4:
这一整合涉及向GPT-4提供关于三个PubTator API的描述:
(i)查找实体ID,用于检索PubTator实体标识符;
(ii)查找相关实体,基于输入实体和指定关系来识别相关实体;
(iii)导出相关搜索结果,返回包含特定实体关系文本证据的PubMed文章标识符。
我们的指令引导GPT-4将用户问题分解为可通过这些API解决的子问题,执行函数调用,并将回答综合成连贯的最终答案。
例如,为了回答“哪些药物可以治疗乳腺癌?”
(i)GPT-4首先使用查找实体ID API找到乳腺癌的PubTator实体标识符(@DISEASE_Breast_Cancer);
(ii)然后,它使用查找相关实体API通过“治疗”关系来识别与@DISEASE_Breast_Cancer相关的实体;
(iii)最后,GPT-4调用导出相关搜索结果API,获取包含这些关系证据的PubMed文章标识符。
图5 检索增强生成结果对比
仅使用 GPT-4、GPT-4 和 GPT-4 与 PubTator 3.0 一起测试的所有 8 个查询的文章引用精度。结果总结为 “正确引用的文章数 / 引用的文章总数”。补充数据中提供了完整的回答。
4.4 提供API访问与数据格式
PubTator 3.0 通过其应用程序接口(API)提供程序化访问,并支持批量下载。API(网址:https://www.ncbi.nlm.nih.gov/research/pubtator3/)支持关键词、实体和关系搜索,还支持以基于XML和JSON的BioC格式以及制表符分隔的自由文本格式导出注释。
PubTator 3.0 文件传输协议(FTP)站点(网址:https://ftp.ncbi.nlm.nih.gov/pub/lu/PubTator3)提供已注释文章的批量下载以及实体和关系的提取摘要。程序化访问支持更灵活的查询选项;
例如,对于信息需求“哪些化学物质可以降低JAK1的表达?”,可以直接通过API(例如:https://www.ncbi.nlm.nih.gov/research/pubtator3-api/relations?e1=@GENE_JAK1&type=negative_correlate&e2=Chemical)查询,或者通过筛选批量关系文件来获取答案。
Conclusion and Inspiration
PubTator 3.0提供了一套全面的特性工具,使研究人员能够有效地导航不断扩大的生物医学文献资源,加速研究进程,并为科学发现解锁宝贵的见解。文献还提供了数据可用性信息,包括在线界面、API和批量FTP下载的链接,以及各个组件的源代码。
但PubTator 3.0同时仍具有局限性:
(1)实体和关系抽取的局限性:尽管PubTator 3.0已支持多种实体类型和关系类型,但都是数据预处理,预存储,而非实时处理,尤其是对未标注的新型实体和复杂关系的处理仍有挑战。
(2)自动化标注的准确性:尽管系统表现出色,但自动化标注仍会受到训练数据的影响,某些复杂情况可能导致识别错误或不准确的关系抽取。
(3)全文支持不足:目前PubTator的关系抽取功能仅限于文摘,未来可以扩展至全文文献,进一步提升处理的全面性。
喜欢我们,别忘了关注我们,除了分享记录 JC Lab 活动之外,我们也会在之后多多分享干货~记得点 ⌈ 赞 ⌋ 和 ⌈ 在看 ⌋ 哦~
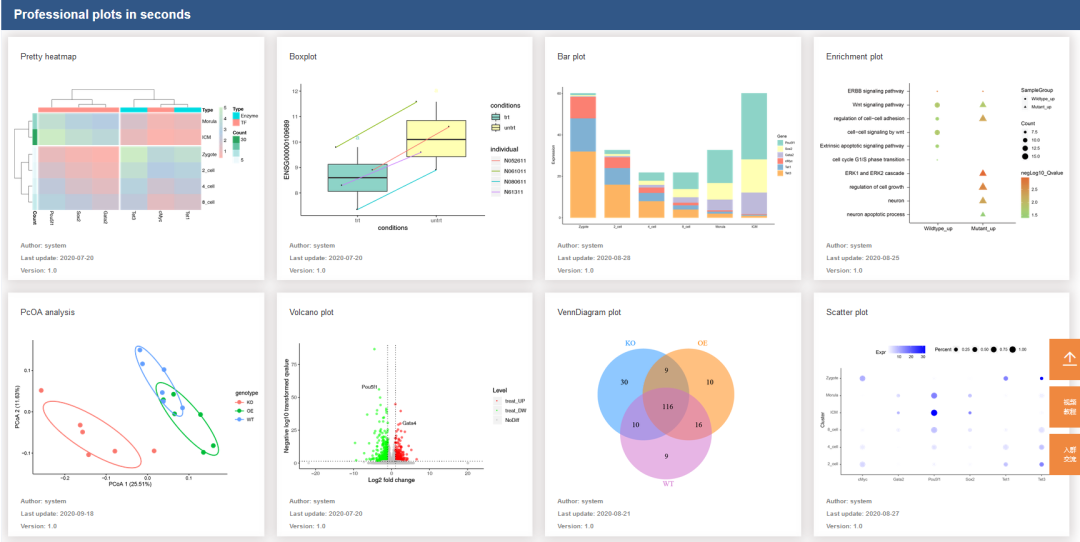
高颜值免费 SCI 在线绘图
(
点击图片直达
)
最全植物基因组数据库IMP
(
点击图片直达
)
往期精品
(
点击图片直达文字对应教程
)
机器学习