韦恩图
韦恩图是用来反映不同集合之间的交集和并集情况的展示图。一般用于展示2-5个集合之间的交并关系。集合数目更多时,将会比较难分辨,更多集合的展示方式一般使用upSetView。
较早的文章列举了多个在线工具轻松绘制各种Venn图。这篇文章讲解下如何用R代码一步出图。
韦恩图一步法
假设有这么一个矩阵,第一列为不同集合中的ID,第二列为集合的名字,无标题行,存储为venn.txt。
a ehbio1
b ehbio1
c ehbio1
d ehbio1
e ehbio1
f ehbio1
g ehbio1
h ehbio2
i ehbio2
j ehbio2
k ehbio2
e ehbio2
f ehbio2
g ehbio2
a ehbio3
b ehbio3
h ehbio3
j ehbio3
i ehbio3
f ehbio3
g ehbio3
a ehbio4
b ehbio4
h ehbio4
d ehbio5
e ehbio5
y ehbio5
x ehbio5
sp_vennDiagram.sh -f venn.txt -a ehbio1 -b ehbio2 -c ehbio3 -d ehbio4 -g ehbio5
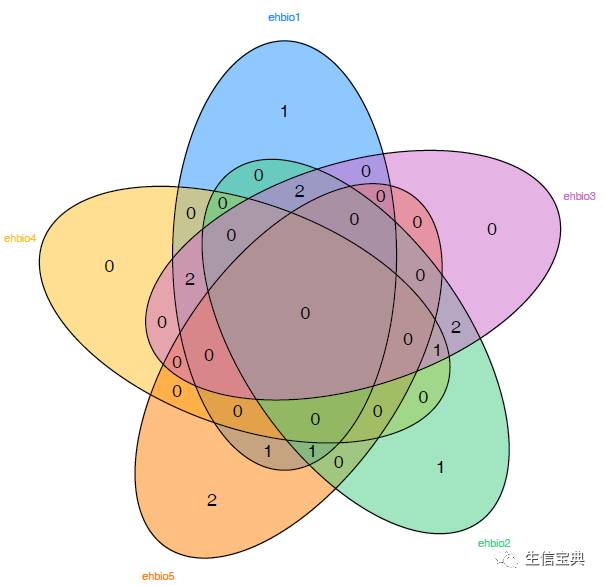
sp_vennDiagram.sh -f venn.txt -a ehbio1 -b ehbio2 -c ehbio3 -d ehbio5
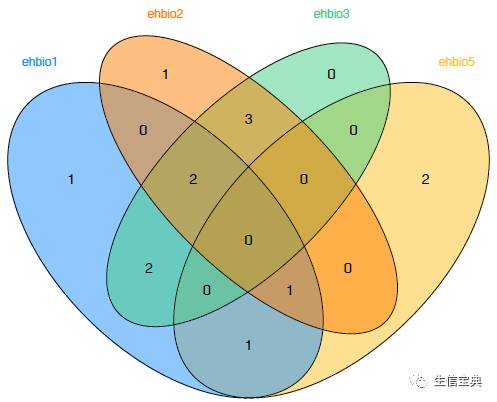
sp_vennDiagram.sh -f venn.txt -a ehbio1 -b ehbio2 -c ehbio3
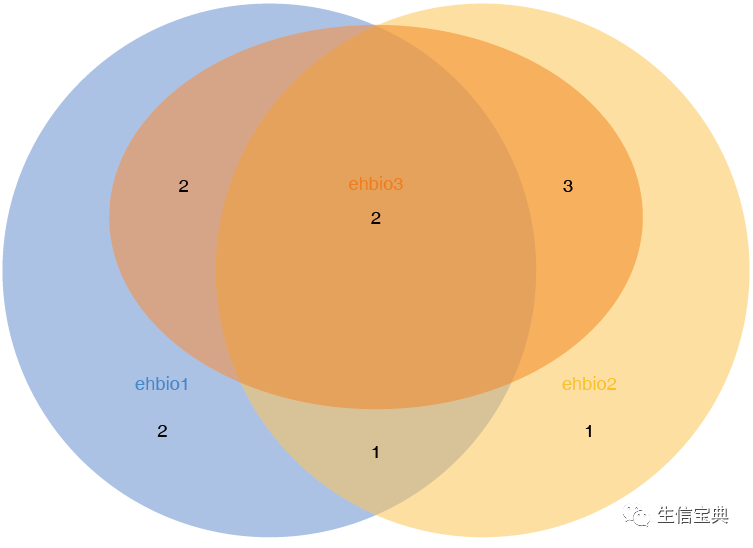
sp_vennDiagram.sh -f venn -F TRUE -n "120, 110, 50" -l "'EHBIO','SXBD'"
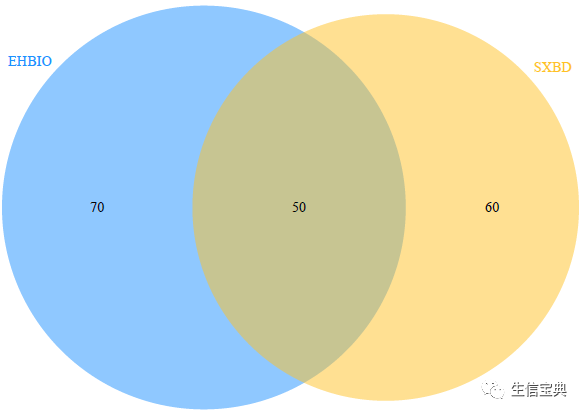
For two-set venn, the format is represents (length_a, length_b, a_b_overlap).
For three-set venn, the format is “100, 110, 90, 50, 40, 40, 20” represents (length_a, length_b, length_c, a_b_overlap, b_c_overlap, a_c_overlap, a_b_c_overlap).
For four-set venn, the format is represents (length_a, length_b, length_c, a_b_overlap, a_c_overlap, a_d_overlap, b_c_overlap, b_d_overlap, c_d_overlap, abc_overlap, abd_overlap, acd_overlap, bcd_overlap, abcd_overlap).
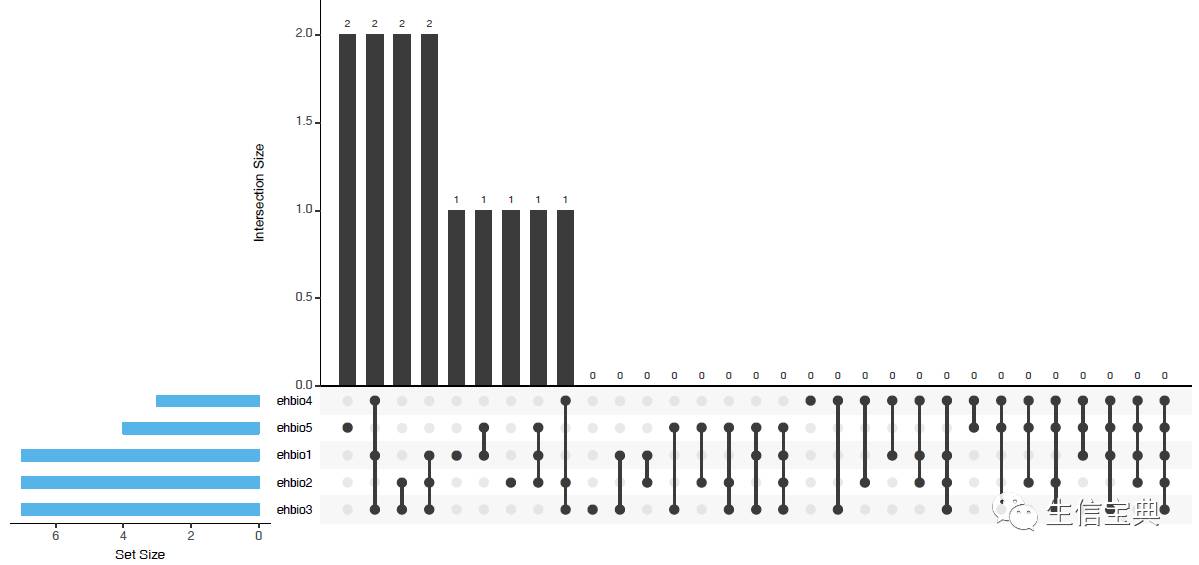
UpSetView展示
对于集合比较多的时候,包括上面提到的5个集合的交并集情况,如果只是为了展示个炫图,还可以,但如果想解释结果,就会比较头疼,难判断区域的归属。
因此对于这种多集合情况,推荐使用UpSetView展示,看效果如下。
测试数据,存储为upsetview.txt (第一行为集合的名,每个集合一列;每一行为一个ID,如果对应ID出现在这个集合则标记1,否则标记0):
pattern ehbio1 ehbio2 ehbio3 ehbio4 ehbio5
a 1 0 1 1 0
b 1 0 1 1 0
c 1 0 0 0 0
d 1 0 0 0 1
e 1 1 0 0 1
f 1 1 1 0 0
g 1 1 1 0 0
h 0 1 1 1 0
i 0 1 1 0 0
j 0 1 1 0 0
k 0 1 0 0 0
x 0 0 0 0 1
y 0 0 0 0 1
sp_upsetView.sh -f upsetview.txt
UpSetR: http://www.caleydo.org/tools/upset/ 采用连线的方式展示不同的组合之间共有的和特有的项目,对于特别多的组合尤其适用。
单个点表示特有,连起来的点表示共有,相当于venn图中重叠的部分。
垂直的柱子代表的是Venn图中的数字,看连接的点判断归属。
水平的柱子代表对应样品中Item的总数。
为了推广,也为了激起大家的热情,如果想要sp_vennDiagram.sh和sp_upsetView.r脚本,还需要劳烦大家动动手,转发此文章到朋友圈,并后台留言索取。
也希望大家能一起开发,完善功能。
Reference
R绘图学习
R语言学习 - 热图简化
R语言学习 - 线图绘制
R语言学习 - 箱线图(小提琴图、抖动图、区域散点图)
R语言学习 - 散点图绘制
程序学习心得
生物信息之程序学习
如何优雅的提问
加入易汉博,人生不白活

关注生信宝典,换个角度学生信

关注宏基因组,再专业一点
















