
在真核生物中广泛存在的MLL家族蛋白复合体,是一类重要的组蛋白甲基转移酶,催化组蛋白H3K4的甲基化。在人体内有至少六种MLL同源蛋白,根据序列和功能不同又分为三类:MLL1/MLL2,MLL3/MLL4以及SET1A/SET1B(在酵母中只有Set1复合体,又被称为罗盘复合物,COMPASS)。这些复合物作为转录共激活因子,通过
催化
H3K4
甲基化/
或通过招募其它转录相关蛋白与中介因子
,激活转录,
在发育过程中扮演了至关重要的角色
。而这些蛋白的突变,包括基因组易位使它与其它转录因子融合,则会造成多种疾病,如混合谱系白血病(MLL),淋巴瘤,Kabuki综合征等。解析这些复合体的结构,将有助于开发药物的新靶点,为患者带去福音。但由于该复合物结构复杂,对其结构的解析一直是一个很大的挑战。
2018年,斯坦福大学Georgios Skiniotis组的屈前辉博士等人以及华盛顿大学Ning Zheng组的Peter Hsu等人在Cell上同期分别发表了酵母中的COMPASS复合物的高分辨率的冷冻电镜结构和X射线晶体结构
[1, 2]
。这些工作向我们展示了具有完整甲基催化活性的COMPASS酶核心的组装机制,阐述了复合物各亚基调控催化能力的分子机制。然而,COMPASS/MLL复合物如何识别底物核小体仍不清楚。而本篇Nature由上海交通大学的黄晶与雷鸣课题组合作,发表了
人源
MLL1
与MLL3
复合物催化中心与核小体结合时
的多个高分辨率电镜结构。(图1)该结果与酵母中的结果相符,并且提供了MLL复合物与核小体互作的界面,以及H2BK120ub1增强MLL酶活的机制。
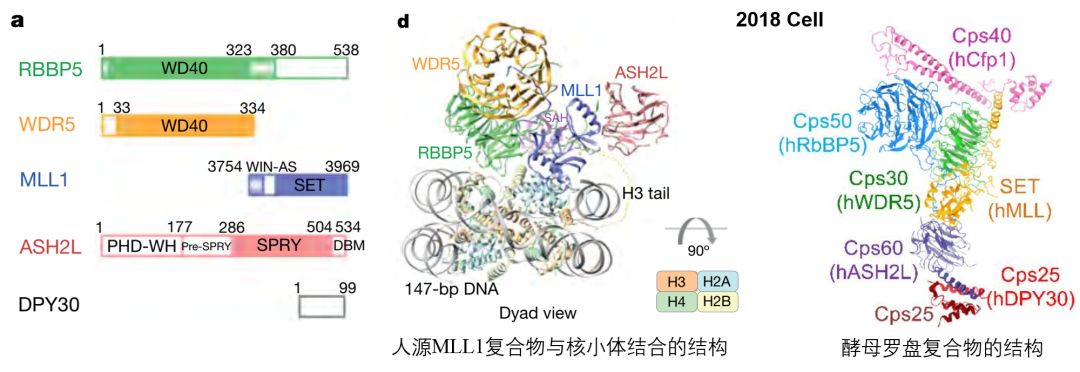
图 1 人源MLL1-H2B泛素化核小体复合物的结构。
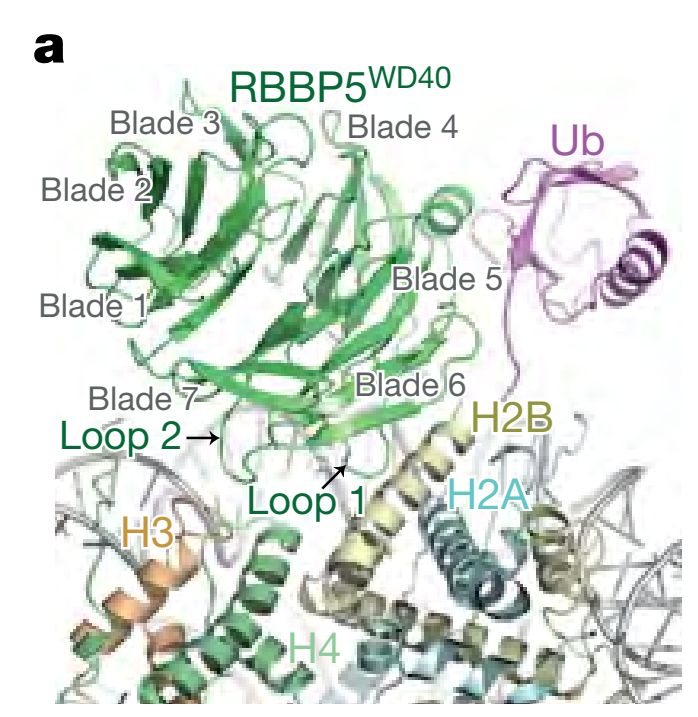
图 2 MLL1复合物与泛素化核小体结合的界面。a. RBBP5
WD40
与H2B–H4 及 H2BK120ub1结合。
核小体的泛素化H2BK120ub1可以促进H3K4甲基化的生成,为探明其机制,本研究采用带/不带H2BK120ub1的核小体进行研究。本研究主要发现:
1)人源MLL1-H2B泛素化核小体复合物的结构大体呈
Y
形
“横躺”在核小体上(图1d),与核小体有很大的接触面积,
MLL1
酶活域位于中央,ASH2L
的SPRY
结构域在最底部
。这与2018年Cell报道的酵母COMPASS复合物结构一致(图1最右)
[1]
。
2)
RBBP5
蛋白的WD40
结构域负责结合核小体的泛素化基团
,
嵌入H2B-H4的缝隙中,帮助MLL1复合物结合核小体(图2)。而去除该结构域将显著降低MLL1催化核小体H3K4甲基化的活性。
3)
MLL1
的催化域SET
也与H2A
的C
端有结合
,有助于MLL1复合物对核小体的识别。
4)
ASH2L
的pre-SPRY
结构域则与DNA
结合
,去除该结构域会使MLL1复合物不能结合核小体,催化活性消失。
5)
研究者又用未泛素化的核小体与MLL1复合物进行结合与结构解析
,发现其结合界面变少,催化效率(
k
cat
/
K
m
)降低2倍。

图 3 MLL1和MLL3复合物的结构不同。
有趣的是,本研究发现在MLL1复合物中,
MLL1
有一段“激活区域”会插入到WDR5
的桶状结构的中心
(图3c),与之前鉴定的RBBP5上的“活化片段”遥相呼应,同时删掉这两段序列则大幅度降低MLL1的催化能力。MLL1这一段“激活区域”并不存在于MLL3中,取而代之的是
MLL3
的SET
结构域氨基端接触WDR5
(图3f),这反而
抑制了
MLL3
的活性
,使其只能催化H3K4到
一甲基化
。去除WDR5会大大激活MLL3的活性,使其可以催化H3K4到二甲基化。(示意图见图3h)。
WDR5是第一个被发现的
组蛋白
H3K4
位点的阅读器(2005, Cell
)
,最初认为可以识别H3K4me2和me3,激活Hox基因簇,调控脊椎动物发育
[3]
。它的7个WD40重复序列组成的桶状结构,中央似乎刚好可以用来嵌入组蛋白尾巴。
2006
年
,有三个课题组解析了
WDR5
和H3K4
甲基化肽段的结晶
结构,结果说明WDR5的桶状结构在体外的确可以结合组蛋白尾巴
[4-6]
,但也发现
甲基化与否对
WDR5
的结合能力影响不大
[5, 6]
,修正了原有的认识。很快,
2008
年
,有3篇文章又报道
WDR5
可结合MLL1
[7-9]
,一时间WDR5究竟结合谁成了一道谜题。2018年报道的COMPASS冷冻电镜结构中发现WDR5的同源蛋白Cps30结合着Set1氨基端保守的Win序列
[1]
。本篇文章解析的MLL与核小体的复合物结构中,
WDR5
的桶状结构中嵌入的是MLL
蛋白的尾巴,而非组蛋白尾巴
。从这里我们可以看到,科研求真的道路时多么曲折。我也相信,越来越多的好的发现,会如黄金罗盘般指引我们的方向。
总的来说,本篇文章细致地揭示了
人源
MLL1
和MLL3
复合物结合核小体时
的结构,很好地阐明了各个蛋白精细调控蛋白结合与催化活性的分子机制,它们的协作将H3K4的甲基化修饰准确地加到了常染色质上,开放的染色质结构使得MLL和核小体“亲切接触“。本文也揭示了
H2BK120ub1
与H3K4
甲基化
这两种修饰间交流(
crosstalk
)的分子机制。本文与近期报道的DOT1L和PRC2复合物结合核小体的结构
[10-15]
一起,突出了结构生物学在理解组蛋白修饰中的重要作用,帮助我们更好地了解表观遗传学的调控机制。
之前报道的WDR5蛋白能识别H3K4不同甲基化程度的小肽,尽管在这些复合物结构中并未发现WDR5直接结合底物核小体,并不能排除其可能在染色质高级构造中发挥作用。
此外,对各类MLL发挥的不同生理作用的理解有待于进一步的研究。
屈前辉博士点评:
催化H3K4甲基化的蛋白酶Set1/MLL在真核生物中高度保守,且依赖于多个亚基组装形成高活性复合物:酵母中唯一的Set1/COMPASS复合物催化H3K4的单、双和三甲基化;而小鼠和人类细胞中至少包含六种Set1/MLL蛋白(MLL1-4和SETD1A/1B),敲除任何一种MLL都会导致小鼠死亡,意味着各自发挥着不可替代的生理功能。
冷冻电镜技术的快速发展极大地推动了对这些构造复杂的生物大分子的结构研究。继2018年8月Cell杂志上同期发表来自两个课题组的酵母COMPASS冷冻电镜(Qu
et al
.)和晶体结构(Hsu
et al
.)后,近期国际上其他研究团队又分别独立在Nature (Xue
et al
.; )和BioRxiv (Hsu
et al
.; Park
et al
.)上发表了酵母COMPASS或人源MLL与底物核小体复合物的冷冻电镜结构,进一步向我们展示了H3K4甲基化酶的作用机制:
-
酵母COMPASS和人源MLL整体构象类似,且结合核小体的方式大体一致,主要通过RbBP5、ASH2L和SET催化结构域识别核小体
-
与未修饰的核小体相比,H2B泛素化后的核小体增强了与COMPASS/MLL的亲和力,促进了H3K4甲基化
-
Xue
et al
.等人获得了人源MLL1和MLL3与核小体的多个复合物结构,发现存在于MLL1而非MLL3上一段特有序列(MLL1
AS
)结合WDR5和RbBP5,对催化活性很重要。他们还发现WDR5可以抑制MLL3的甲基化能力,这有可能解释为什么MLL3是单甲基化酶而MLL1则具三甲基化活力。此外,H2B上的泛素能结合RbBP5不同位置,意味着泛素化调节的动态变化。
-
Hsu
et al
.等人分析了酵母COMPASS与核小体的结构,发现Set1蛋白上一段富含碱性氨基酸序列ARM(在其他物种的高度相似的Set1蛋白中保守,但不存在于MLL1-4上)能形成螺旋结构介导泛素化的核小体与COMPASS的相互作用,而在没有泛素化的核小体复合物中则呈无规则结构。借此他们认为这段ARM序列的构象变化可能参与泛素化对COMPASS催化活性的调节过程。
-
Xue
et al
.等观察到泛素主要结合RbBP5蛋白的Blade5或Blade6,而Hsu
et al
.等发现泛素与Swd1(RbBP5同源蛋白)的氨基和羧基末端(靠近Blade7)作用,以及结合SET结构域的氨基端。此外,MLL1和COMPASS结合H2A的位置也略有不同。这有可能反映了不同的COMPASS/MLL复合物选择性识别底物核小体的特点。值得一提的是,这些样品都经过了化学交联处理,不同交联程度也可能产生一定差别。
总言之,这些研究工作首次揭示了COMPASS/MLL作用于底物核小体的分子机制,展现了H2B泛素化修饰影响H3K4甲基化的结构基础。
屈前辉博士简介:
主要从事表观遗传学与细胞信号传递领域研究。2012年获中科院生物物理研究所生化与分子生物学博士学位,2012-2017年在美国德州西南医学中心、密歇根大学进行博后训练,2017年获聘斯坦福大学研究科学家。2018年在Cell上发表酵母罗盘复合体结构解析。此外还在Science、Nature、Journal of Cell Biology等国际著名期刊上发表了多篇文章。
[1] QU Q, TAKAHASHI Y H, YANG Y, et al. Structure and Conformational Dynamics of a COMPASS Histone H3K4 Methyltransferase Complex[J]. Cell, 2018,174(5):1117-1126.
[2] HSU P L, LI H, LAU H T, et al. Crystal Structure of the COMPASS H3K4 Methyltransferase Catalytic Module[J]. Cell, 2018,174(5):1106-1116.
[3] WYSOCKA J, SWIGUT T, MILNE T A, et al. WDR5 associates with histone H3 methylated at K4 and is essential for H3 K4 methylation and vertebrate development[J]. Cell, 2005,121(6):859-872.
[4] HAN Z, GUO L, WANG H, et al. Structural basis for the specific recognition of methylated histone H3 lysine 4 by the WD-40 protein WDR5[J]. Mol Cell, 2006,22(1):137-144.
[5] COUTURE J F, COLLAZO E, TRIEVEL R C. Molecular recognition of histone H3 by the WD40 protein WDR5[J]. Nat Struct Mol Biol, 2006,13(8):698-703.
[6] SCHUETZ A, ALLALI-HASSANI A, MARTIN F, et al. Structural basis for molecular recognition and presentation of histone H3 by WDR5[J]. EMBO J, 2006,25(18):4245-4252.
[7] PATEL A, DHARMARAJAN V, COSGROVE M S. Structure of WDR5 bound to mixed lineage leukemia protein-1 peptide[J]. J Biol Chem, 2008,283(47):32158-32161.
[8] SONG J J, KINGSTON R E. WDR5 interacts with mixed lineage leukemia (MLL) protein via the histone H3-binding pocket[J]. J Biol Chem, 2008,283(50):35258-35264.
[9] PATEL A, VOUGHT V E, DHARMARAJAN V, et al. A conserved arginine-containing motif crucial for the assembly and enzymatic activity of the mixed lineage leukemia protein-1 core complex[J]. J Biol Chem, 2008,283(47):32162-32175.
[10] WORDEN E J, HOFFMANN N A, HICKS C W, et al. Mechanism of Cross-talk between H2B Ubiquitination and H3 Methylation by Dot1L[J]. Cell, 2019,176(6):1490-1501.














