相信不少老师跟同学都碰到过下面的问题:自己的项目课题完成了,SCI文章也已经发了一篇,终于喊出憋了很久的心思:Oh,my god!我可以毕业了。这时只听见老板一声魔吼“这个课题还得继续研究下去!你要至少发两篇SCI!”
Oh,my god!
谁能告诉我老板肿么了?
课题“看似”已经完结,其实,我们手头上的课题只是实验室大课题的其中一小部分,我们发表的文章也只是解开了其中一小个谜团而已,真正的科学问题可不是一篇SCI文章就能够完整解决的。
生物体内含有多种多样的调控因子,包括转录因子、小RNA、lncRNA、代谢物、DNA甲基化、组蛋白修饰等,这些调控因子在多个组学水平上都有可能对同一个生物学问题产生调控作用;同时,这些调控因子之间又可能会相互作用,也就是说多组学之间是相互补充相互调控的。
拿植物体内磷元素平衡来说。磷是植物生长发育必需的一种重要营养元素,植物缺磷时会产生应激反应,表现出mass减少、叶片枯黄等现象。但在恢复磷元素的供给时,这些现象又可以调整恢复。目前,在各个组学水平上都已经发表了不少研究成果。因此,要想研究磷平衡的维持调控机制,就可以从多个方面入手,得到不一样的结果,相互验证与补充。
下面我们通过两篇分别利用不同组学研究水稻中磷元素平衡的SCI文章,来深入说明这条研究思路。
转录水平
2013年发表在The Plant Cell上的文章是利用RNA-seq从转录水平上来研究的。
对这共126个样本进行RNA-seq测序,差异基因表达分析发现,磷饥饿早期和中期(
3d及之前
),差异表达基因很少,说明机体还没有开始应答;在磷饥饿晚期(
7d后
)机体开始响应,差异表达基因急剧增加(
图1
)。

图1 基因差异表达结果
接着对这些差异基因进行趋势分析,研究在磷饥饿与恢复过程中的基因表达模式。趋势分析的结果把这些差异基因分成了8个模块,其中class1和class4中的基因既响应磷饥饿,也响应磷恢复,所以很可能参与了机体内磷元素平衡的调节。
这些响应基因在磷恢复短时间内就回复到正常表达水平,如class1中的270个上调基因在恢复磷供给后1h就回复到正常表达水平,这些基因包括一些在磷平衡中起着关键作用的基因,如磷转运蛋白PT、SPX、参与脂类代谢的MGD2基因等(
图2
)。
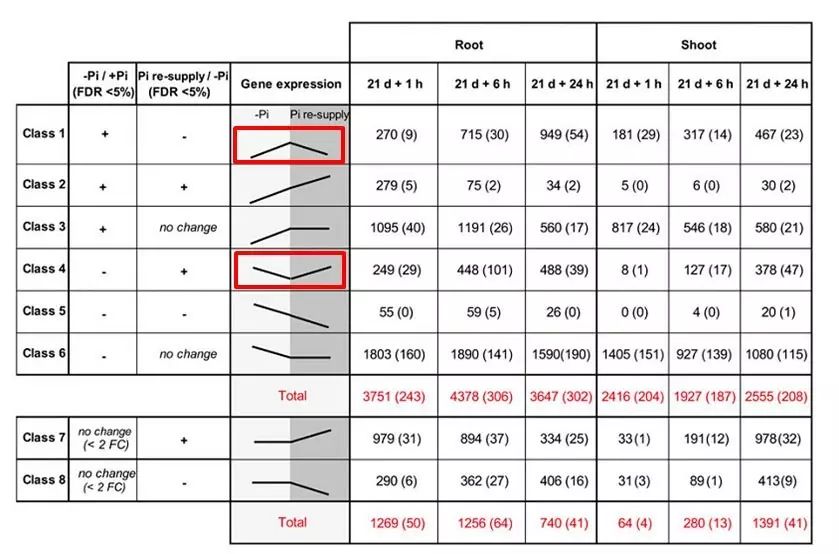
图2根和芽响应磷饥饿的基因表达模式
DNA甲基化水平
2015年发表在eLIFE上的文章是利用全基因组甲基化测序从DNA甲基化水平上来研究的
。

首先对这些样本进行RNA-seq研究转录组层面的变化,结果与上一篇文章的结果类似,也是磷饥饿7d后机体开始响应,差异基因的数目和差异倍数随着磷饥饿时间的延长不断增加;在磷恢复1d后,40%的差异基因表达水平就回复到了正常状态,磷恢复3d后,80%的差异基因表达水平就回复到了正常状态(
图3
)。
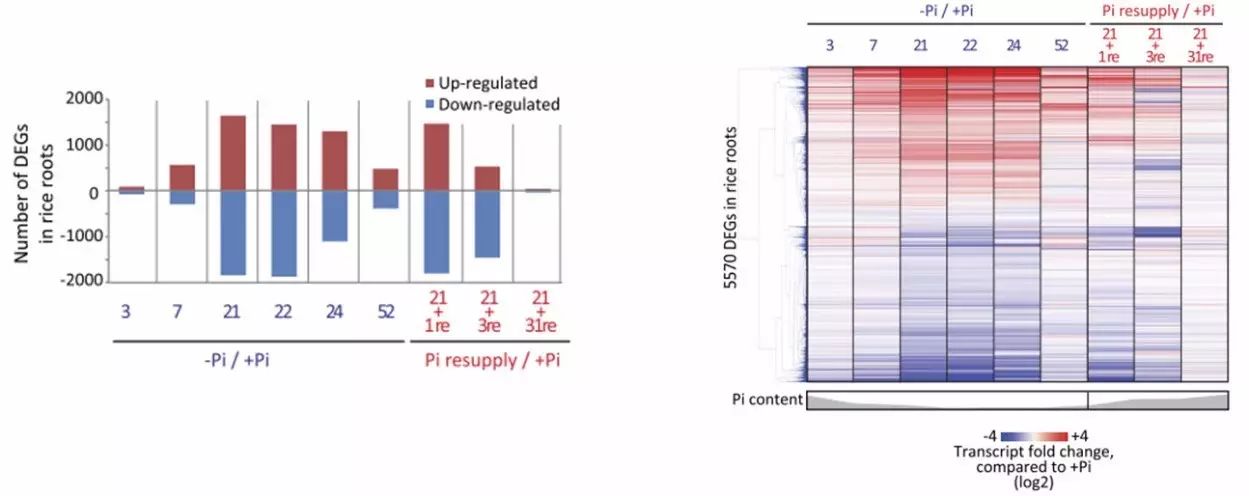
图3 转录组研究结果
然后对样本进行全基因组甲基化测序(
WGBS
),共得到175个差异甲基化区域(
DMR
),其中84%的DMR高甲基化。与转录本水平的变化相似,DNA甲基化水平的变化随着磷饥饿时间的延长而越来越显著。与转录本水平变化在磷恢复3天后就大部分回复正常不同,磷饥饿导致的DNA甲基化水平变化在磷恢复3天后继续维持,在磷恢复31天后才渐渐趋向正常(
图4 A
),这说明营养元素缺乏导致的差异甲基化状态可以维持较长一段时间,即使是恢复到正常的营养状态。
研究这些DMR在基因组中的位置,发现与水稻基因组中90%的转座子区域(
TE
)重叠(
图4 C
)。
对DMR相关基因进行分析,发现95%表达量上调,并且与磷平衡功能相关,如磷酸转运蛋白PT、SPX基因、MGD2、IPS2等(
图4 E
)。
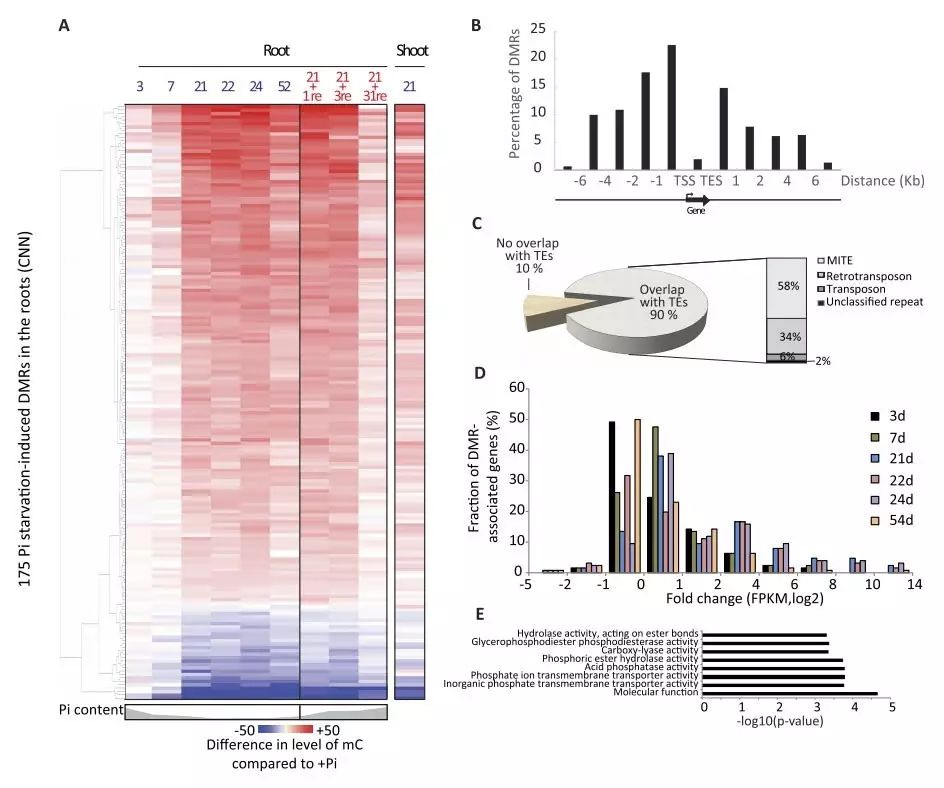
图4 磷饥饿引起的DNA甲基化变化
对这些DMR的甲基化类型进行研究,发现绝大部分都是CHH类型,并且几乎与TE区重叠(
图5

















