前几期我们介绍了扩增子测序分析流程,包括:微生物群落组成分析,α多样性分析,β多样性分析。接下来几期将结合R语言将扩增子测序分析结果可视化,从而更直观地展现群落结构及丰度分布。R是一门用于统计计算和作图的语言,其强大的绘图功能主要由众多的图形函数来实现,包括:高级绘图函数,低级绘图函数,图形参数。本期主要介绍通过柱状图,热图,韦恩图实现对微生物群落组成分析的可视化。
输入文件
:在OTU聚类和物种注释后,通过对物种丰度表排序,均一化等处理后,获得不同分类层级下物种丰度表,例如:tax_table.p.txt

其中,第一行代表样本名,第一列代表门水平下的物种,sample1拟杆菌相对丰度为0.1154。
图形实现
:
#输入文件
data
注:header设置文件第一行为行名,row.names设置文件第一列为列名,sep设置分隔标识符为tab键
#设置颜色
col
#画图
x
注:legend.txt设置图例内容,args.legend设置图例位置和大小,xaxt设置横轴坐标是否显示,col设置颜色,ylab设置纵坐标标题内容,cex.axis设置坐标字体大小
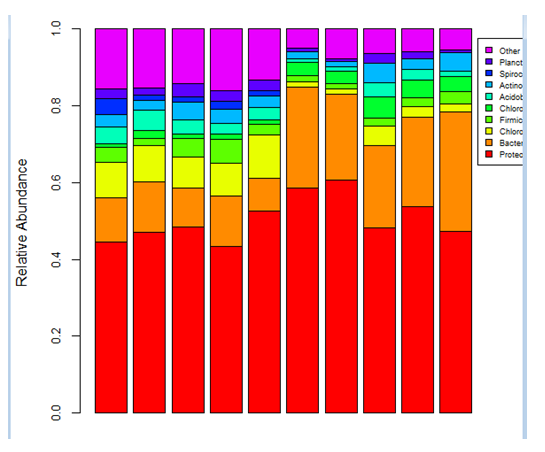
#添加坐标
axis(at=x,labels=colnames(data), cex.axis=0.6,font=2,side=1,las=2)
注:labels设置添加的坐标内容,cex.axis设置坐标字体大小,font设置字体,side设置添加的坐标轴位置,las设置字体方向
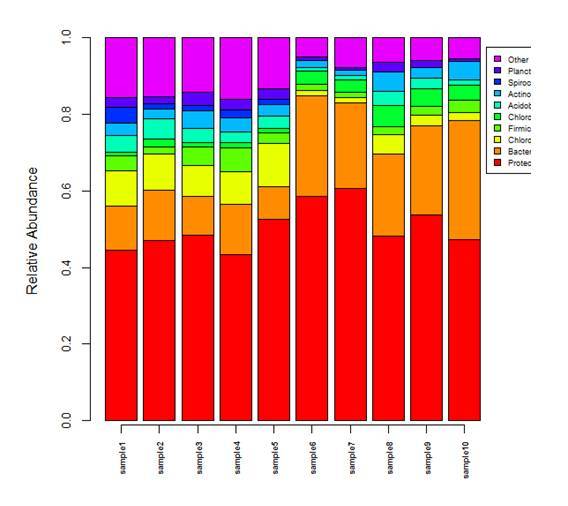
输入文件
:tax_table.g.txt, 物种丰度表(与上同)
group.list,样品分类信息表,格式如下:

图形实现
:
#文件输入
data
group
#设置group行名
colnames(group)
#设置数据框annotation_col(只有一列Group,且行名为样本名)
annotation_col
rownames(annotation_col)

#安装并加载R包pheatmap
install.packages('pheatmap')
library(pheatmap)
#画图
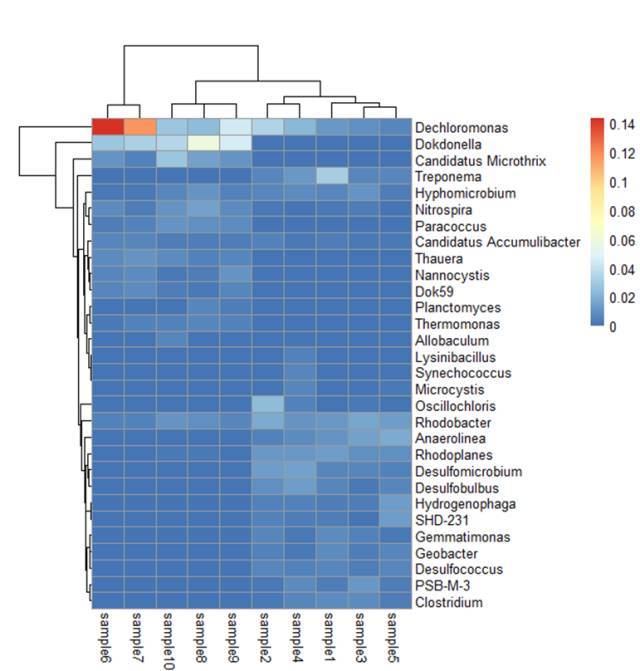
pheatmap(head(data,n=30))
注:提取前30个物种相对丰度值画热图,如图所示图形很丑。为了看一个物种在不同样本中的丰度分布,需要消除样本间差异,即对丰度表进行标准化处理。
pheatmap(head(data,n=30),scale='row')
注:scale设置标准化参数,取值为“row”“column”或“none”
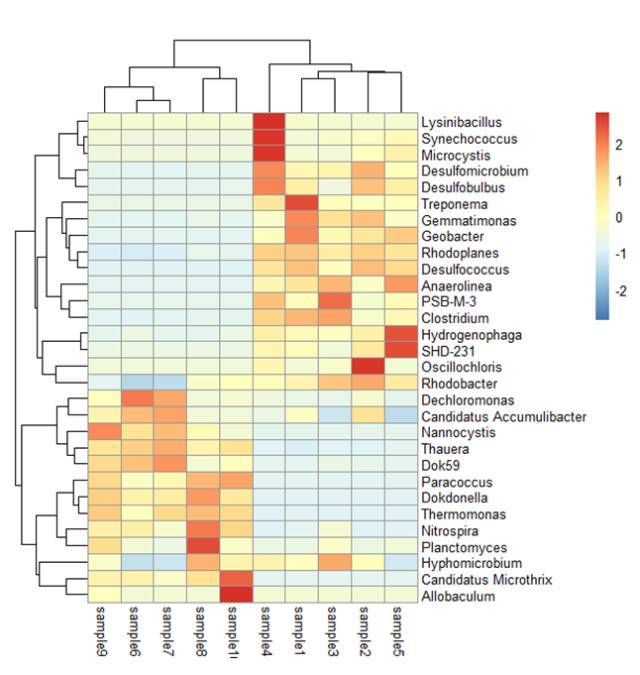
pheatmap(head(data,n=30),scale='row',annotation_col=annotation_col)
注:annotation_col设置样本分类

其他重要参数:
clustering_distance_rows = 'correlation'#设置行聚类使用的聚类方法,也可以自定义方式
breaks#用于调整区间
cluster_row = FALSE#表示行不聚类
legend = FALSE#表示右侧图例不显示
display_numbers = TRUE#表示在热图中格子显示对应的数字
cellwidth, cellheight#表示热图中小方格的宽度和高度
fontsize#表示字体大小
filename #将图形保存成图片文件
main#设置标题















