大家好!我是新加盟生信人团队的小编montreal,和前几位小编遥相呼应,目前在大洋彼岸的美洲大陆最具法兰西风情的Q省Q市Q大学攻读博士学位。相信很多朋友在研究中都遇到过有关进化树作图的问题。从本期开始,我将为大家带来
进化树作图
系列专题,希望能对有着不同专业背景的读者在进化树作图(请注意不是建树)方面有所帮助。
想必大家都知道Newick格式是目前进化树做常用到的一种格式了。比如,对于下面这棵树:
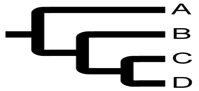
小编作图,欢迎转载或修改
用Newick格式来表示,就是:(A,(B,(C,D)))。很简洁吧?目前,作为最常使用的进化树格式,Newick在各种作图软件中得到了广泛应用。本文将首先回溯历史,带你了解Newick的前世今生。
时光回转三十年。1986年6月26日,美国新罕布什尔州(New Hampshire)多佛市(Dover)的一家龙虾馆里,七位科学家一边吃着龙虾,一边进行着轻松的学术讨论。对于这次会议,能找到的资料极为有限,但其结果却对整个系统发育学(phylogenetics)产生了重要的影响。因为正是在这次会议中诞生了一种全新的、用字符与括号来对进化树进行描述的格式。这次在新罕布什尔的会议实际上由一系列非正式的讨论构成,而最后一次讨论(还有第二次讨论)正是在一家名为Newick’s Lobster House的龙虾馆举行,也许是龙虾鲜美令人难以忘怀的缘故,大家决定用Newick龙虾馆来命名这种格式(学界也称之为New Hampshire format)[1]。

Adapted from Google Map. Terms of use:
https://www.google.com/intl/en_hk/help/terms_maps.html
BTW;这家建于1948年的龙虾馆至今仍在营业。听去过的人说味道确实不错,推荐fried oysters和Gulf shrimp,大家有兴趣可以去尝下鲜,地址:Address: 431 Dover Point Road, Dover, NH 03820。
从此,Newick从新罕布什尔的龙虾馆慢慢走入世界各大学的图书馆,尽管从未在正式文献中被专门论述过
[1]
。七位与会嘉宾都是当时分子进化和计算机学界的翘楚,让我们看看他们都是谁:James Archie, William Day, Joe Felsenstein,Wayne Maddison, Christopher Meacham, F. James Rohlf, David Swofford
[1]
。没听说过?没关系,其中的几位在下文和本系列专题后续的文章中还会多次出现。多说一句,本次会议的发起人
[1]
、著名分子进化学家,时任华大西雅图(University of Washington, Seattle)遗传系教授的Joseph Felsenstein还有一位亲弟弟,Lee Felsenstein,是一位知名的电子工程师,对于PC的早期发展有巨大贡献
[2]
,大家不要搞混。
针对Newick格式,需要注意的是不同的树会有多种的Newick格式对应 [1]。让我再回到本文开始的例子:
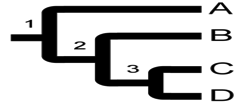
小编作图,欢迎转载或修改
以下三种方式:(A,(B,(C,D))),(A,(B,(D,C))),(A,((C,D),B)),都可以表示这棵树。此外,实际应用中很多的树是无法定根的(rooted)。这里所谓的“根”就是祖先,不能定根也就是我们无法知晓祖先在哪(如图所示,1、2还是3)。用Newick来表示的时候,一般的原则是人为地定一个根。还是上面这棵树,如果对于一棵不定根的树(unrooted tree),那么(A,(B,(C,D))),((C,D),(B,A))和(D,(C,(B,A)))表示的都是同一棵不定根树(unrooted tree)[1]。
Newick的独角戏唱了一年,就迎来了竞争对手Nexus,一种包含有多种信息并以模块化存储的新格式。与Newick不同,Nexus有一篇在正式期刊发表的文章,专门介绍这种格式。这篇文章于1997年刊载在分子进化和系统分类学(systematics)顶级期刊
Systematic Biology
上,尽管此前Nexus已在多种软件中(比如MacClade 3和PAUP 3)被使用[3] 。

作者的名字眼熟吗?对,其中的
David Swofford
和
Wayne Maddison
,正是当年新罕布什尔
“
龙虾七子
”
中的两位。不过为什么排在第一位的也叫
Maddison
?莫非?
你没有猜错,这两个Maddison是亲兄弟,而且还是双胞胎!至于哪个是哥哥,哪个是弟弟,有了解的读者欢迎在后面留言,反正我是分不出,请见下图:

Copyright: Wayne Maddison Lab, CC 3.0
BTW:Maddison兄弟之前都在亚利桑那大学(University of Arizona)做老师,其间经常被人搞混,甚至自己的学生也无法分辨。时常有人向哥哥咨询一件事,几天后却向弟弟询问结果,搞得对方一头雾水。后来也许是大家实在受不了,Wayne Maddison遂改换门庭,去往加拿大的英属哥伦比亚大学(University of British Columbia)任教。
作者在文中明确指出了发明
Nexus
格式旨在提供一种具有延展性(
expandability
),包容性(
inclusivity
),可移植性(
portability
),以及可操作性(
processibility
)的格式。而这些特性的实现,最重要的就是
Nexus
模块(
modularity
)的设计。让我们看一个示例:
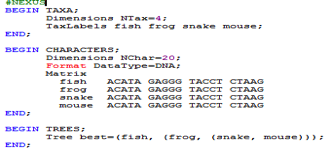 小编作图,欢迎转载或修改
小编作图,欢迎转载或修改
这里面的内容确实不少。不过请注意,
Nexus对于树的描述(或者说tree block),实际上和Newick如出一辙
,所以严格地说,在描述进化树方面,它没有任何创新,只是照搬Newick格式。
有趣的是,上面提及的这几位在进化树格式发展史中具有里程碑意义的人物——Newick龙虾馆会议的组织者Joseph Felsenstein,Nexus格式的发明者David Swofford和Maddison兄弟——分别摘取了第二、第一和第五第六届系统分类学家(systematic biologist)主席终身成就奖(Presidents' Award for Lifetime Achievement)[4]。该奖由系统分类学家协会(Society for Systematic Biologists)每隔三年或以上颁发一次,迄今只有六位得主,堪称系统分类学界最高荣誉[4]。当然,这绝不仅是对于Newick和Nexus的嘉奖。几位科学家的贡献还有很多,比如三个著名的进化软件:Phylip、PAUP和Mesquite,也正分别出自他们之手。
Nexus虽然与Newick同出一源,却由于其更加丰富的内容和复杂的模块化结构,使得很多软件中两者不能兼容:前者有PAUP,Mesquite和MrBayes这样的拥趸,而后者也得到了Phylip,MEGA以及RAxML等软件的青睐。时至今日,如果把两种格式的名字同时输入谷歌搜索框,得到的几乎都是“How to convert nexus files to Newick format?”之类的提问。
Newick和Nexus的二人转在2009年终于迎来了搅局者,它就是phyloXML,一种基于XML并整合了树和其他相关数据的新格式。在phyloXML里,不同的属性用
…
的方式描述,而XML良好的扩展性也使得phyloXML有着更灵活的设计方式[5]。可以说,这种格式是同Newick有着鲜明区别的、也是真正意义上的一种新的进化树格式。该文作者,美国桑福德-伯纳姆医学研究所(Sanford-Burnham Medical Research Institute)的剥尸猴Christian Zmasek和来自印第安纳大学布鲁明顿分校(Indiana University, Bloomington)的Mira Han,在文中强调,生物学的飞速发展和研究的日益深入使得人们有必要将包括物种、基因名、枝长(branch length)、甚至是成种事件(speciation)等广泛信息整合到进化树中,并以一种全新的格式呈现出来。于是二人一道开发了phyloXML。下图(左)展示了一个包含有多种信息的phyloXML文件。下图(右)则是基于phyloXML的一棵包含物种信息和蛋白质结构域信息的树。

小编作图,欢迎随意转载或修改
phyloXML近年来不断完善,现已被包括iTOL在内的许多作图工具所采用,并得到了BioPerl和BioPython的支持,有着越来越广泛的使用群体,与Newick和Nexus渐成三足鼎立之势。有兴趣的读者可以前往phyloXML的大本营
http://www.phyloxml.org
———看看关于phyloXML更多的内容。
除了上述三种进化树格式之外,还有其他的一些“小众”群体。由于篇幅所限不再一一介绍。本期内容至此介绍完毕,有问题的朋友欢迎在下方留言。预知后续内容,请锁定生信人公众号。
参考资料
http://evolution.genetics.washington.edu/phylip/newicktree.html
https://en.wikipedia.org/wiki/Lee_Felsenstein
Maddison, D. R., Swofford, D. L. and Maddison, W. P. (1997), Nexus: An extensible file format for systematic information.
Syst. Biol.
, 46:590–621.
http://www.systbio.org/presidents-award.html
Han M.V. and Zmasek C.M. (2009) phyloXML: XML for evolutionary biology and comparative genomics.
BMC Bioinformatics
, 10:356.
点击以下「
关键词
」,查看往期内容:
一个物种一个家
TCGA
|
小工具
|
数据库
|
组装
|
注释
|
基因家族
|

















