热图美化
上一期的绘图命令中,最后一行的操作抹去了之前设定的横轴标记的旋转,最后出来的图比较难看。
上次我们是这么写的
p "samples") + theme_bw() + theme(panel.grid.major = element_blank()) + theme(legend.key=element_blank())
为了使横轴旋转45度,需要把这句话
theme(axis.text.x=element_text(angle=45,hjust=1, vjust=1))
放在
theme_bw()
的后面。
p 45,hjust=1, vjust=1))
最后的图应该是下边样子的。
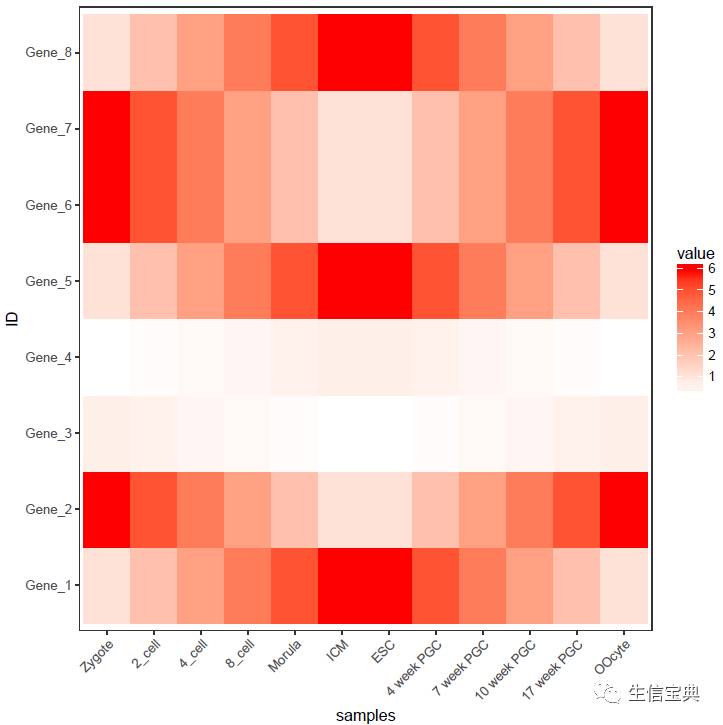
上图的测试数据,数值的分布比较均一,相差不是太大,但是Gene_4和Gene_5由于整体的值低于其它的基因,从颜色上看,不仔细看,看不出差别。
实际应用中,异常值的出现会毁掉一张热图,如下是一个例子。
data 5,mean=5), rnorm(5,mean=20), rnorm(5, mean=100), c(600,700,800,900,10000))
data 5, byrow=T)
data rownames(data) 1:4]
colnames(data) "Grp", 1:5, sep="_")
data
Grp_1 Grp_2 Grp_3 Grp_4 Grp_5
a 5.958073 5.843652 3.225465 4.886184 3.411362
b 19.630582 20.376791 20.744580 18.534027 20.638288
c 100.351299 99.849900 102.197343 98.583629 99.540488
d 600.000000 700.000000 800.000000 900.000000 10000.000000
data$ID data
Grp_1 Grp_2 Grp_3 Grp_4 Grp_5 ID
a 5.958073 5.843652 3.225465 4.886184 3.411362 a
b 19.630582 20.376791 20.744580 18.534027 20.638288 b
c 100.351299 99.849900 102.197343 98.583629 99.540488 c
d 600.000000 700.000000 800.000000 900.000000 10000.000000 d
data_m "ID"))
head(data_m)
ID variable value
1 a Grp_1 5.958073
2 b Grp_1 19.630582
3 c Grp_1 100.351299
4 d Grp_1 600.000000
5 a Grp_2 5.843652
6 b Grp_2 20.376791
p "samples") + theme_bw() + theme(panel.grid.major = element_blank()) + theme(legend.key=element_blank()) + theme(axis.text.x=element_text(angle=45,hjust=1, vjust=1)) + theme(legend.position="top") + geom_tile(aes(fill=value)) + scale_fill_gradient(low = "white", high = "red")
p
dev.off()
输出的结果是这个样子的
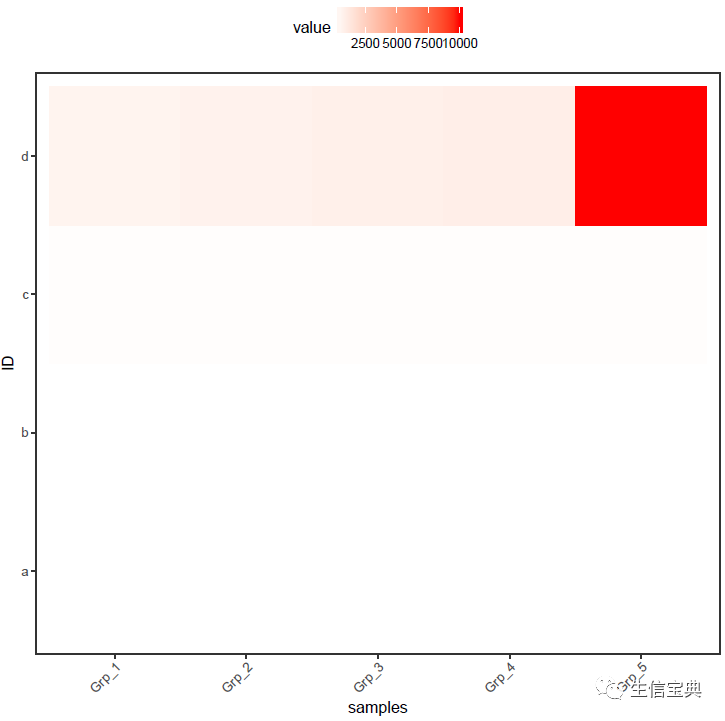
图中只有右上角可以看到红色,其他地方就没了颜色的差异。这通常不是我们想要的。为了更好的可视化效果,需要对数据做些预处理,主要有
对数转换
,
Z-score转换
,
抹去异常值
,
非线性颜色
等方式。
对数转换
为了方便描述,假设下面的数据是基因表达数据,4个基因 (a, b, c, d)和5个样品 (Grp_1, Grp_2, Grp_3, Grp_4),矩阵中的值代表基因表达FPKM值。
data 5,mean=5), rnorm(5,mean=20), rnorm(5, mean=100), c(600,700,800,900,10000))
data 5, byrow=T)
data rownames(data) 1:4]
colnames(data) "Grp", 1:5, sep="_")
data
Grp_1 Grp_2 Grp_3 Grp_4 Grp_5
a 6.61047 20.946720 100.133106 600.000000 5.267921
b 20.80792 99.865962 700.000000 3.737228 19.289715
c 100.06930 800.000000 6.252753 21.464081 98.607518
d 900.00000 3.362886 20.334078 101.117728 10000.000000
# 对数转换
# +1是为了防止对0取对数;是加1还是加个更小的值取决于数据的分布。
# 加的值一般认为是检测的低阈值,低于这个值的数字之间的差异可以忽略。
data_log 1)
data_log
Grp_1 Grp_2 Grp_3 Grp_4 Grp_5
a 2.927986 4.455933 6.660112 9.231221 2.647987
b 4.446780 6.656296 9.453271 2.244043 4.342677
c 6.659201 9.645658 2.858529 4.489548 6.638183
d 9.815383 2.125283 4.415088 6.674090 13.287857
data_log$ID = rownames(data_log)
data_log_m = melt(data_log, id.vars=c("ID"))
p "samples") + ylab(NULL) + theme_bw() + theme(panel.grid.major = element_blank()) + theme(legend.key=element_blank()) + theme(axis.text.x=element_text(angle=45,hjust=1, vjust=1)) + theme(legend.position="top") + geom_tile(aes(fill=value)) + scale_fill_gradient(low = "white", high = "red")
ggsave(p, filename="heatmap_log.pdf", width=8, height=12, units=c("cm"),colormodel="srgb")
对数转换后的数据,看起来就清晰的多了。而且对数转换后,数据还保留着之前的变化趋势,不只是基因在不同样品之间的表达可比 (同一行的不同列),不同基因在同一样品的值也可比 (同一列的不同行) (不同基因之间比较表达值存在理论上的问题,即便是按照长度标准化之后的FPKM也不代表基因之间是完全可比的)。
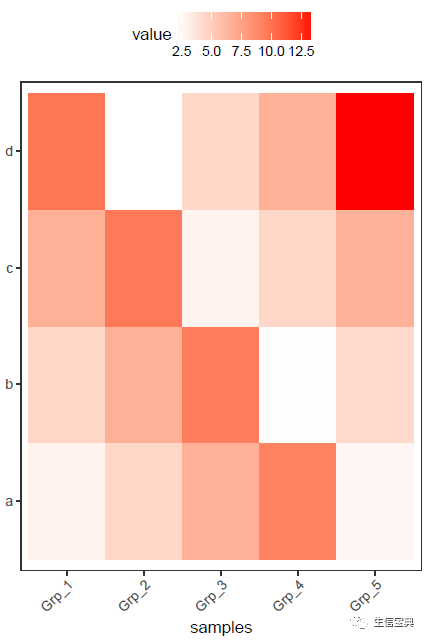
Z-score转换
Z-score
又称为标准分数,是一组数中的每个数减去这一组数的平均值再除以这一组数的标准差,代表的是原始分数距离原始平均值的距离,以标准差为单位。可以对不同分布的各原始分数进行比较,用来反映数据的相对变化趋势,而非绝对变化量。
data_ori "Grp_1;Grp_2;Grp_3;Grp_4;Grp_5
a;6.6;20.9;100.1;600.0;5.2
b;20.8;99.8;700.0;3.7;19.2
c;100.0;800.0;6.2;21.4;98.6
d;900;3.3;20.3;101.1;10000"
data T, row.names=1, sep=";", quote="")
data 1,var)!=0,]
data
Grp_1 Grp_2 Grp_3 Grp_4 Grp_5
a 6.6 20.9 100.1 600.0 5.2
b 20.8 99.8 700.0 3.7 19.2
c 100.0 800.0 6.2 21.4 98.6
d 900.0 3.3 20.3 101.1 10000.0
data_scale 1,scale)))
colnames(data_scale) data_scale
Grp_1 Grp_2 Grp_3 Grp_4 Grp_5
a -0.5456953 -0.4899405 -0.1811446 1.7679341 -0.5511538
b -0.4940465 -0.2301542 1.7747592 -0.5511674 -0.4993911
c -0.3139042 1.7740182 -0.5936858 -0.5483481 -0.3180801
d -0.2983707 -0.5033986 -0.4995116 -0.4810369 1.7823177
data_scale$ID = rownames(data_scale)
data_scale_m = melt(data_scale, id.vars=c("ID"















