正文
本文是完整翻译版,生信宝典公众号同一期还有导读版
文献阅读与翻译 - Gaining comprehensive biological insight into the transcriptome by performing a broad-spectrum RNA-seq analysis (Nature Communications DOI: 10.1038/s41467-017-00050-4)
摘要
RNA-sequencing
(
RNA-seq
)是转录组研究的重要技术。自从
RNA-seq
技术问世以来,已经开发了大量的分析工具。虽然有有一些研究尝试评估最新的分析工具,但是还没有综合评价分析流程评估单个或组合分析工具的优劣。在这篇文章中,研究人员深入探索了大量的
RNA-seq
分析流程。这些流程不仅包括表达分析技术,也包含了RNA variant-calling、RNA编辑和RNA融合检测技术。具体来说,研究人员检查了短读长和长读长的
RNA-seq
技术,包括了39个分析工具的120种组合,15个样品的490种分析,样品包括生殖细胞、癌细胞和干细胞数据集,最后报道了分析工具的性能,提出了一个综合
RNA-seq
分析手册:
RNACocktail
。这个手册包含获得高准确性的分析流程。通过与实时定量
PCR
数据的比较验证,表明作者提出的流程可以帮助研究着获得更多准确的生物相关的研究信息。
简介
高通量二代测序(
NGS
)
RNA-seq
将转录组分析引入一个新的时代。
RNA-seq
需要各种分析流程以满足测序技术、样品类型、基因组获取以及计算机资源的需求。
不同的分析流程具有显著不同的分析准确度、速度和代价
。因此,在受到代价和性能限制的条件下研究
RNA-seq
分析每一步使用哪个或哪些分析工具获得最高的准确性显得尤为重要。通常,整体最佳的流程用于特定样品分析可能是次优的,找出最优的分析流程更具挑战性,这需要分析大量不同的数据集。
有很多研究比较了不同
RNA-seq
分析工具的性能。但是,这些研究主要关注
RNA-seq
分析的某一步,或者局限于比对和定量。因此,综合系统分析有助于最大化了解
RNA-seq
数据。为了解决以前研究的限制,研究人员深入调查了
RNA-seq
分析的所有主要步骤,评价了不同步骤下分析工具组合的准确性、效率和一致性,提出了一个综合的
RNA-seq
分析流程手册。他们认为
RNACocktail
分析流程可以获得高准确性。
RNACocktail
分析流程在检测不同样品生物相关的差异表达基因和临床重要的转录本得到了进一步的验证。
RNACocktail
分析流程是开源的并且可以在网站http://bioinform.github.io/rnacocktail/免费下载使用。
结果
数据集
为了进行综合评估,研究人员分析了不同种类的
RNA-seq
数据, 包括15个
Illumina
和
Pacific Biosciences
(
PacBio
)数据集。这些数据具体来自正常人类样品NA12878、人类MCF-7乳腺癌细胞、H1人类胚胎干细胞(hESC)以及测序质量控制联盟(
SEQC
)。
RNA-seq分析流程手册
下图展示了研究人员提出的综合分析
RNA-seq
数据的流程手册。在流程中的每一步都列出了常用分析方法 (使用的工具名和版本号请看补充表2)。下面会详细阐述每一个步骤。

RNACocktail
分析流程手册。
RNACocktail
是一个综合的
RNA-seq
数据分析手册。本图概括了
RNA-seq
分析关键步骤中广泛使用的分析工具。
研究人员可以使用这个图展示的工作流程分析
RNA-seq
数据
。
通过短读长进行转录本检测
识别表达的转录本往往是RNA-seq分析的第一步。通常先将
reads
比对回参考基因组(或者参考转录组),然后根据
reads
比对结果拼装转录本。比对到参考基因组可以检测新的转录本,但需要消耗大量计算资源。而比对回参考转录组要容易得多,但检测不到新的转录本。另外如果没有可靠的参考基因组或者转录组存在,也可以从头组装转录本。
基于参考的转录本识别
比对和结合点预测
RNA-seq
reads
拼接比对到参考基因组要根据外显子-内含子边界拼接
reads
。下面研究人员使用短读长的
Illumina
hESC、NA12878、SEQC、100 bp和300 bp长的MCF7数据集评价了
TopHat
、
STAR
和
HISAT2
的性能,如图所示。
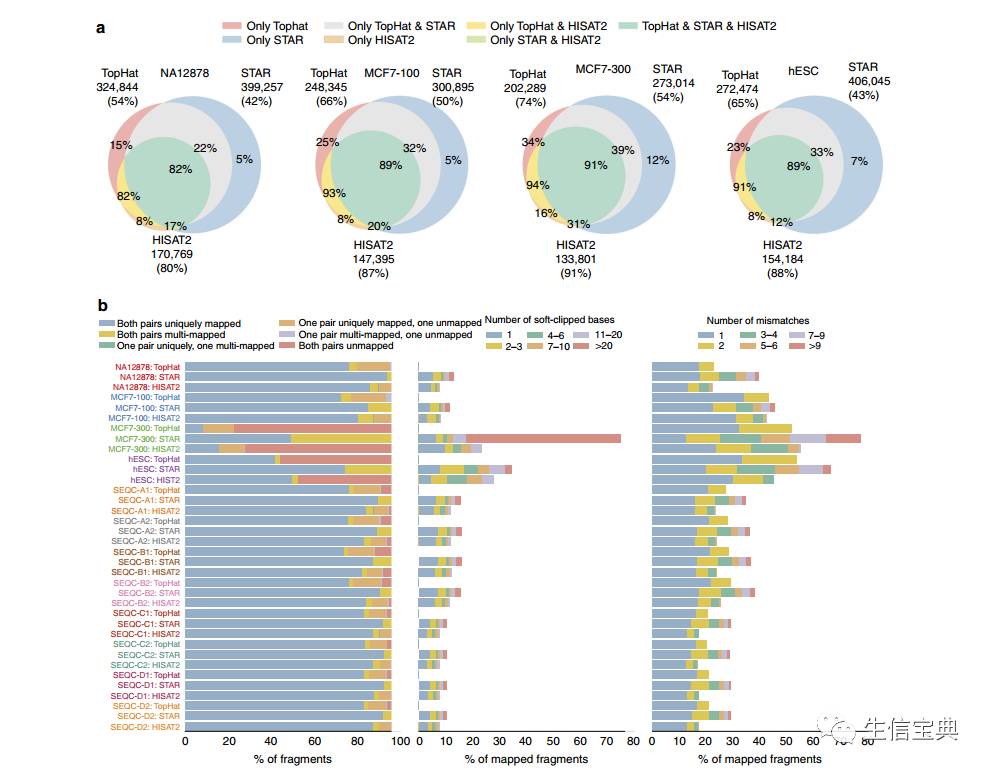
不同比对方案的性能比较。a.不同方案检测到的拼接位点之间的overlap以及与dbEST数据库可靠的拼接点相比的验证率。可靠的EST拼接点至少包括两个EST支持的拼接点。圆的大小反映了每个方案检测到的拼接点数量。图中展示了每个工具检测到的拼接点数量和验证率(位于小括号中)。b.read比对分析。左图展示测序片段比对状态的分布(展示了NA12878、MCF7和SEQC样品的双端reads比对状态,对于hESC样品,展示了单端reads的比对状态,蓝色表示独一无二比对到基因组,橙色表示基因组上有多个比对位置,红色表示没有比对到基因组上)。中图展示了比对回基因组的片段上碱基被软件去除 (
soft-clip
)的数量分布。右图展示了比对回基因组的片段错配碱基的数量分布。
R语言学习 - 韦恩图
轻松绘制各种Venn图
HISAT2
在所有样品中拥有最高的拼接点验证率,但是其预测的拼接点数量小于
TopHat
和
STAR
,如图a所示。
STAR
具有最高比例的独一无二比对到基因组上的reads,尤其是300读长的MCF7样品(b)。与
TopHat
和
HISAT2
不同,
STAR
会把双端reads比对到基因组,否则移除双端reads,以避免单端reads的比对。另一方面,STAR获得了较低质量的比对,具有更多的
soft-clipped
比对和错配碱基(b)。
TopHat
禁止截断reads(
b
)。对长读长样品MCF7-300和单端read样品hESC的分析结果表明,与TopHat和
HISAT2
相比,
STAR
具有更高的容忍性,接受碱基错配和
soft-clipping
以将更多的reads比对回参考基因组。在比对速度方面,
HISAT2
比
STAR
快2.5倍,比
TopHat
快大约100倍。
基于比对的转录组组装
拼接比对之后,表达的转录本集合通过转录组组装来识别。下面研究人员关注两个广泛使用的基于比对的转录组发掘工具
Cufflinks
和
StringTie
。作为这两个组装工具的输入,我们使用了上述讨论的三个比对工具。我们以
Ensembl
参考转录组注释为指导。
除了短读长转录本预测工具外,也研究了转录本检测和预测工具(
IDP
)。
IDP
通过短读长比对以帮助长读长的转录本检测,是一个混合的转录本预测工具。
IDP
通过
GMAP
和
STARlong
的长读段比对和
TopHat
、
STAR
和
HISAT2
的短读段比对进行评价。此外,也分析了长读段转录本预测工具
Iso-Seq
。
研究人员将预测的转录本与
GENCODE
v
19中的参考转录组注释比较,不存在于参考转录组中的转录本认为是假阳性(
FPs
)。
Cufflinks
和
StringTie
预测了许多单个外显子转录本,大部分是假阳性(图
a
)。
StringTie
预测的转录本数量比
Cufflinks
多50–200%。
IDP
在不同的样品中预测最少的外显子,它预测不出单外显子的基因。对于多个外显子转录本来说,
IDP
预测的转录本数量和
Cufflinks
相似(图
a
),而且,其预测的外显子数量分布与
GENCODE
更相似。
Iso-Seq
算法预测的94%的单个外显子转录本和77%的多个外显子转录本在
GENCODE
上是没有的,这反映了其组装准确度不高,但是具有更高的敏感度检测新转录本。
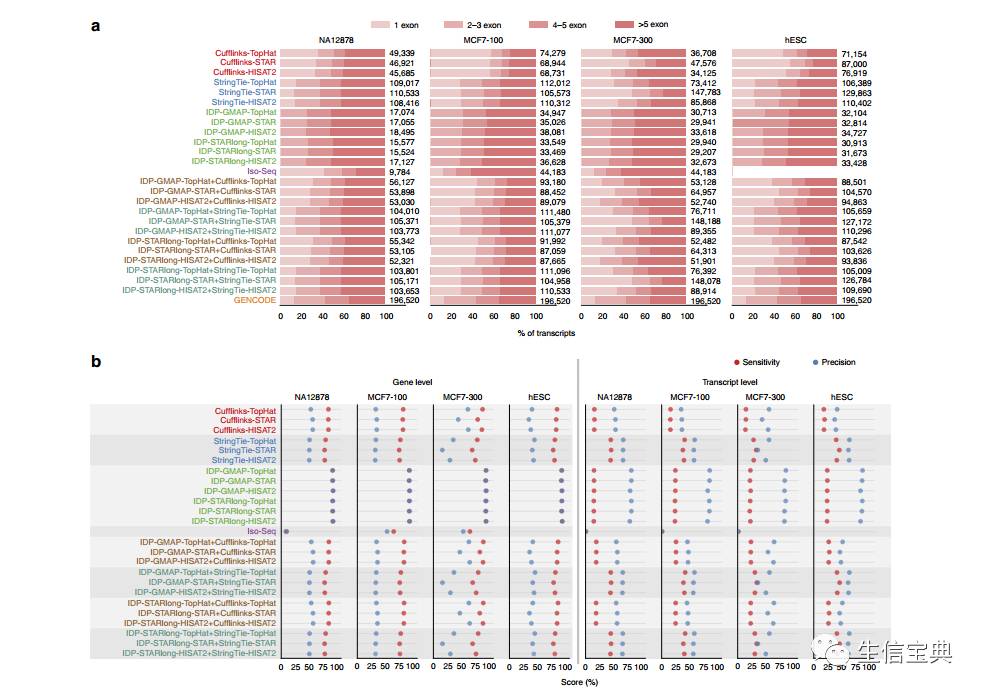 不同转录组重构方案的性能比较。a.不同的组装算法预测的转录本外显子数量分布。标签标明了组装工具、长读段比对工具(
不同转录组重构方案的性能比较。a.不同的组装算法预测的转录本外显子数量分布。标签标明了组装工具、长读段比对工具(
IDP
)、短读段比对工具,之间用”-“分开。b.基因和转录本水平上不同转录组重构方法的敏感度和准确度。
GENCODE
参考转录组注释用作金标准。使用短读长和长读长转录本预测的组合方法(用”+”标记)稍微提升了短读长转录本预测方案的性能。
R语言学习 - 散点图绘制
对于MCF7-300样品,有
STAR
工具的组合预测出更多的转录本(图a)。
IDP
与长读段比对工具
GMAP
和短读段比对工具
HISAT2
联合使用会预测出更多转录本(图a)。
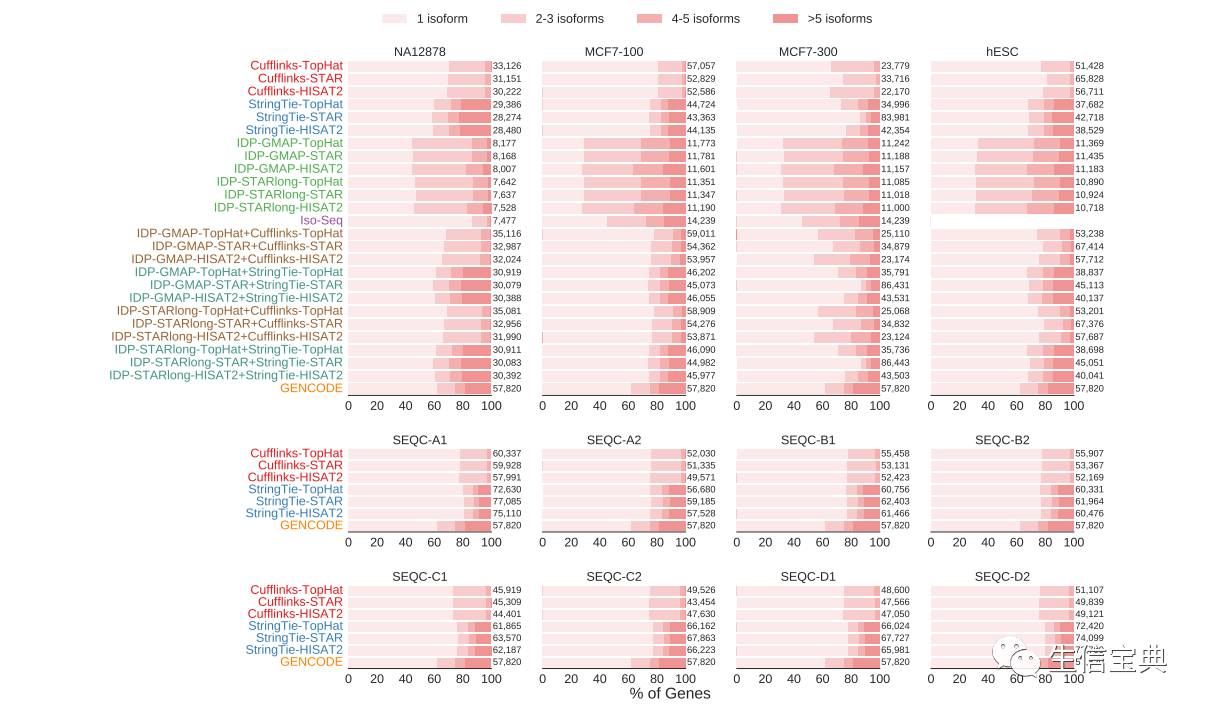
与短读段组装工具不同,
IDP
会检测到一个基因的多个转录本(图)。与
Cufflinks
相比,
StringTie
预测更多的基因,而且每个基因超过5个转录本(图)。
StringTie
的预测结果最好地匹配了
GENCODE
上每个基因转录本数量的分布。
不同转录组重构算法预测的每个基因转录本数量归一化后的分布比较。标签标明了组装工具、长读段比对工具(
IDP
)、短读段比对工具,之间用”-“分开。使用短读长和长读长转录本预测的组合方法(用”+”标记)稍微提升了短读长转录本预测方案的性能。
IDP
在基因水平评价中获得了最好的准确度和灵敏度(图b)。
Cufflinks
比
StringTie
更敏感和准确。对于
MCF
7-300样品,组合工具之间性能差异较大。与
StringTie
组合使用的
TopHat
和
HISAT2
表现比
STAR
更佳。
Iso-Seq
敏感度最低,准确度在
IDP
和
Cufflinks
、
StringTie
之间。
在转录本水平上,
IDP
在准确度方面表现最佳(图b)。
IDP
的预测局限于多个外显子的转录本,它的灵敏度稍高于
Cufflinks
,低于
StringTie
。在短读长组装工具中,在转录本水平,
StringTie
的灵敏度和准确度都高于
Cufflinks
,分别高出25%和11%(图b)。
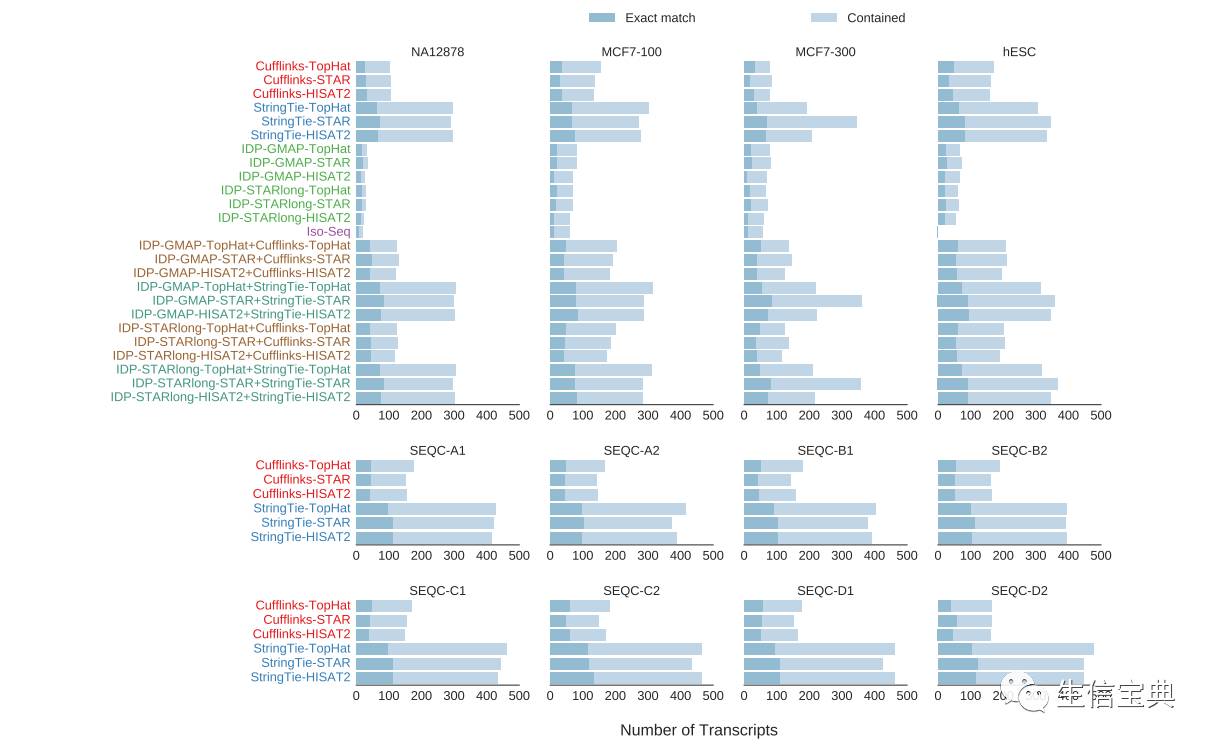
不同的工具预测
Ensembl
没有注释但
GENCODE
v19注释的3681个新的转录本,结果表明
StringTie
预测到了最多的转录本,比
Cufflinks
多2.5倍,比
IDP
多6.5倍(图)
。
不同转录组重构算法预测新的转录本性能比较。新的转录本是
GENCODE
(v19)注释的但在
Ensembl
中没有注释的多外显子转录本。标签标明了组装工具、长读段比对工具(
IDP
)、短读段比对工具,之间用”-“分开。使用短读长和长读长转录本预测的组合方法(用”+”标记)稍微提升了短读长转录本预测方案的性能。
StringTie
是运行最快的工具,完成组装比
Cufflinks
快约60倍,比
IDP
快约50倍
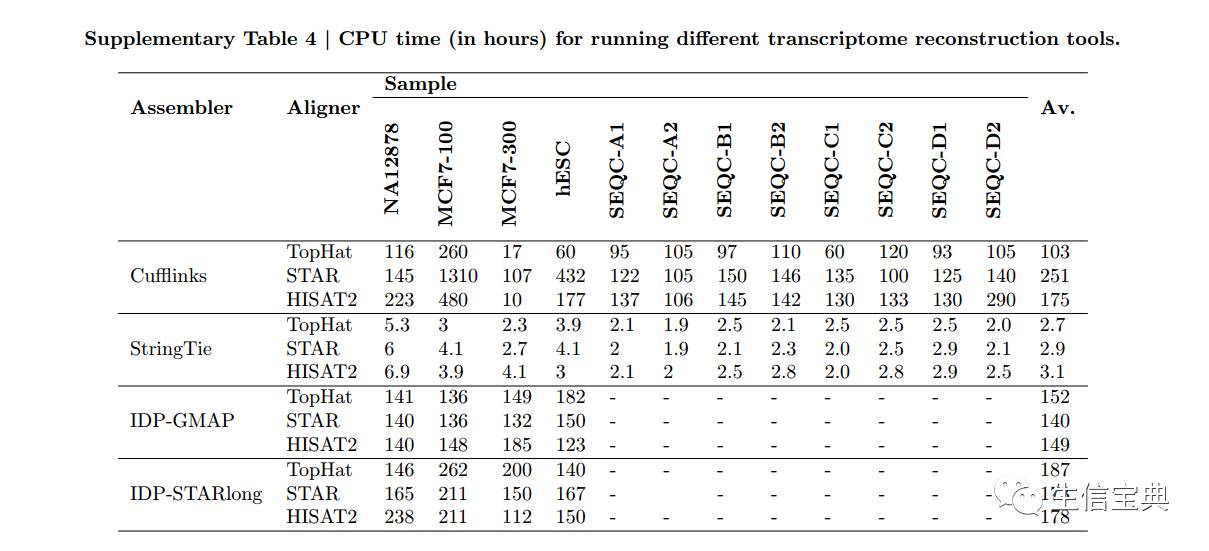
(默认输入是错误校正过的数据和比对后的数据),如下表所示。
转录本定量
基于比对的转录本定量
经典的比对分析是将reads比对回参考基因组或者参考转录组,之后估计转录本丰度。
如果研究目的是测量已知的和新的转录本丰度,比对回参考基因组后使用
Cufflinks
和
StringTie
进行组装和丰度估计。如果使用参考转录组是发现不了新的转录本的,reads可以直接比对到转录组之后使用
RSEM
和
eXpress
进行丰度估计
。
不经过比对的转录本定量
不经过比对的定量方法直接将reads分配给转录本,这与拼接比对方法相比需要更少的计算资源。
Sailfish
、
Salmon
、
quasi-mapping
和
kallisto
四种工具可以解决哪个转录本可以生成哪个read,或者寻找部分比对回转录组的reads。
下面我们比较了基于基因组比对的工具
StringTie
和
Cufflinks
(使用不同的比对工具)、基于转录组的比对工具
eXpress
和
Salmon-Aln
、不经过比对的工具
kallisto
、
Sailfish
(with quasi-mapping)、
Salmon-SMEM
和
Salmon-Quasi
、基于长读段的技术
IDP
(使用不同的短读长和长读长比对工具)的性能。
基于log尺度表达水平之间的Spearman等级相关分析的不同定量方法聚类分析表明采用相似方法的定量工具聚在一起(图a)。不经过比对的工具与StringTie聚的更近。Salmon-SMEM与基于转录组比对的工具
eXpress
和
Salmon-Aln
聚在一起,
Salmon-SMEM
运行速度更快。
基于短读段的技术中,两个不经过比对的工具
kallisto
和
Salmon-SMEM
对MCF7-100和MCF7-300样品具有最一致的预测结果(图b,c)。对于MCF7-100和MCF7-300样品,
IDP
表现出较高的一致性(图b),尤其是删除低表达的基因后一致性更高(图c)。
通常,
StringTie
与高效的比对工具如
HISAT2
组合是最高效的基于比对方法,但是不进行比对的工具也非常有效(运行速度比比对工具快一个数量级)
。之前的研究表明与比对方法相比,定量方法对丰度估计影响更大,我们的研究结果也证实了这一点。
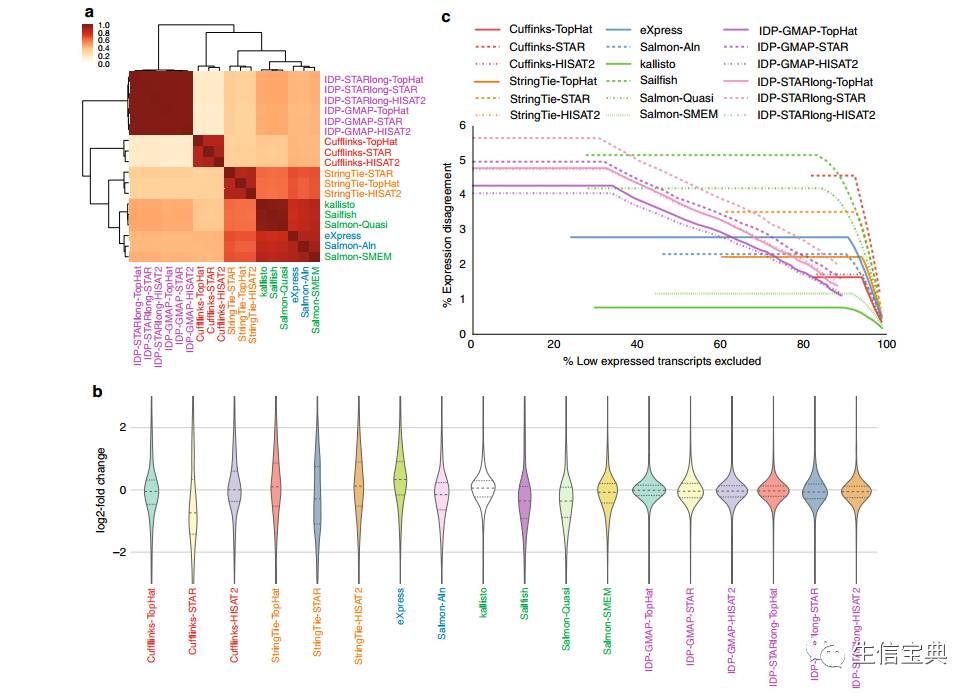
转录本丰度估计工具的性能比较。a.基于NA12878样品log表达的spearman等级相关分析的不同方案的聚类分析比较。b. MCF7-100和MCF7-300样品之间log2-fold表达变化的分布。每种方法中,虚线表示分布的平均值,点线表示四分位数。c.用不同阈值去掉底表达转录本后在 MCF7-100和MCF7-300样品之间表达差异百分比。
R语言学习 - 箱线图一步法
R语言学习 - 箱线图(小提琴图、抖动图、区域散点图)
R语言学习 - 热图简化
R语言学习 - 线图绘制
差异表达
不同样品和条件下差异表达基因的识别是
RNA-seq
分析的重要目标。有多种方法检测差异表达基因,包括基于计数技术的
DESeq
、
limma
和
edgeR
、基于组装技术的
Cuffdif
和
Ballgown
、不经过比对定量进行差异分析的
sleuth
。
首先,通过从
SEQC
样品(SEQC-A vs. SEQC-B)1001个基因中检测差异表达的基因与反转录定量PCR(
qRT-PCR
)测量的表达变化进行比较来评价工具的性能(图)。与其他工具相比,DESeq2表现最佳。
sleuth
、
edgeR
和
limma
性能较差。
Cuffdiff
和
Ballgown
的准确度没有基于计数的工具准确度高。对于AUC-30的测量,
edgeR
表现最佳。
我们比较了不同的工具在预测SEQC数据集中的92个外部RNA控制联盟
spike-in
基因的准确度。对于Spearman等级相关系数和RMSD测量,
DESeq2
获得最佳的性能。对于AUC-30测量,Cufflinks和
Ballgown
表现最佳。基于计数的工具比基于组装的工具更高效。不经过比对技术如
Salmon
和
kallisto
能够获得高质量的预测结果。















