
现在点击解螺旋下方菜单可以直达“直播间”、“学习社区”和“课程网站”,学习更加方便。
作者:子非鱼
转载请注明:解螺旋·临床医生科研成长平台
世人皆知,细胞身娇体贵难养活,养好细胞实属不易。而这其中最烦的是污染、最怕的是掉包。因为尽管研究僧们有方法把一切污染的源头扼杀在摇篮之内,却没有火眼金睛辨别已经换了马甲的错误细胞,因而细胞一旦被李代桃僵,再资深的科研者都会阴沟里翻船。
话说,每每细胞进行华丽丽的“大变身”后,都会让研究僧们回忆起被错误细胞系支配的恐惧之中。现如今,荷兰的学者在进行文献调查之后,于Plos One发表文章指出又有3万余篇的研究论文因细胞系的错认而纷纷阵亡(咦, 为什么要说又呢?);而这些文献还有着相当高的引用率,2016年就有超过4万余篇论文引用过这些文献。
显然,这已不是错认细胞系所引起的第一例冤假错案了。去年6月,RetractionWatch网站的一则消息就声称,某一著名高校就曾因误将Hela细胞(宫颈癌细胞)当做KB细胞(口腔上皮样癌细胞)使用,以至于已发表的关于肿瘤新分子靶标的文章被撤稿。
去年8月底,瑞典乌普萨拉肿瘤学家Bengt Westermark通过DNA-指纹技术将从全球最大的生物材料保藏库(ATCC)获得的目前广泛应用的脑癌细胞系U87(据统计,相关论文已达2000多篇)与50年前建立的原始肿瘤样本进行比较,发现两者并不匹配;同时,目前也没有人能肯定目前使用的细胞系的真正起源。
唯一庆幸的是,尽管U87不是最初的U87,但是它仍然来自胶质母细胞瘤组织,这就意味现在使用的U87细胞系仍可以反应出脑肿瘤的生物学,而相关论文是无需被撤稿的。
现阶段,Hela细胞就经常因更换马甲且混迹在各类细胞系中,而被冠以细胞界“隔壁老王”的称号。然而这还只是冰山一角,文章中还指出有超过451个细胞系均已被其他细胞进行假冒顶替。
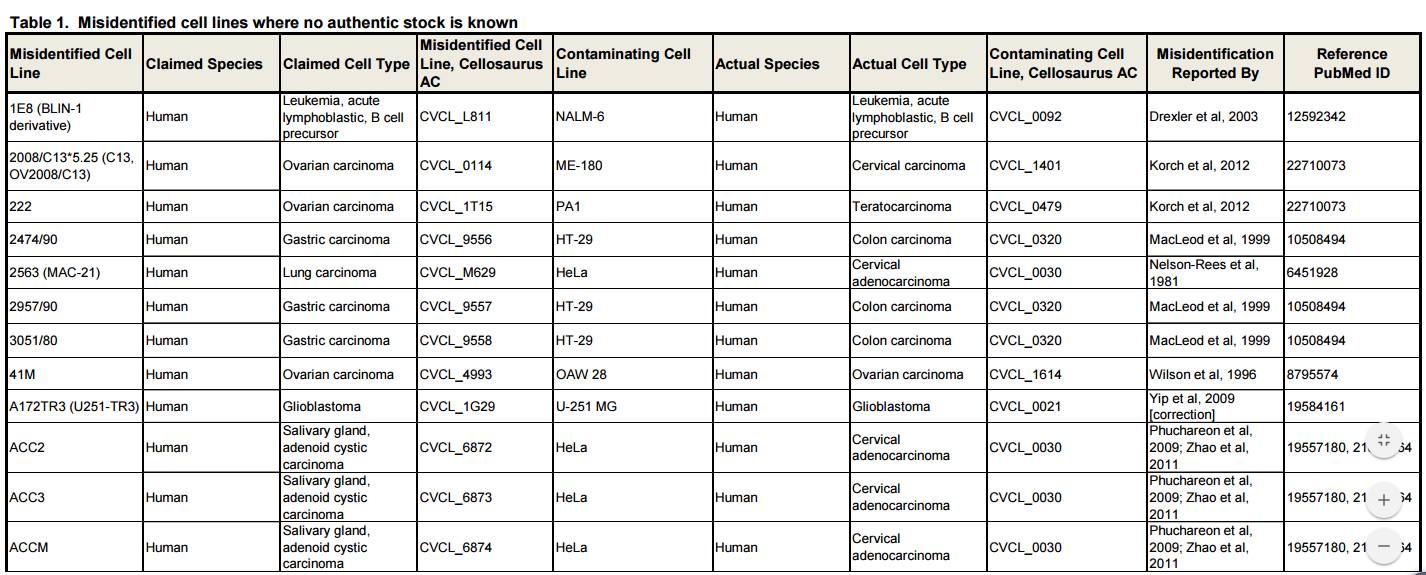
比如前列腺癌细胞系ALVA-31完全就被PC-3细胞系顶包,而最初证实来源于前列腺癌组织的JCA-1细胞其实是来源于T24膀胱癌组织;而于1994年新建的淋巴细胞系(F2-4E5, F2-5B6, P1-1A3 and P1-4D6)则被确定为来源于肝癌组织。下图只截取了表格中的一部分内容,在公众号内回复“细胞系”,可查看表格中更多被错认的细胞系。
其实,人类和动物细胞培养物的错误鉴定是一个长期的问题,而最初意识到此问题却要追溯到上世纪50年代。核型分析和免疫学方法最先被应用于细胞鉴定,当大量的种系错误鉴定被报道后,才促使ATCC在1962年建立细胞系鉴定库。
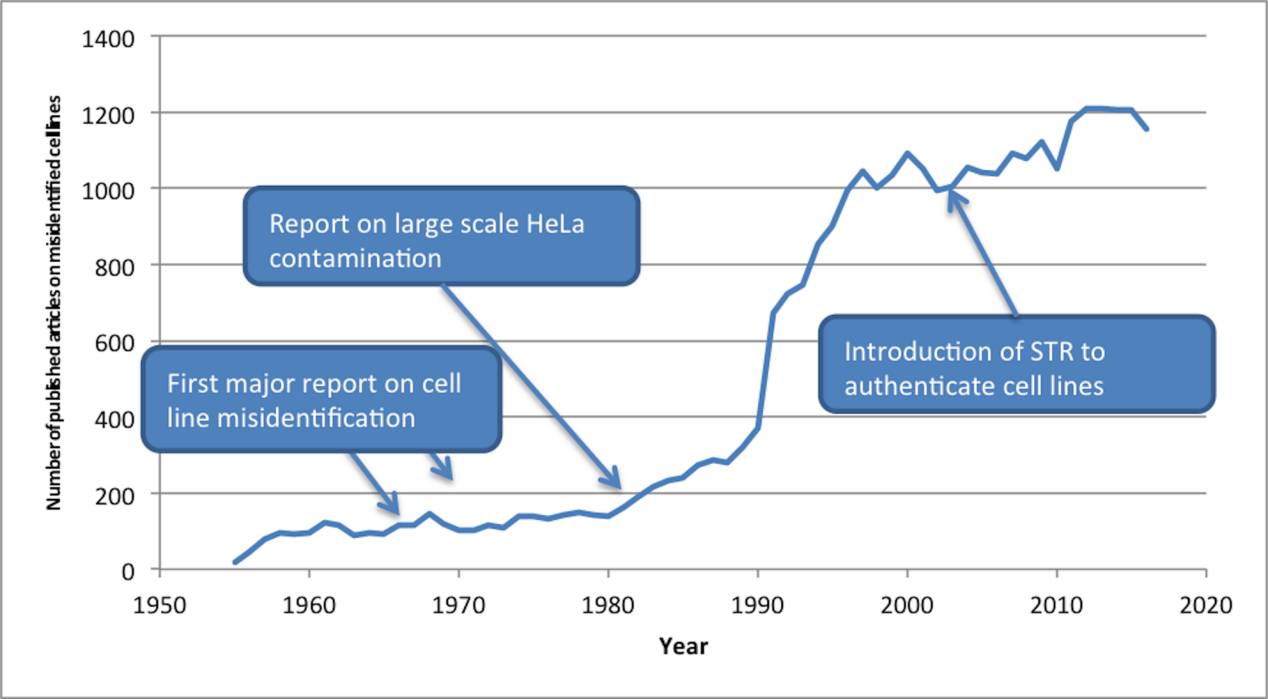
1966年Stanley Gartler通过对同工酶表达水平的鉴定,首次提出来源于18个独立个体的细胞系,如正常肠上皮细胞(Int-407)、正常羊膜细胞(WISH)、正常肝细胞(Chang liver)、喉癌(Hep-2)和口腔癌(KB),可能全是Hela细胞的观点。
而直至1981年,科学家Walter Nelson-Rees在利用核型分析对细胞进行鉴定后,才证实了细胞库中的一系列细胞系确实存在着大量的Hela细胞交叉污染,并将该成果发表于Science期刊上。然而,这份研究报告在当时并不被广大科研者们所认可,甚至还受到了许多同行的攻击,连NIH也终止与他的合同。
此后,细胞错误鉴定现象愈演愈烈,问题逐步扩大。从1969到2004年,错误鉴定培养物的使用在PubMed数据库中增加了大约10倍,而使用培养细胞发表的论文就增加了2-2.5倍。
到2004年,除了Hela细胞,还有许多其他细胞系在不同“外衣的伪装”下盛行于世界各地的实验室。因而,许多研究领域(尤其是肿瘤学、生物化学/分子生物学,药理学和细胞生物学等领域)的研究者都深受其害。
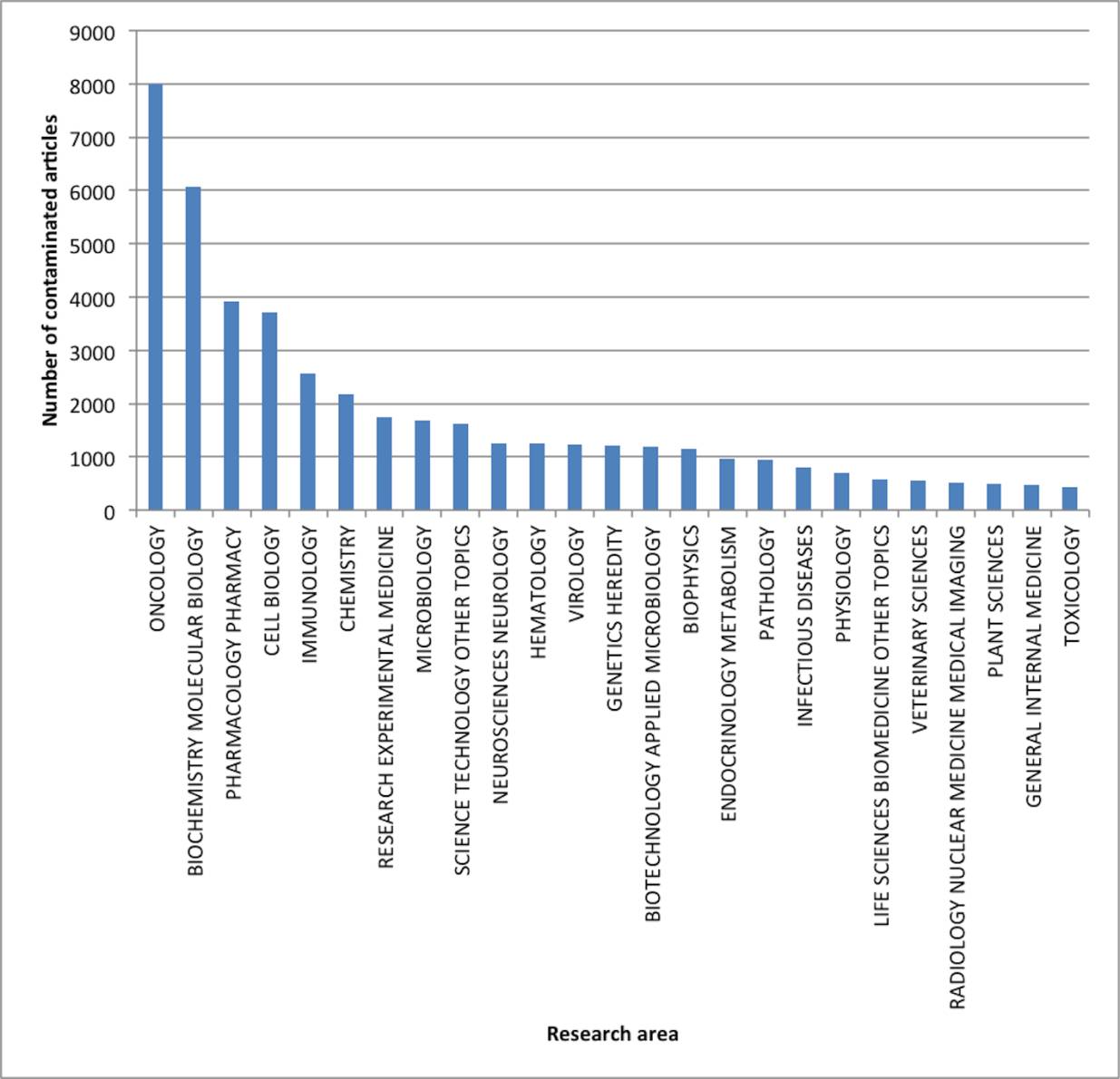
而针对这50年期间(1950年-2000年)细胞错误鉴定被长期忽视的现象,这三大群体需要负责:科研者、期刊杂志和基金管理部门。论文作者本人其实并不情愿发表一项基于使用错误鉴定细胞的声明;而期刊杂志编辑则担忧引入细胞鉴定质控后,有意愿投稿其杂志的作者人数将减少。
其中,最令人惊奇的还是基金管理组织的态度,他们总是会拒绝考虑处理甚至不承认细胞错误鉴定问题。例如,NIH发表于2007年的通告就忽视了科学家个体和审稿人未能解决细胞错误鉴定这一问题。
好在近年来,他们的态度均已发生转变,NIH、ATCC、Nature和Science等杂志就对此现象多次发出呼吁,要求研究者对细胞进行鉴定。
Nature杂志更是从2015年5月份起就已经要求投稿者审查论文中使用的细胞系,以此来确保实验数据的准确性。但值得注意的是,如果一项研究使用了错误鉴别的细胞系,而作者却能给出合理的解释,说明错误的细胞系不影响研究结果,那么文章也不会被自动拒稿。反之,若Nature杂志认为理由不充足,也可能会要求作者删除数据。
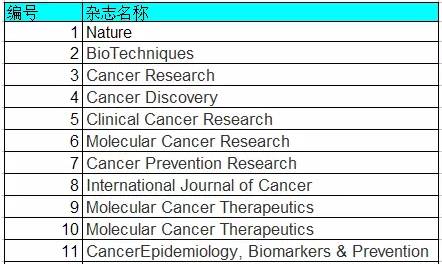
除了Nature杂志外,还有以下期刊杂志要求投稿者做细胞系审查鉴定。
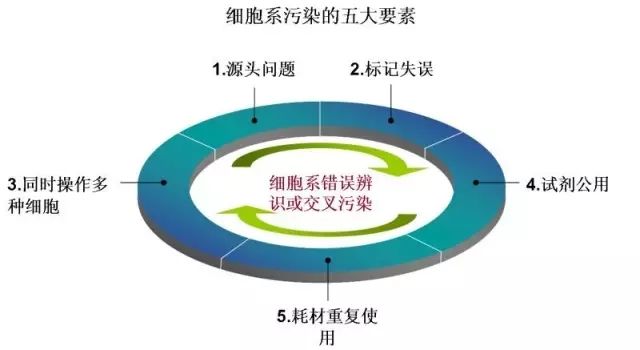
通常,细胞的培养被认为是一种对技能少有要求的技术,其所用的必要设备如孵育箱等使用起来并无限制,因而,此种条件下尝试建立一种新的细胞系会导致交叉污染,也是不足为怪的。在送至德国的微生物和细胞培养收藏所的550个白血病和淋巴瘤细胞系中,59/395(15%)被初创者送来的细胞系以及23/155(15%)的二级来源是错误的。
或许细胞错误鉴定最直接的原因就是实验员在常规操作过程中对细胞培养容器进行了错误标记,而这种情况多由工作超负荷或缺乏注意力所导致。然而就这么恍恍惚惚的随手一贴,你的细胞就已经不是你认识的那个细胞了。
另外,细胞的交叉污染以及污染细胞的过量生长也是细胞鉴定错误的常见原因。试剂公用、耗材重复使用或同时操作多种细胞均有可能导致这样的结果。
而在长期的细胞培养中,即使没有外源细胞的入侵,细胞也有可能因为培养的环境而发生一定的改变,细菌、支原体的污染或者药物的处理也可能会改变细胞的种类。如此看来,不仅来自外界的污染令人防不胜防,自家的叛徒也着实让人心焦。
显然,由被交叉污染或错误辨识的细胞系不仅会导致研究结论错误、结果不可重复以及临床细胞治疗中产生的灾难性后果,也会严重浪费科研者的时间、精力和金钱,让科研者们头疼不已。
为了避免错认细胞系,不少方法已被用来检测交叉污染,包括同工酶分析、核型分析、人类白细胞抗原(HLA)分型,免疫分型和DNA指纹识别等。这些方法都可以鉴别出某一种细胞系,但是分辨能力各不一样,且由这些方法在不同实验室所得到的数据也不尽相同,以致没有任何一种方法可以用来建立一个标准参考数据库。
目前,ICLAC、ATCC等权威机构鉴定细胞系的“金标准”是通过STR(Short Tandem Repeat,短串联重复序列)分型来做细胞鉴定。而STR分型确是具有极大优势的:快速、经济、易于自动化,能得到可重复的数据,且数据格式适合建立一个标准参考数据库等。

现阶段许多实验室均采用STR分型(已广泛用于亲子鉴定)来鉴定人源性细胞系。这不仅是因为STR位点具有高度多态性,也是因为每一个STR位点均能被PCR扩增且扩增产物标记上不同颜色的荧光,使得扩增产物很容易通过片段大小和颜色来区分。随后就可根据所得的STR分型结果与专业的细胞STR数据库比对,进而推算出样品所属的细胞系或可能的交叉污染的细胞系名称。
而STR分型的唯一局限性,就在于它不能检测来源于其他物种的污染细胞,因为用人类或高等灵长类特异性引物不会扩增其它物种细胞的DNA。此时,需要在PCR中采用物种特异性引物来检测来源于其它物种的污染细胞。但如果应用于其它物种的STR分型已被建立(目前局限于少数几个具有重要商业价值的物种),STR分型无疑可被用来鉴定污染细胞。
参考文献:
1.The ghosts of HeLa: How cell line misidentification contaminates the scientific literature (doi:https://doi.org/10.1371/journal.pone.0186281)
2.Origin of theU87MG glioma cell line: Good news and bad news
(doi:10.1126/scitranslmed.aaf6853)
3.Cell line misidentification: the beginning of the end(doi:10.1038/nrc2852)















