浙大生信博士团队倾力打造的一个科研人员学习交流的公众微信平台。我们致力于科研社区服务,分享最前沿的科技进展,提供生信分析方法,解读经典分析案例,公众数据库的挖掘和临床数据统计分析。在此我们欢迎各位的加入!戳这里生信草堂公众号原文,请多关注哦~
近年来,环状RNA(circRNA)成为新兴的明星分子,越来越多的环状RNA被发现参与众多的病例生理过程。随着高通量测序技术的日益成熟,基于测序reads的环状RNA预测方法成为人们讨论的热点。来自于丹麦奥尔胡斯大学(Aarhus
University)的研究人员利用普通的RNA-Seq数据比较了5种常用的环状RNA预测软件(见表1)。
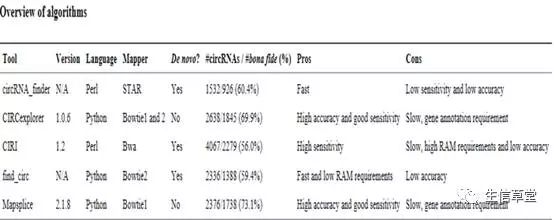
表1. Overview of algorithms
首先,研究人员用5个软件分别对同一个rRNA-depleted RNA-Seq数据集进行分析。他们发现各个算法给出的环状RNA数目从1500(circRNA_finder)到4000(CIRI)不等,并且只有854个同时被5个软件发现(图1)。
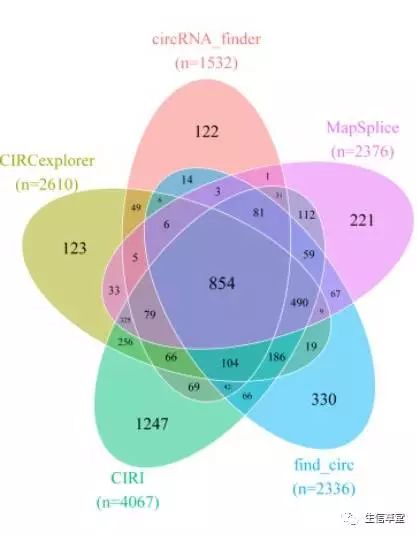
图1:5种常见环状RNA预测软件结果对比
为了验证软件给出的circRNA是否可信,研究人员试图引入线性RNA酶消化(RNase R)的RNA-Seq数据来判断预测到的circRNA是否存在假阳性。
结果显示不同的软件给出的circRNA对RNase R的抵制效率不同,其中,CIRI表现最差,有28.03%的假阳性率(见图2)。
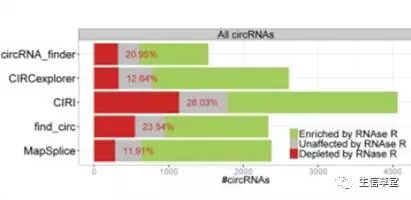
图2:各个软件预测假阳性率比较
研究人员还关心每个软件预测出的表达量最高的100个circRNA是否真的是环状。他们分别以junction read数目对环状RNA进行排序,观察表达量高的前100个环状RNA是否被线性RNA酶消化。
同样,在CIRI的预测中高表达的环状RNA有超过半数(63%)不可靠。MapSplice和CIRCexplorer是表现最好的两款软件,分别只有9%和6%的circRNA被消化(图3)。
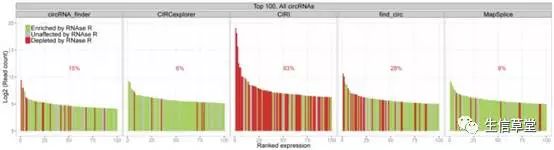
图3:Top100环状RNA可信度比较
通过比较现有的circRNA预测软件,我们可以看到不同的算法表现差异较大,用户在使用的时候需要小心。
CIRCexplorer和MapSplice输出最可信的circRNA列表,主要的原因是这两个算法依靠已知的基因注释文件,明确的序列注释信息可以帮助他们降低假阳性率,但也限制了这两个软件不能发现de novo的环状RNA。
CircRNA_finder和find_circ也有着很高的准确性,并且这两个软件可以独立于基因注释信息运行,预测全新的环状RNA。
由于单个软件往往在一个方面存在着一定的局限性,且数据表明能够被多个算法预测到的环状RNA有着较高的可信度,因此,在实际项目中,推荐大家多使用两到三个环状RNA预测软件,进而取它们的交集。
我们也会在接下来的推文中重点讨论几个预测软件的使用,欢迎大家持续关注。
参考文献:
Hansen
T B, Venø M T, Damgaard C K, et al. Comparison of circular RNA
prediction tools[J]. Nucleic Acids Research, 2016, 44(6):e58.
点公众号菜单里科研攻略-数据挖掘,查看往期系列~














