上期我们已经介绍了如何对测序下机数据进行拼接,过滤低质量,嵌合体,以及去接头等详细的操作步骤,基于上期质控后的clean序列,本期我们将介绍微生物分析流程的聚类OTU和物种注释模块。通常情况下,两端reads拼接后得到的序列数目远大于环境中物种数目,为了能够从各层级物种水平分析物种的种类和丰度,需要对序列进行聚类和注释。
上期去除嵌合体后的序列seqs.nonchimeras.fna文件作为这里的输入文件,并重新命名为
sample.fa
;
参考序列为Greengene的参考序列
97_otus.fasta
和物种分类注释
97_otu_taxonomy.txt
;
安装Qiime软件;
通过设定一个阈值,将相同或者相似的序列归为一个OTU;使用Qiime软件里的pick_otus.py,并选择默认算法uclust,相似性97%(默认)进行OTU聚类
python pick_otus.py -i sample.fa --denovo_otu_id_prefix OTU -l sample_otus.log --threads 3
-i
输入文件;
--denovo_otu_id_prefix OTU
名的前缀;
-o
输出文件夹;
-m
聚类方法, sortmerna, mothur, trie, uclust_ref, usearch, usearch_ref, blast, usearch61, usearch61_ref,sumaclust, swarm, prefix_suffix, cdhit, uclust共14种可选,默认为uclust;
-c
聚类算法,furthest, nearest, average三种可选,默认为furthest;
-s
相似度,默认为0.97;
--minlen=MINLEN
序列最短长度,默认为64;
--usearch_fast_cluster
快速比对,默认为false;
-r
参考序列
-z
容许序列反向后聚类,默认为false;
--threads=THREADS
线程数目,默认为1。
输出文件;
sample_otus.log
#日志文件,程序运行的一些参数等信息,见图1
sample_otus.txt
# 聚类结果文件,第一列为OTU编号,后面为序列名称,每条序列以tab隔开,见图2
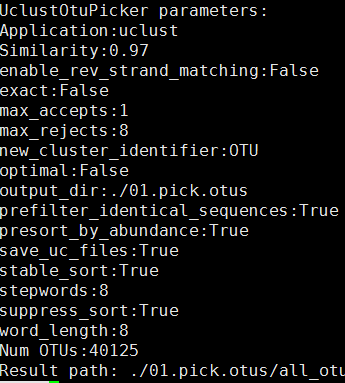
图1 日志文件
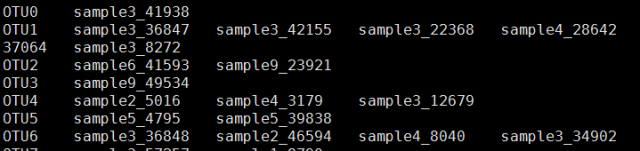
图2 聚类结果文件
由于每个OTU中的序列不完全相同,因此需要选取一条代表性序列作为该OTU的序列,用于后续分析;
python pick_rep_set.py -i sample_otus.txt -f sample.fa -o sample_rep_set.fasta
-f
输入的序列文件,即拼接获得的序列文件,fasta格式;
-m
选取代表序列的方法,可选择random, longest, most_abundant, first,默认为fisrt;
-o
输出的代表性序列文件;
-l
日志文件;
-s
基于OTU或者序列名排序,默认OTU;
-i
输入OTU聚类文件;
输出文件
sample_rep_set.fasta
对每个OTU代表性序列进行注释,可以认为每个OTU近似为一个物种;
Python assign_taxonomy.py --reference_seqs_fp database/97_otus.fasta --id_to_taxonomy_fp database/97_otu_taxonomy.txt -i sample_rep_set.fasta -o rdp_assigned_taxonomy





