|
|
各位小伙伴们大家好,江江包又来啦,江江包致力于为小伙伴们提供独特而前沿的课题方向和研究思路,今天江江包与大家一起来学习一篇聚焦
多组学和代谢重编程
的非常新颖的文章!学到就是赚到!
代谢重编程可以说是最近的研究热点了,作为一种在肿瘤发生发展和免疫环境调节中非常重要的机制,在国自然标书的申请中也是非常热门。今天江江包来和大家一起学习一篇武汉协和医院的文章,文章
聚焦于核苷酸代谢重编程在肿瘤免疫和预后中的作用
,快来跟江江包一起看看吧:
1、多组学数据构建预后模型
:该研究整合了多组学数据,建立了核苷酸代谢指数的预后模型,使用外部数据集进行验证,并根据敲低实验验证了其在抑制胃癌细胞增殖和迁移中的重要性。
2、代谢重编程和免疫微环境的关联
:研究详细分析了不同组患者的临床特征、免疫浸润、免疫检查点分子表达等,揭示了核苷酸代谢重编程在调控肿瘤免疫微环境中的关键作用。
综合来看,该研究确定了核苷酸代谢重编程相关的预后基因,能可靠预测患者的预后,并确定了能从免疫治疗中受益的患者群体,为临床治疗提供了新的角度。~
ps:研究热点的相关研究不仅需要学习经典的研究套路,还需要拓展新的角度和方法。如果你也想在紧跟研究热点,扫码联系江江包吧,助你迅速进步!
|
|
定制生信分析
云服务器租赁
加好友
备注“66”
领取试用
|
|
|
|
题目
:
整合多组学和核苷酸代谢重编程特征分析揭示胃癌的免疫学和预后特征
杂志
:Cancer Cell International
|
研究背景
胃癌(GC)是全球第二大癌症。虽然免疫疗法可以延长患者生存时间,但并非对所有患者都有效。核苷酸代谢重编程是癌症发展的一个重要特征,与肿瘤细胞的增殖、抗凋亡、血管生成、免疫逃逸和转移密切相关。然而,目前关于核苷酸代谢与胃癌之间关系的系统性研究仍较为缺乏。研究旨在通过整合多组学数据,构建核苷酸代谢相关的预后模型,以预测胃癌患者的预后和免疫治疗反应,进而为胃癌的临床治疗提供新的策略。
研究思路
研究整合多组学数据和核苷酸代谢重编程特征,揭示了胃癌的免疫学和预后特征。首先使用无监督聚类构建核苷酸代谢相关指数(NMRI),筛选了四个特征基因,将胃癌患者分为高低两组。进一步分析两组患者的临床特征、免疫微环境、对免疫治疗的反应、药物敏感性等等,并通过敲低特征基因的实验验证了特征基因对胃癌细胞的增殖和迁移的作用。
|
主要结果
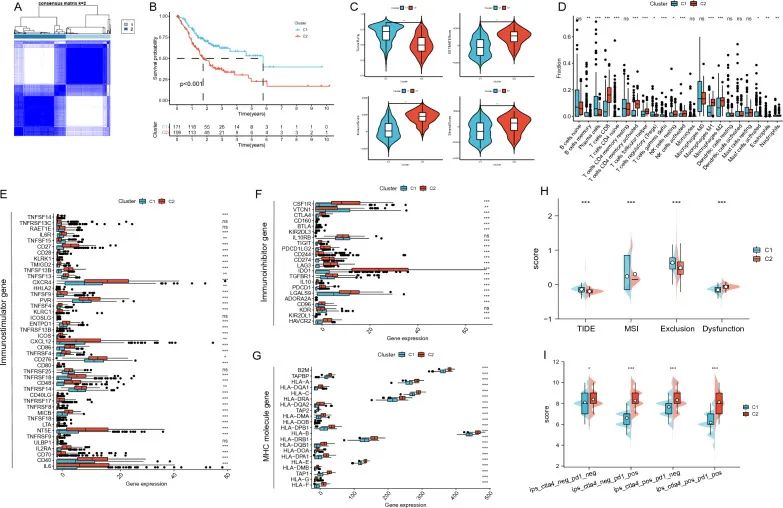
1.核苷酸代谢的聚类分析以及不同聚类之间的免疫差异
通过对胃癌患者进行核苷酸代谢相关基因的聚类分析,
患者被分为两类
,且这两类具有良好的内部稳定性和一致性(图1A)。根据Kaplan-Meier曲线(图1B),Cluster 1组患者的预后显著优于Cluster 2组(p < 0.05)。
利用ESITIMATE方法分析两组之间的免疫微环境差异,发现Cluster 2在免疫评分、基质评分、ESITIMATE评分和肿瘤纯度方面均优于Cluster 1
(图1C)。根据CIBERSORT算法(图1D),
两组之间的免疫细胞浸润水平存在显著差异,Cluster 2的CD8 T细胞比例显著高于Cluster 1(p < 0.05
)
。进一步比较两组之间的常见免疫刺激基因、免疫抑制基因和MHC分子基因的表达,发现Cluster 2的大多数免疫刺激基因、免疫抑制基因和MHC分子基因的表达均显著高于Cluster 1(图1E-G)。此外,Cluster 2的TIDE评分和功能障碍显著高于Cluster 1,而Cluster 1的MSI和排除评分则显著高于Cluster 2(图1H)。最后,
分析了抗PD-1和抗CTLA-4治疗的效果
,结果显示CTLA-4(-)PD-1( +)、CTLA-4( +)PD-1(-)和CTLA-4( +)PD-1( +)的评分在Cluster 2中显著高于Cluster 1,
表明Cluster 2对抗CTLA-4或抗PD-1治疗的效果显著优于Cluster 1
(图1I)。
|
|
|
|
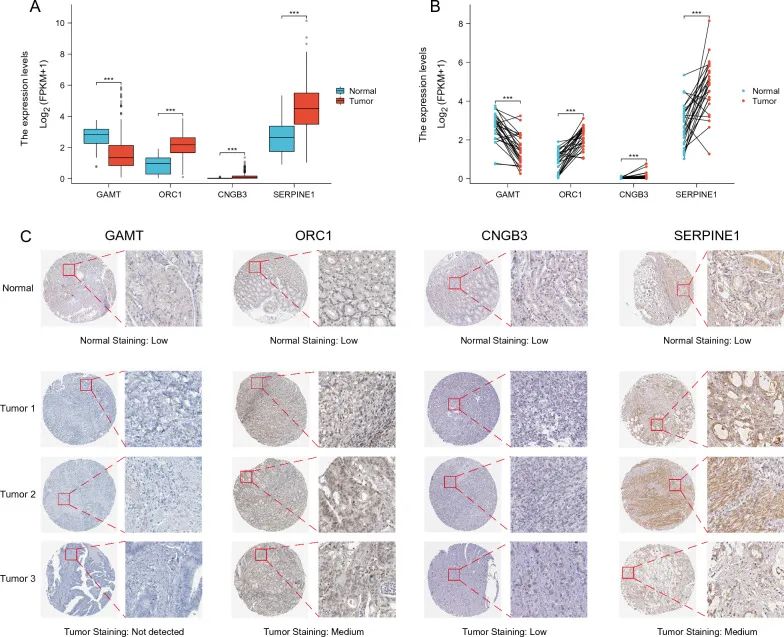
从GeneCards数据库中收集了核苷酸代谢相关基因
,使用单因素Cox回归分析,构建了核苷酸代谢相关指数(MNMRI),
筛选出四个具有独立预后价值的基因
。比较TCGA数据库中未配对胃癌组织和正常胃组织,结果表明
GAMT基因在胃癌组织中的表达显著下调,而ORC1、CNGB3和SERPINE1的表达则显著上调
(图2A)。在配对的胃癌组织和正常胃组织中也观察到了类似的表达差异(图2B)。此外,
从HPA数据库中获取了免疫组织化学染色结果
,GAMT在胃癌组织中的IHC染色强度降低,而ORC1、CNGB3和SERPINE1在肿瘤组织中的IHC染色强度均有所增加,验证了预后模型的结果(图2C)。这些结果表明,
GAMT、ORC1、CNGB3和SERPINE1基因在胃癌中的表达模式与其在正常组织中的表达显著不同
。
|
|
|
|
分析了NMRI对预测胃癌患者预后的作用。比较高低NMRI组在总生存期(OS)、疾病特异性生存期(DSS)、无病间隔期(DFI)和无进展生存期(PFI)方面的差异,结果表明高NMRI组的预后显著差于低NMRI组(图3A-D)。
使用GSE84437队列作为验证队列
,发现
高NMRI组患者的预后显著差于低NMRI组
(
图3E)。单因素和多因素回归分析结果表明,NMRI是一个独立于其他临床特征的风险因素(图3F-G)。此外,不同临床特征(T、N、M和分级)之间的NMRI评分差异显著(图3H)。
ROC曲线显示了NMRI是短期和长期胃癌患者生存状态的更准确的预测指标(图3I-J)
。最后,基于NMRI和其他临床特征(年龄、临床分期、T、N和M)构建了列线图(图3K-L)。这些结果表明,NMRI在预测胃癌的预后方面非常有效。

|
|
|
|
4.
NMRI与肿瘤微环境的基因集富集分析及相关性研究
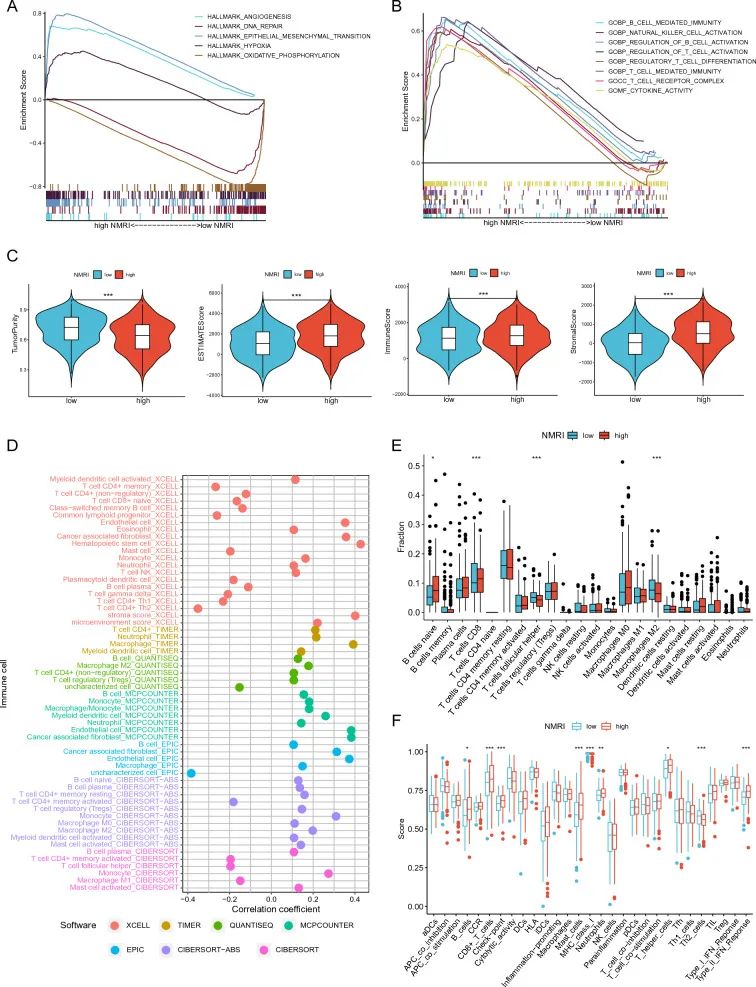
为了探索与NMRI相关的癌症特征通路,
在高低NMRI组进行了GSEA分析
。高NMRI组在血管生成、上皮间质转化和缺氧信号通路中显著富集,而低NMRI组则显著富集在DNA修复和氧化磷酸化信号通路中显著富集(图4A-B)。在肿瘤微环境得分(ESTIMATE得分、免疫得分和基质得分)方面,
高NMRI组的得分显著高于低NMRI组
,而肿瘤纯度得分则显著低于低NMRI组,这表明高NMRI组具有更高水平的免疫浸润(图4C)。
使用ESTIMATE算法评估胃癌患者肿瘤微环境中的免疫细胞浸润
,结果显示NMRI与大多数免疫细胞呈正相关(图4D)。CIBERSORT算法结果表明,高NMRI组的免疫刺激性CD8 T细胞水平显著高于低NMRI组,而免疫抑制性M2型巨噬细胞水平则显著低于低NMRI组(图4E)。此外,ssGSEA算法的结果显示,
高NMRI组患者的免疫细胞浸润和免疫相关活动水平显著高于低NMRI组
(图4F)。这些结果表明,高NMRI值的患者在胃癌中可能也具有显著的免疫浸润水平。
|
|
|
|
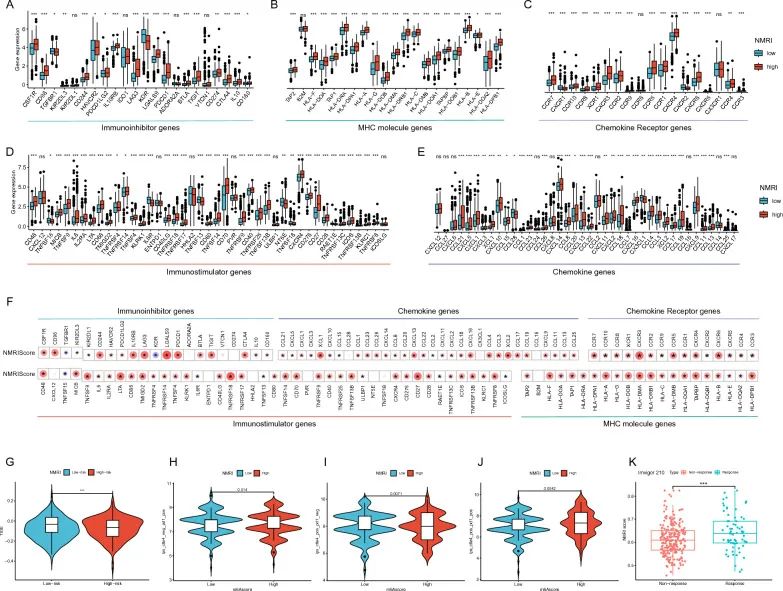
为了研究NMRI与免疫微环境的关系,
比较高低组之间常见的免疫检查点、MHC分子、细胞因子和受体的表达水平
。高NMRI组中大多数这些基因的表达显著高于低NMRI组(图5A-E)。进一步分析显示,
NMRI与免疫刺激基因、免疫抑制基因、MHC分子、细胞因子和细胞因子受体之间存在显著的正相关关系
(图5F)。TIDE评分结果显示,高NMRI组的TIDE评分显著高于低NMRI组,表明高NMRI组的免疫检查点抑制剂治疗效果更好(图5G)。此外,免疫原性核心(IPS)评分结果显示,高NMRI组的IPS评分显著高于低NMRI组,
表明高NMRI组患者对免疫治疗的响应更好
(图5H-J)。最后,
通过外部真实免疫治疗数据集(Imvigor210)验证NMRI对免疫治疗效果的预测
,结果显示响应
抗PD-L1免疫治疗的患者组的NMRI评分显著高于非响应组
(图5K)。这些结果表明,NMRI可以预测胃癌患者对免疫治疗的反应,高NMRI组患者可能会获得更好的免疫治疗效果。
|
|
|
|
检查了高/低NMRI组对标准胃癌化疗和靶向治疗药物的反应
,以便为这些患者的临床用药情况提供信息.。结果显示低NMRI组对顺铂、吉西他滨、甲氨蝶呤、二甲双胍和吉非替尼等药物的敏感性更高,而高NMRI组对帕唑帕尼、倍他洛汀、达沙替尼、伊马替尼和舒尼替尼等药物的敏感性更高(图6A-B)。此外,分子对接结果显示,对SERPINE1蛋白结构(ID: 7AQG)进行分子对接后,
发现了六种与SERPINE1结合亲和力最高的小分子药物
,分别是Fenugreekine、Portulacaxanthin II、Leucovorin、Kuwanon J、Blumeatin和Schizotenuin F(图6C-H)。例如,Portulacaxanthin II与SERPINE1的氨基酸序列Gln-123、Thr-120、Met-110、Gly-108和Leu-105形成氢键,其中Gln-123作为受体,Thr-120、Met-110、Gly-108和Leu-105作为供体。
|















