这或者可能几乎是最新最全(适应最新广告法)的KEGG数据库使用说明了。KEGG数据库介绍将分为两期推送,本期主要介绍KEGG数据库的基本信息以及之间的联系,下期将介绍KEGG数据库的注释方法以及其他软件实现途径和神秘的API。
(文章图片略多,建议在WiFi下阅读,土豪随意。)
KEGG,(Kyoto Encyclopedia of Genes and Genomes,京都基因与基因组百科全书),是一个整合了基因组,化学和系统功能信息的数据库,旨在揭示生命现象的遗传物质与化学蓝图。它(http://www.kegg.jp/kegg/)是由日本的 Kanehisa Laboratories于1995年创建整理的一个知识库,是基于使用一种可计算的形式捕捉和组织实验得到的知识而形成的系统功能知识库。另外,KEGG数据库具有强大的图形功能,它利用图形介绍众多的代谢途径以及各途径之间的关系。
KEGG数据库是一个综合数据库,其主要分为四类,分别是系统信息,基因组信息,化学信息,健康信息。可以通过颜色编码区分。KEGG四类数据库之间的关系如图1.
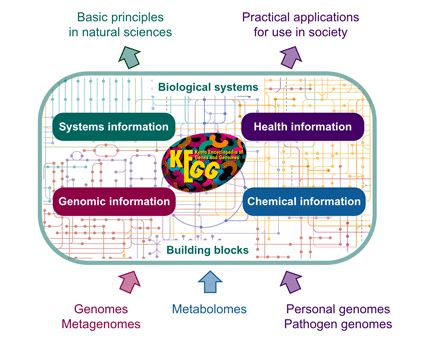
图1 KEGG四类数据库之间的联系
其中这四类又分别包含不同的子库。KEGG数据库中包含各种各样的数据对象,这些数据对象是为了用来对生物系统进行计算机模拟的。因此,各个数据库中的数据记录都被称为KEGG对象。这些对象可以通过KEGG对象标识符来识别,标识符由一个与数据库相关的前缀加五个数字构成。具体如图2所示。核心数据库是
KEGG PATHWAY
和
KEGG ORTHOLOGY
数据库。
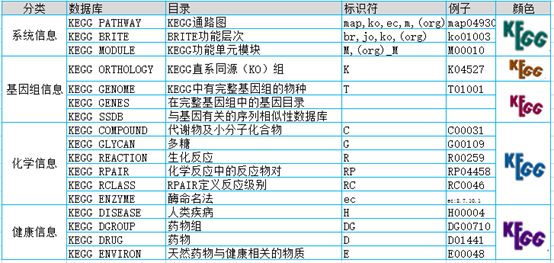
图2 KEGG子库目录
-
KEGG PATHWAY Database(
代谢通路数据库
)
-
KEGG BRITE Database(
分层分类数据库
)
-
KEGG MODULE Database
(功能模块数据库)
-
KEGG ORTHOLOGY Database
(直系同源数据库)
-
KEGG GENES Database(
基因数据库
)
-
KEGG GENOME Database(
基因组数据库
)
2.1、KEGG PATHWAY Database
KEGG PATHWAY数据库是一个手工画的代谢通路的集合,包含以下几方面的分子间相互作用和反应网络:新陈代谢、遗传信息加工、环境信息加工、细胞过程、生物体系统、人类疾病、药物开发。同时它也经常更新。
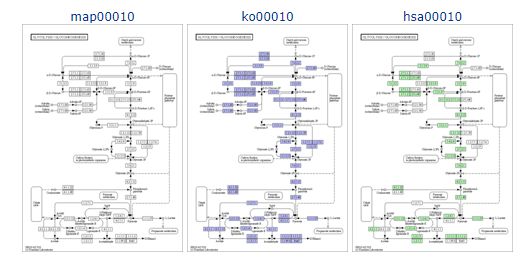
PATHWAY的五种类型:仅仅第一种参考通路(referencepathway)图是手动画出来的,其他的通路图都是通过计算产生的。pathway中的每一个框(或线)都对应一个或多个K编号、EC编号及R编号。几类代谢通路区别:
-
map -Reference pathway:对于代谢相关的通路,在referencepathway中,一个点同时表示一个基因、这个基因编码的酶及这个酶参加的反应
-
ko -Reference pathway (KO):ko通路中的点只表示基因
-
ec -Reference pathway (EC):ec通路中的点只表示相关的酶
-
rn -Reference pathway (Reaction):Reaction通路中的点只表示改点参与的某个反应、反应物对及反应类型
-
org- Organism-specific pathway map:对于所有的代谢和非代谢通路,K编号都被认为是基因的标识符,这个标识符在每一个物种中对应该物种中的某个基因,从而得到物种特异性的pathway。
同一代谢通路不同类型通路图的区别,例如Metabolic pathways通路图,如下图:
在KEGG PATHWAY 数据库(http://www.kegg.jp/kegg/pathway.html#metabolism)中查询代谢通路,如图3.
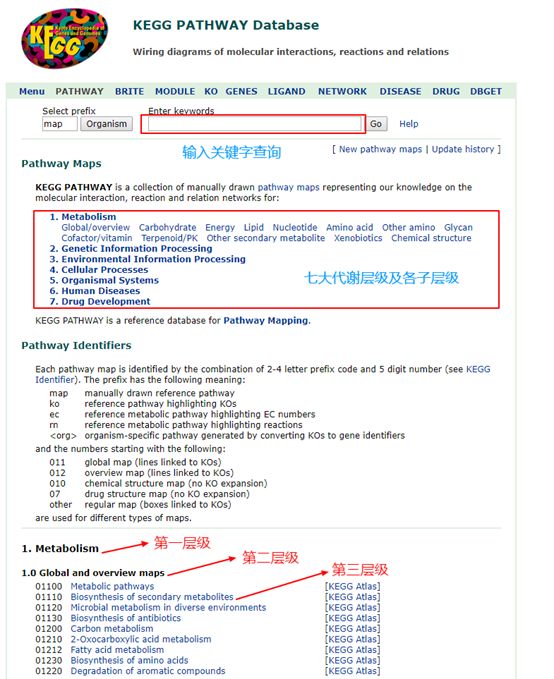
图3 KEGG PATHWAY 数据库查询
例如查询map00120代谢通路结果。如图4(http://www.genome.jp/dbget-bin/www_bget?pathway:map01200)。代谢通路中的图例说明如图5.
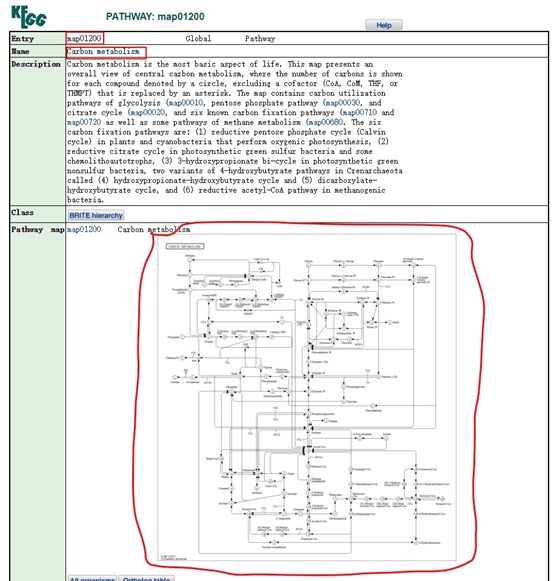
图4 map00120代谢通路图
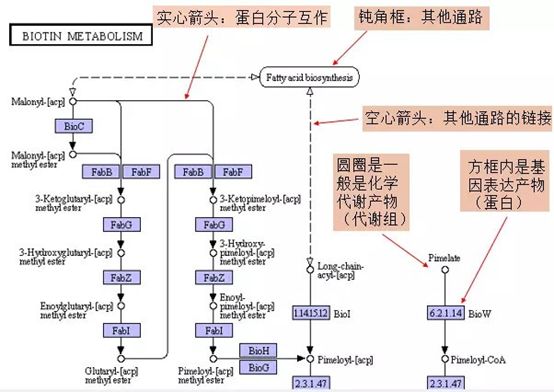
图5 代谢通路图图例说明
2.2 KEGG BRITE Database
KEGG BRITE是一个层级分类的数据库,包含生物系统各个方面的知识。相对于KEGG PATHWAY仅限于分子间相互作用和反应,KEGG BRITE包含了许多不同的关系类型。例如,可以查询酶和底物之间的关系,也可以查询某种酶的同源基因。
2.3 KEGG MODULE Database
KEGG MODULE是一个人工定义的功能单元的集合。被用于已测序基因组的注释和生物学上的解释。各个模块使用M开头的编号及与其对应的一系列K开头的编号来表示。四种主要的KEGG模块:




