今天跟大家分享的是2020年2月发表在
Nat.Commun .(IF:12.121)
杂志上的一篇文章Combined burden and functional impact tests for cancer driver discovery using DriverPower.在文章中作者描述了一种新的高灵敏度算法DriverPower,用于在全基因组和外显子组测序数据中识别区分癌症的驱动和乘客突变。
Combined burden and functional impact tests for cancer driver discovery using DriverPower
使用DriverPower识别癌症driver基因的综合负荷和功能影响测试
(分享者:科研菌-碎碎冰)
一.研究背景
与癌症发生发展相关的重要基因被称为“驱动基因(driver基因)”,这种基因决定了癌症的走向:当driver基因发生突变后,癌细胞就会活跃起来。driver基因突变占肿瘤中体细胞变异比例少,而且在大多数癌症中,肿瘤内和肿瘤间存在明显的异质性,背景突变率(BMR)都可能存在数个数量级的差异。此外,大规模癌症全基因组测序WGS的出现为人们探索driver基因在非编码区中的作用成为可能。但由于突变对基因组非编码区的影响人们了解甚少,所以也有不小的挑战。大多数最新技术通过突变负荷测试(通过将基因组区域中观察到的突变率与BMR预期的突变率进行比较)或功能影响测试来检测阳性选择信号,从而识别driver基因。由此作者团队开发DriverPower算法——使用突变负荷和功能影响评分来识别编码和非编码癌症driver基因。
二.分析流程
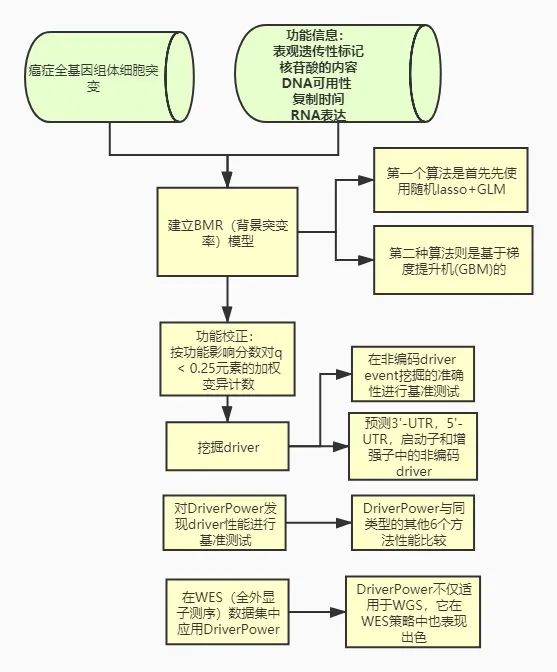
三.结果解读
1.建立BMR模型
作者首先从PCAWG项目获得WGS体细胞变异数据。在所有肿瘤队列中,作者观察到在组织,供体以及基因座水平上的突变率存在很大差异。driver基因突变检测的精确性需要准确估计整个肿瘤基因组中的BMR(背景突变率),此外还需要考虑到肿瘤类型、供体和基因组区域之间的广泛差异(图S1)。DriverPower通过使用与局部BMR共同变化的基因组特征,来建立BMR模型从而解决这个问题。
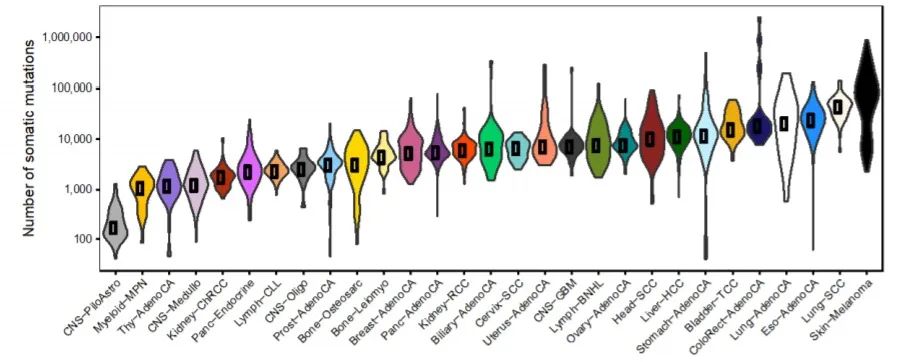
图S1.队列和供体水平的异质性
背景知识:
目前通过体细胞突变识别癌症driver基因的分析方法主要有两种:①背景突变率(BMR)法和②背景突变比例度量法。背景突变率方法的思想是,
评估一个基因在癌症样本中是否含有比预期更多的体细胞突变
。基于比率测量的方法是通过考察一个基因中不同种类体细胞突变数的比例来探测癌症driver基因。
作者研究了两种基于基因组特征的BMR建模算法。第一个算法是首先先使用随机lasso,然后是运用二项式广义线性模型(GLM),第二种算法则是基于梯度提升机(GBM,一种非线性且非参数的树集成算法)的算法。为了评估这两种BMR建模算法,通过随机采样基因组坐标,制作了不重叠的1兆碱基对(Mbp)常染色体元件(n = 2521)和训练基因组元件(n = 867,266)。然后使用五重交叉验证(cross validation,CV)来预测每个元件的突变数。
利用上述元件对模型性能进行评估时,作者的分析结果发现:将两种算法分别构建的模型应用于大型训练人群(如泛癌组)以及应用于测试元件集时,均显示出出色的性能(图1b-c)。
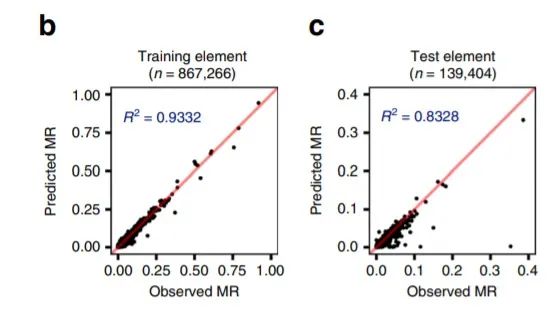
图1.两种算法分别构建的模型的性能评估
其次,作者分析发现,随机lasso+GLM和GBM均可用于以不同方式对特征重要性进行排名。两种方法的特征选择排名均显示H3K9me3(与异染色质相关)和H3K27ac(或其拮抗组蛋白标记H3K27me3)是BMR最重要的预测因子(图S2)。
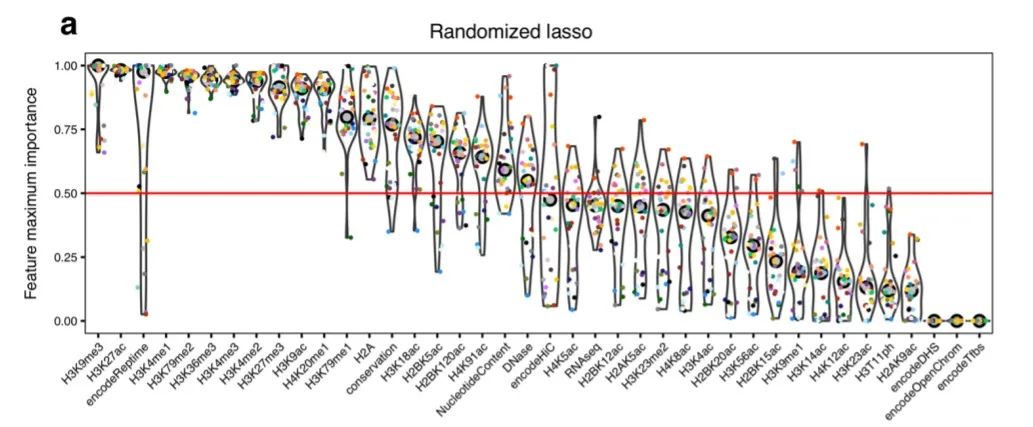
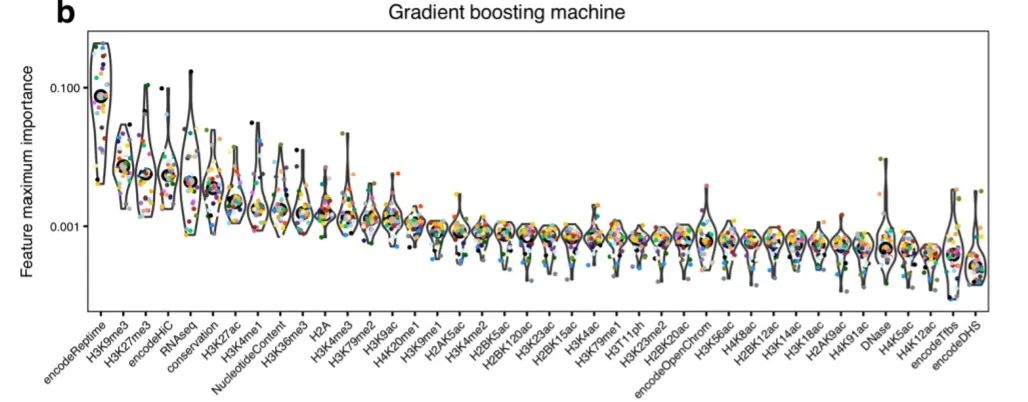
图S2.特征重要性排名
2.功能校正
在以往大多数基于负荷的方法中,均会对突变进行加权处理。但并非所有突变都具有相同的功能结果。为了合并功能结果等信息,DriverPower实现了功能校正。功能校正步骤可以增强具有较高预测功能影响的突变。在当前实施中,作者使用四个已发布的评分方案(CADD16,DANN17,EIGEN18和LINSIGHT19评分)来评估功能影响分数(图2a-b)。
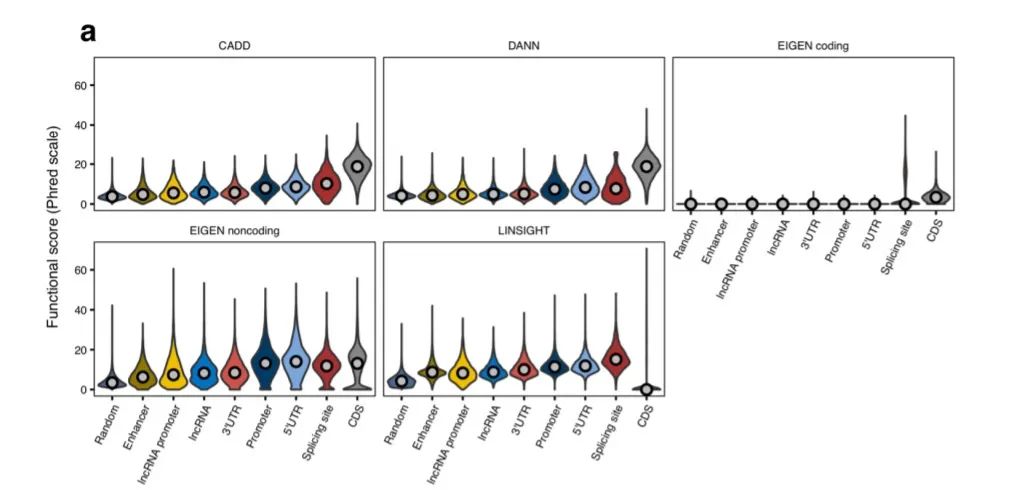
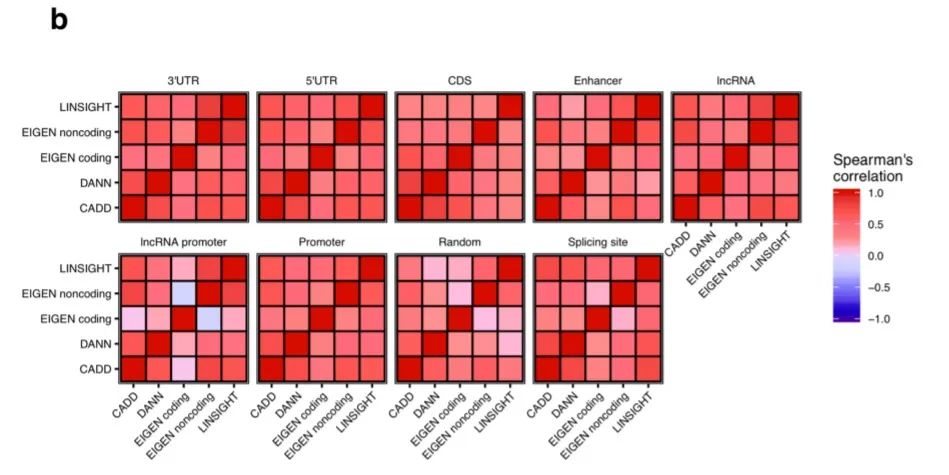
图2.功能影响评分(functional impact scores)
3.候选driver event的发现
作者根据可供参考的driver元件集和其他六个已发布方法的候选driver程序对作者的分析结果进行了基准测试。
其中,使用三个参考driver元件集为:COSMIC癌症基因普查(CGC),PCAWG原始综合driver候选(PCAWG-raw),和PCAWG-consensus driver候选(PCAWG-consensus)。另外,六种已发布的方法中,ExInAtor20,ncdDetect21和LARVA22仅使用突变负荷信息。oncodriveFML23仅使用功能偏差;而MutSig24和ActiveDriverWGS25既可以对突变负荷也能通过功能校正进行建模,但不能通过功能影响评分来建模。
-
CGC是driver的目录,其突变与癌症有关联,是编码和剪接位点驱动的金标准集(即用于计算精确度和召回率)。
-
PCAWG-raw是driver元件的集成,该驱动程序元件由12种不同的驱动程序检测方法对作者在此使用的同一数据调用。
-
PCAWG-concensus是一个保守的集合,它衍生自PCAWG-raw,但通过应用多个严格的过滤器来控制错误发现率。
作者在DriverPower结果中观察到了经过良好校准的p值(图3d),并且编码和非编码driver发现的准确性都很高(图3e)。
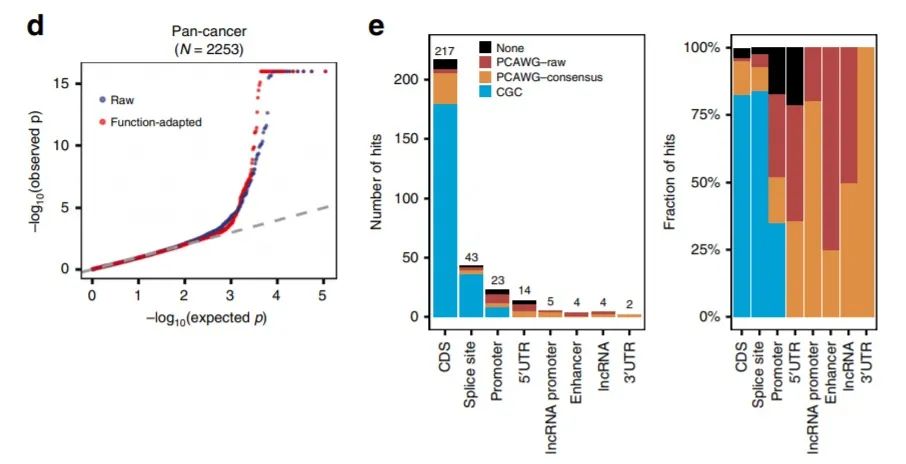
图3.泛癌队列以及由三个参考驱动程序集(CGC,PCAWG-concensus或PCAWG-raw)中包含的DriverPower调用的非编码driver候选的数量和分数
对于蛋白质编码区(CDS),作者利用DriverPower发现了217个显著的(
q
<0.1)候选驱动程序。少数基因(例如TP53)可以在多个队列中作为driver基因。而且作者发现功能信息的合并提高了编码driver发现的准确性(图 4a)。例如,在胰腺导管腺癌(Panc-AdenoCA;
N
= 232),增加“功能调整”后的算法能挖掘到三个额外的driver(
ACVR1B
,
RBM10
和
ZFP36L2
)(图4a)。而如果不合并功能信息,则CGC和CGC / PCAWG挖掘到的driver基因的整体精度均会下降。
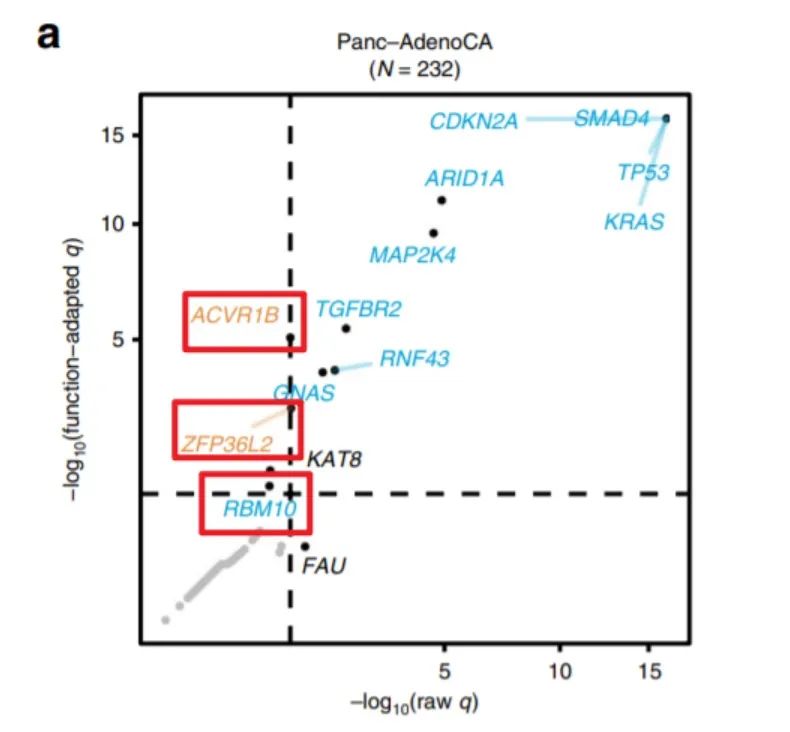
图4.合并功能信息后挖掘到三个额外的driver
均使用相同26个非黑素瘤/淋巴瘤队列和CGC作为金标准集的情况下,DriverPower与其他六种方法进行比较时,DriverPower(精度= 0.84;召回率= 0.79)的F1分数最高(0.81)(图5b-c)。
F1分数(F1 Score),是统计学中用来衡量二分类模型精确度的一种指标。它同时兼顾了分类模型的精确率和召回率。F1分数可以看作是模型精确率和召回率的调和平均,最大值为1,最小值为0。
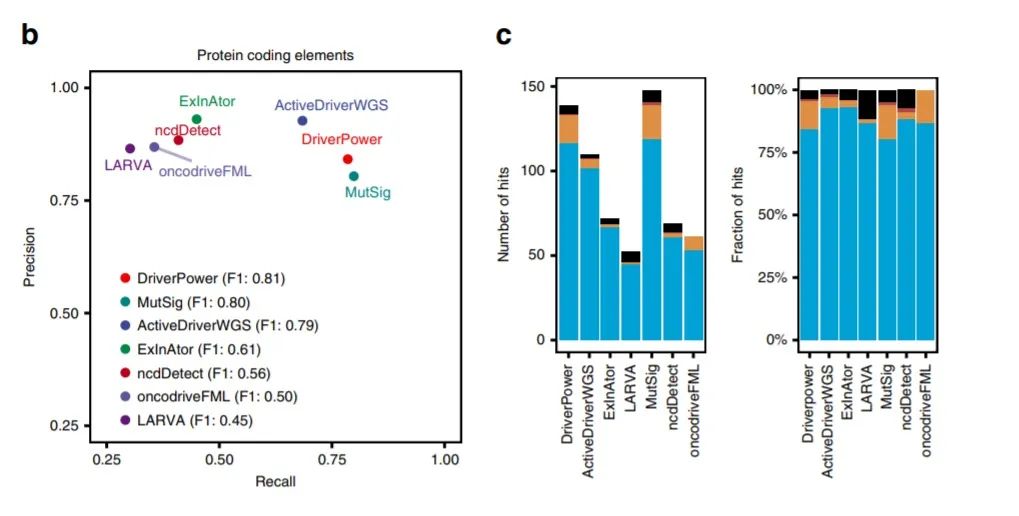
图5.DriverPower与其他六种方法F1得分比较
4.对DriverPower发现driver性能进行基准测试
接下来,作者对DriverPower在非编码driver event挖掘的准确性进行基准测试。在剪接位点driver的识别上,DriverPower(F1 = 0.91)也优于对比的两种方法:ncdDetect(F1 = 0.65)和oncoDriverFML(F1 = 0.32)(图6)。
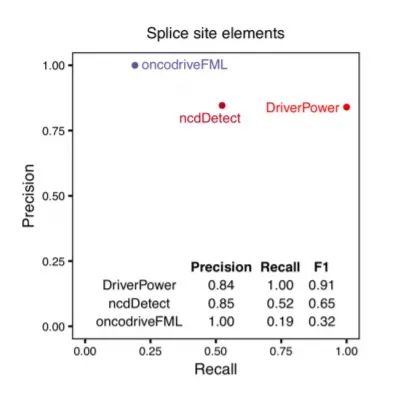
图6.预测影响编码基因剪接位点的driver















