生信草堂
将会与更多的优秀微信公众号合作,把最优秀的微信推文呈现给大家,希望可以帮助读者更多的了解生信技术,培养和提高读者的生信分析能力!
号外,号外,号外
你想和生信分析大神做好朋友么?
你想认识更多爱好生信分析的小伙伴么?
你想让自己的生信分析走上快车道么?
那就赶快加入我们的生信交流微信群吧!
正确加入我们的模式是:
添加我们的微信bioinformatics88为好友
标注“加入生信草堂交流群”
在群里请大家注明自己本名,单位,研究领域
便于小编管理
肠道菌群与人类健康的密切关系越来越受到重视,已经成为时下最热门的研究方向之一。被称为人类“第二基因组”的肠道菌群除了受到环境、饮食、疾病等因素的影响以外,还在一定程度上受到遗传因素影响,也就是说肠道微生物会受到宿主基因的控制。
那么一篇肠道菌群的GWAS研究如何做怎样才能发到高水平的期刊呢?今天就以2016年发表在Nature Genetics上的一篇文章为例,讲讲高水平的肠道菌群的GWAS研究思路是怎样的。
题目:Genome-wide association analysis identifies variation in vitamin D receptor and other host factors influencing the gut microbiota
期刊:Nature Genetics
发表时间:2016年10月
本文主要利用1812人的样本集进行GWAS研究,发现了一系列影响肠道菌群的SNP。之后针对VDR基因上的SNP为例,研究了该基因影响肠道微生物的机制。
大致可以把这篇文章的思路分为三步:

下面我们看看作者如何一步步有理有据地走下来。
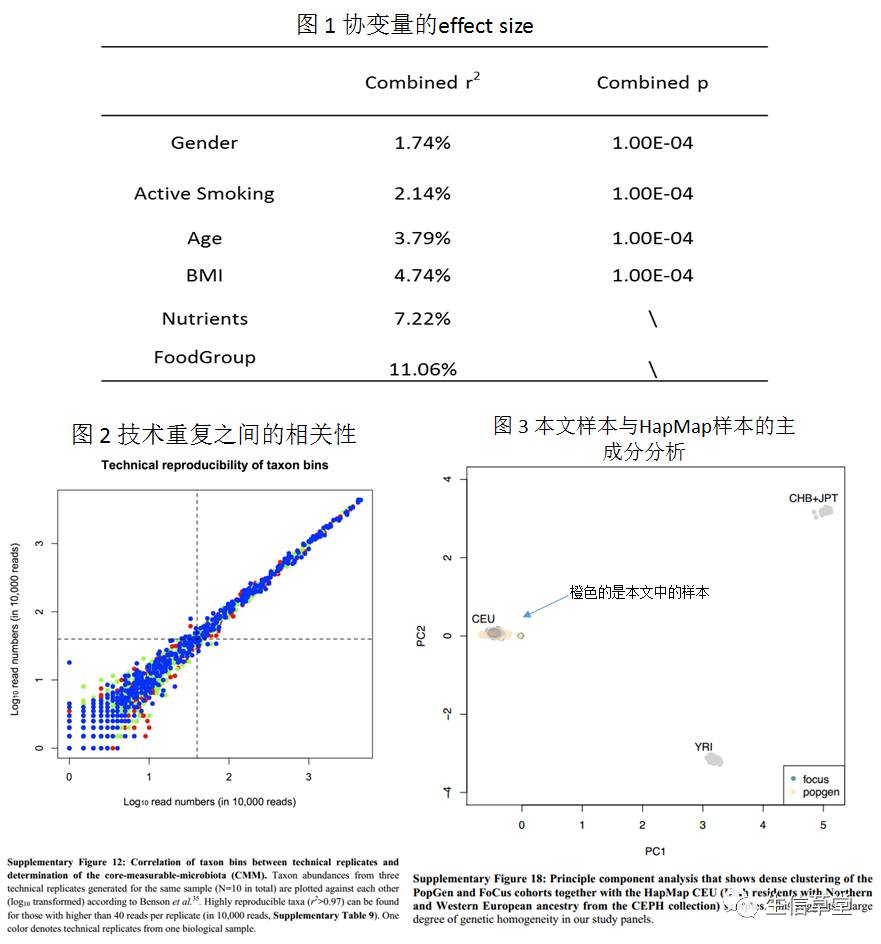
协变量:经过相关性分析确定了年龄,性别,吸烟,饮食等情况对于肠道微生物的影响都是显著的(图1),因此都被纳入关联分析作为协变量。
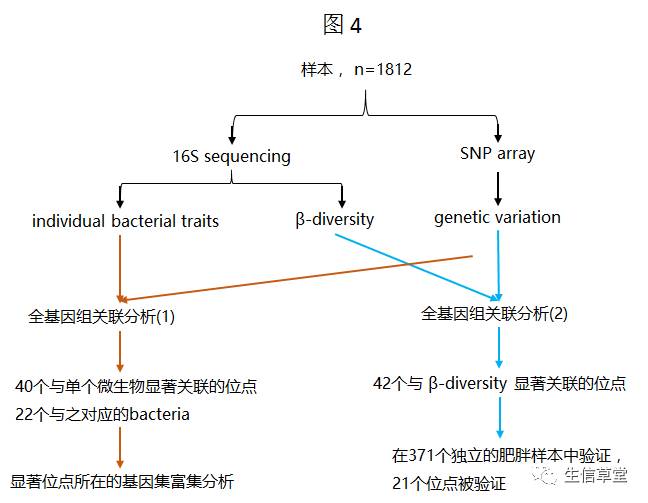
微生物筛选:通过对于10个样本做三次测序的技术重复,并计算两两重复之间的相关性。发现reads大于40时技术重复的相关性r2达到0.97(图2),因此将低于40的分类单位都去除了。最终剩下58个分类单元(core measurable microbiota)用于关联分析。
人群分层:根据计算总体的λGC = 1.00,因此没有人群分层(图3)。后面关联分析时还是将遗传的前3个主成分作为了协变量进行了校正。
样本: n=1812(FoCus + PopGen)
微生物数据: 58个分类单元和40个 40 species-level OTUs.
SNP处理: MAF >0.05 , IMPUTE2 INFO criteria >0.8.
模型: 负二项分布模型.
协变量: BMI, 年龄,性别,遗传主成分1-3, 饮食因素
结果:找到40个与单个微生物显著关联的位点,对于22个bacteria;42个与 β-diversity 显著关联的位点,其中21个在独立的肥胖样本中验证。(图4)
接下来,作者只选了一个位点rs7974353以及其对于的基因—VDR。这个位点并不是两个关联分析的交集,只在β-diversity关联分析中是显著的,但也并不是top显著的位点。
VDR基因是Vitamin D受体基因,而在第一组关联分析后的富集分析结果在出现了response to vitamin 以及 response to vitamin D response to vitamin A等关键词(图5),而且P值非常显著。提示该基因可能与β-diversity以及某些微生物关联。且根据文献查询结果,该基因的产物会形成VDR-RXR二聚体,VD、微生物的次级代谢产物 secondary bile acid 等都可以作为配体与之结合从而发挥功能(图6)。因此作者后面选择该基因作为研究对象。

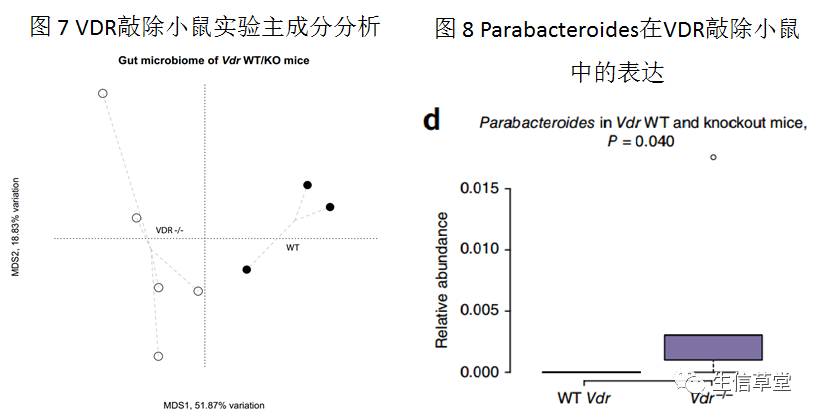
在VDR基因敲除小鼠中证明了VDR基因确实会影响肠道菌群的β-多样性(图7)。同时还发现敲除后的小鼠 Parabacteroides 显著上升(图8),因此VDR与 Parabacteroides 呈负相关。
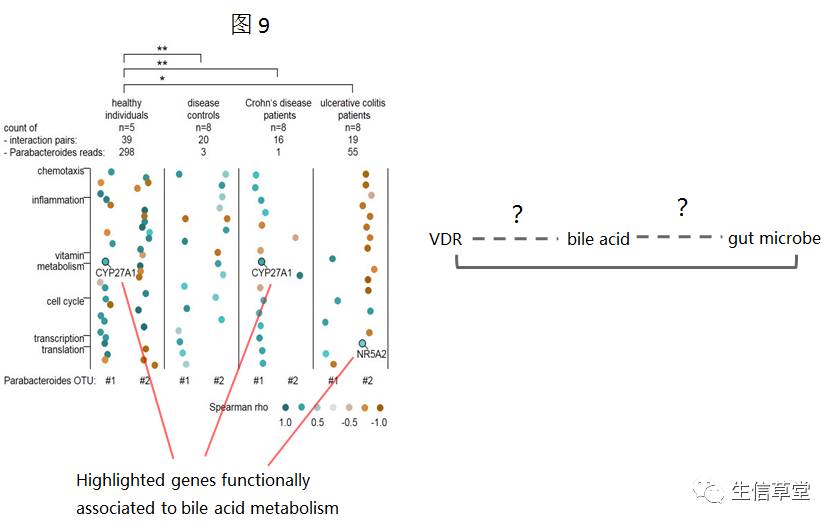
59个结肠活检样本(case/control),疾病组中VDR上调,相应的 Parabacteroides 下调,呈负相关,与小鼠实验中一致。此外在该实验中,还发现bile acid代谢相关基因的表达与 Parabacteroides 的丰富显著相关(图9)。因此提出VDR基因通过影响bile acid代谢来影响肠道微生物(例如 Parabacteroides)。下面,就要验证虚线的关系是否成立。
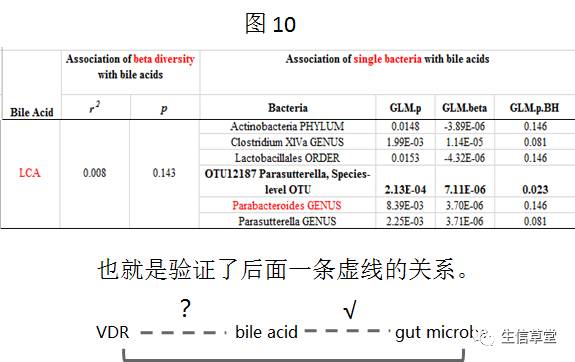
为了验证bile acid是否与肠道菌群相关,作者取了551个样本检测了血清中的5中bile acid 和fatty acid含量,然后与肠道菌群做相关性分析。发现多数bile acid确实与肠道菌群显著关联,其中bile acid 与 Parabacteroides 的相关性也是显著的(图10)。
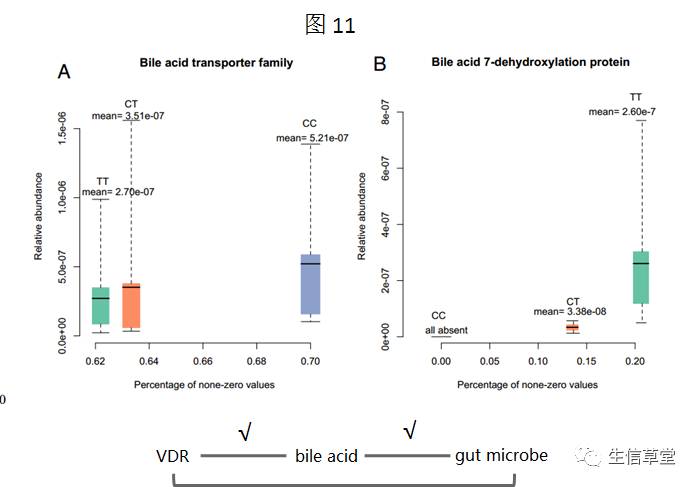
为了验证VDR与bile acid 代谢的相关性,作者选取了122个样本做了肠道菌群的全宏基因组测序,组装得到基因和通路的丰度,并选出与bile acid代谢相关的基因。发现VDR genotype也显著影响肠道微生物中VDR代谢通路的基因丰度(图11)。
本文通过全基因组关联分析,得到了一系列影响肠道菌群的SNP,通过基因功能筛选确定VDR基因为研究目标。并通过一系列分析找到VDR基因可能通过bile acid代谢影响肠道菌群。最后验证了 "VDR genotype--bile acid代谢—肠道菌群” 这样一个调控通路。
Nature Genetics: GWAS证明遗传和饮食决定肠道微生物组结构
circRNA高分文章解读——高通量分析篇(上)
公共数据库,SCI怎么发?(三)
肠道微生态|Nature Medicine揭秘“神药”二甲双胍降糖新机制
GWAS研究的转机|转录因子(PU.1)参与调节阿兹海默症(AD)
精品论文|如何利用分子实验验证GWAS发现的SNP?

















