前面两期分别跟大家分享了系统发育树(一)和系统发育树的构建(二)的基础知识,为什么可以构建系统发育树,为什么要构建系统发育树,以及构建系统发育树的基本流程和不同算法构建系统发育树的优缺点,今天就以一个实例跟大家分享一下具体的构建系统发育树的流程:
序列的准备
多序列建树的话,首先需要将多条序列合并为一条序列,然后再进行多序列比对。由于“.fasta”格式的序列有特殊的格式,所以直接用“cat”命令就可以进行合并,或者用Geneious进行合并,导出fasta格式的序列即可。将所有用于构建系统发育树的序列放在同一个文件夹下,进行合并。
cat *.fasta > tree.fasta

序列比对
使用Mafft进行多序列比对,Mafft的安装可以参考Mafft多序列比对
/path/to/mafft --thread 24 --auto tree.fasta > tree_mafft.fasta
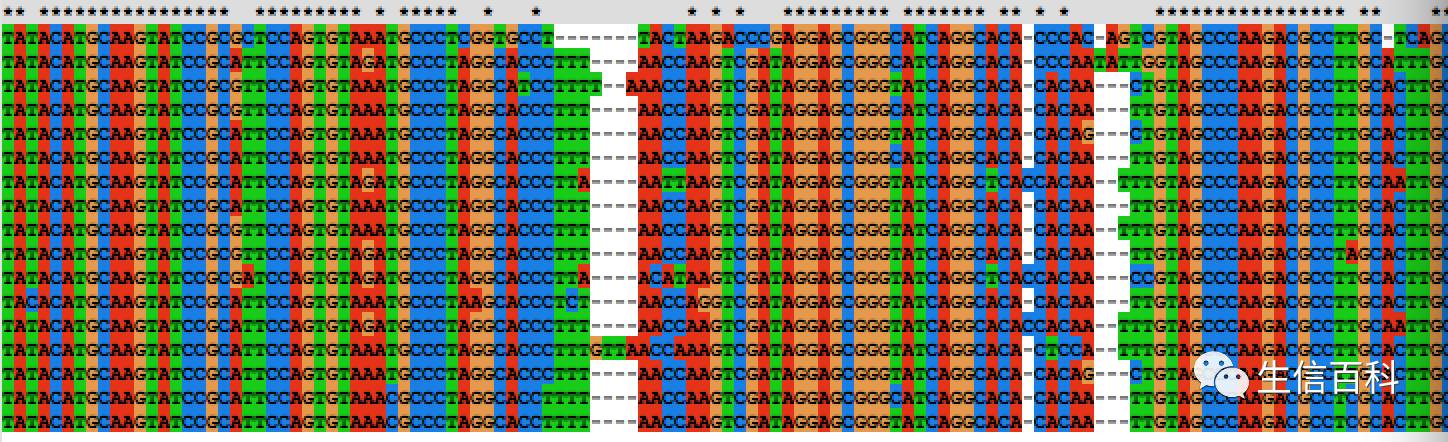
如果你有特殊的要求,请查看mafft的详细参数使用。
去除有争议的位点
使用Gblock进行去掉有争议的位点,可根据实际情况来设定参数。一般情况下,信息位点足够的情况下,默认参数就可以了。
/path/to/Gblocks tree_mafft.fasta -t=p
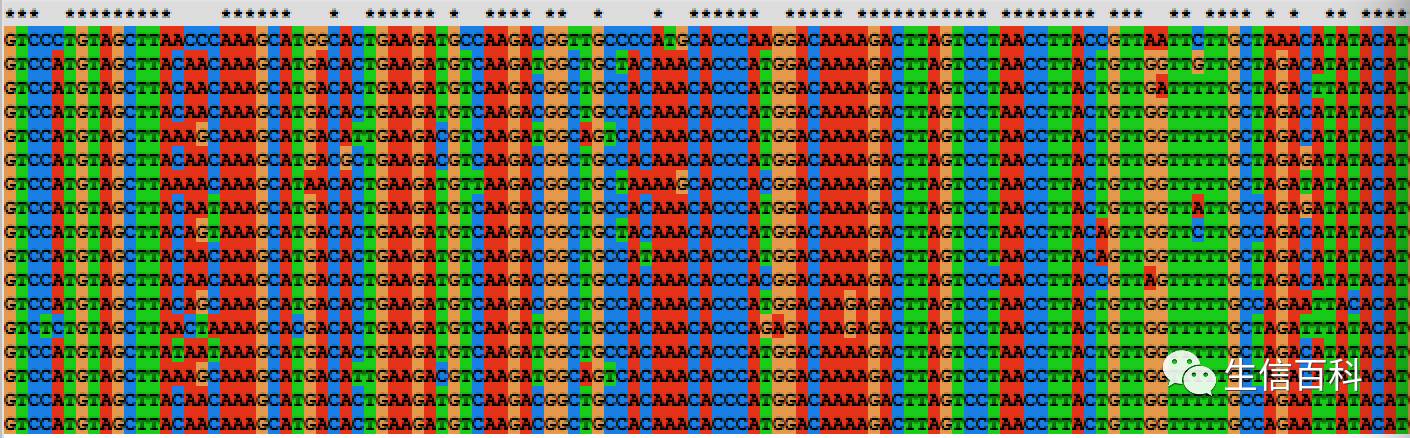
得到结果文件tree_mafft.fasta-gb
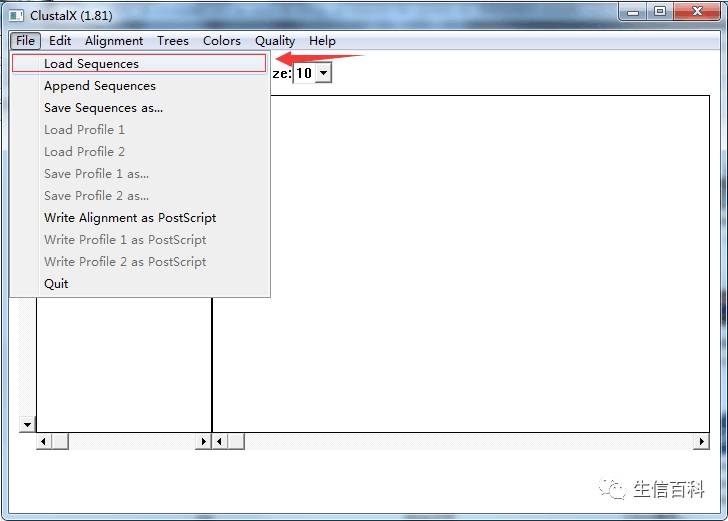
clustalx转格式
第一步
第二步 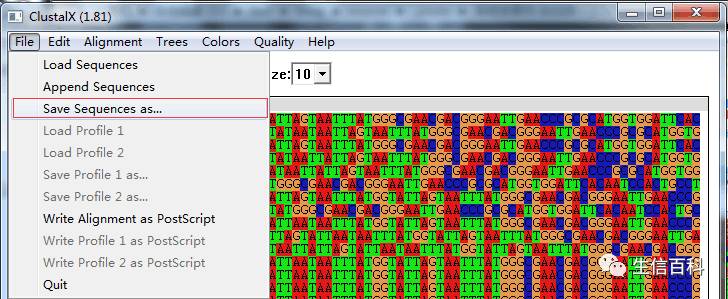
第三步
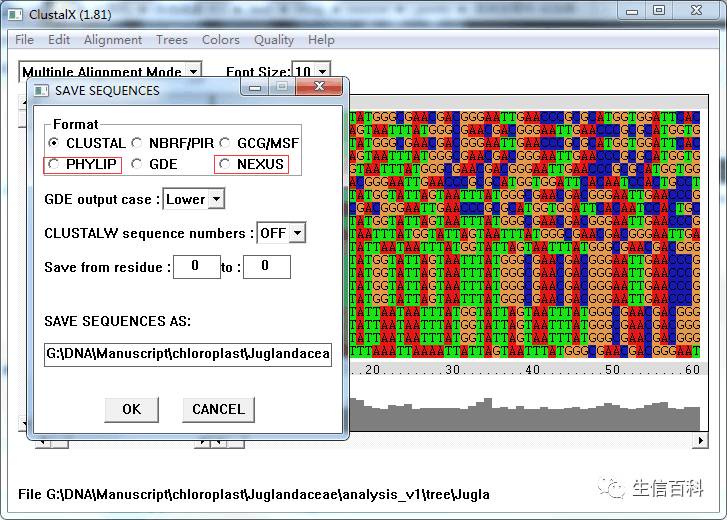
NEXUS用于paup+Modeltest进行模型选择,PHYLIP用于RAxML建树。
模型预测
模型预测的时候需要paup和modeltest两个软件,文件输入格式".nex"格式,在clustalx进行格式转换就可以了。
paup4a154_centos64
exetree_mafft_gb.nex
exe modelblockPAUPb10.nex
得到结果文件model.scores
Modeltest计算paup得到的结果
/path/to/Modeltest3.7.win.exe < model.scores > test.outfile
得到结果文件test.outfile(结果可以用Notepad++打开,或其他类似文本编辑器打开)
根据AIC标准,选择AIC值最小的模型,即为最佳模型。
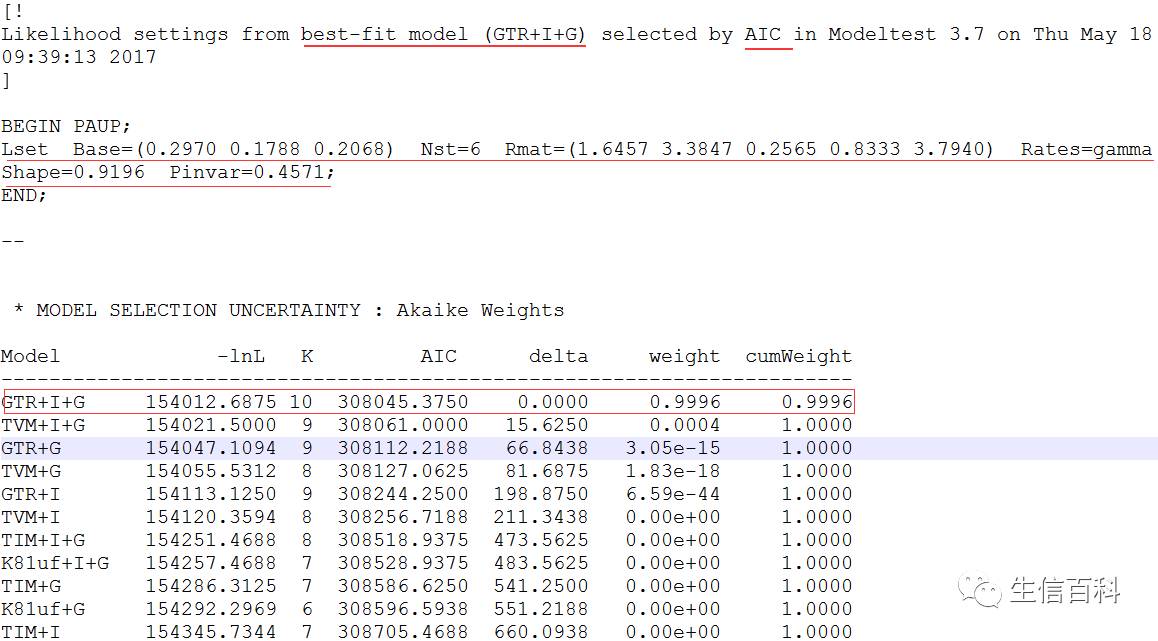
最大似然树
这里推荐使用RAxML进行最大似然法的系统发育树构建。
/path/to/RAxML_v8.1.24/usefulScripts/convertFasta2Phylip.sh tree_mafft.fasta-gb > tree_mafft_gb.phy
注意根据上一步预测的模型进行模型的设定
/path/to/raxmlHPC-PTHREADS-SSE3 -f a -x 12345 -p 12345 -# 100 -m GTRGAMMAI -s tree_mafft_gb.fasta -n test -T 24
带有Bootstrap结果的文件是:RAxML_bipartitions.test
画树
使用Figtree进行系统发育树的显示。需要配置Java运行环境
第一步:打开上一步得到带有Bootstrap的树文件RAxML_bipartitions.test
第二步:将lable1改为Bootstrap
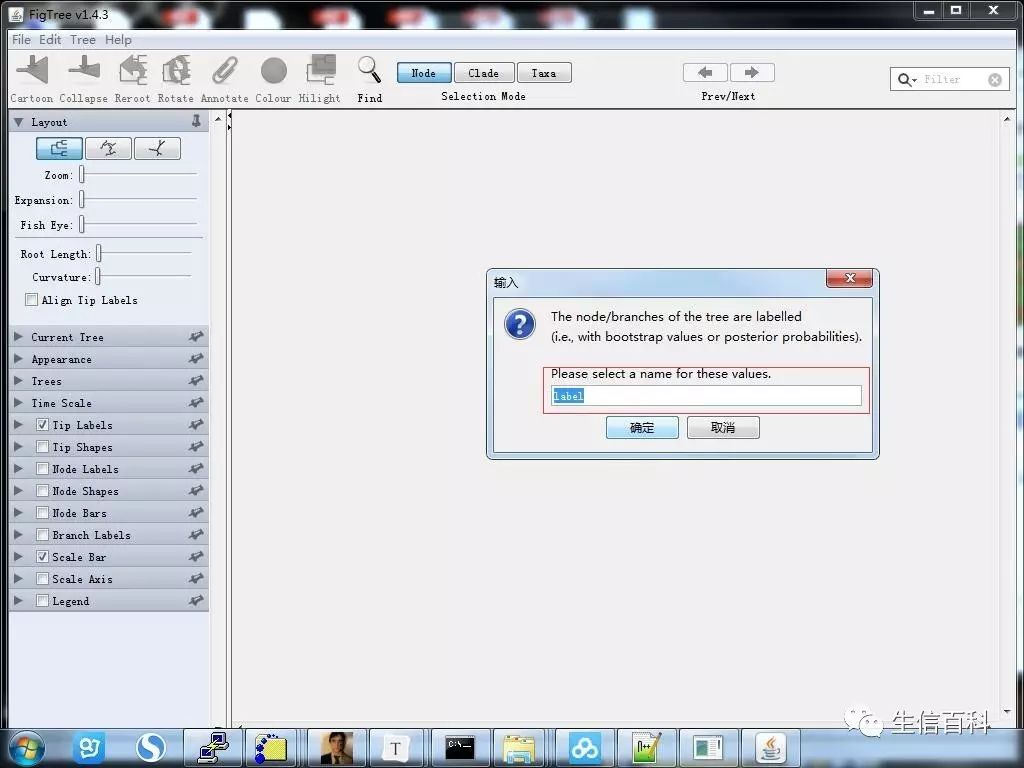
第三步:将外类群作为系统发育树的根(黄色箭头),图中两个红框区域为调节树形的操作,展现个人才艺的时候到了,你们尽情发挥。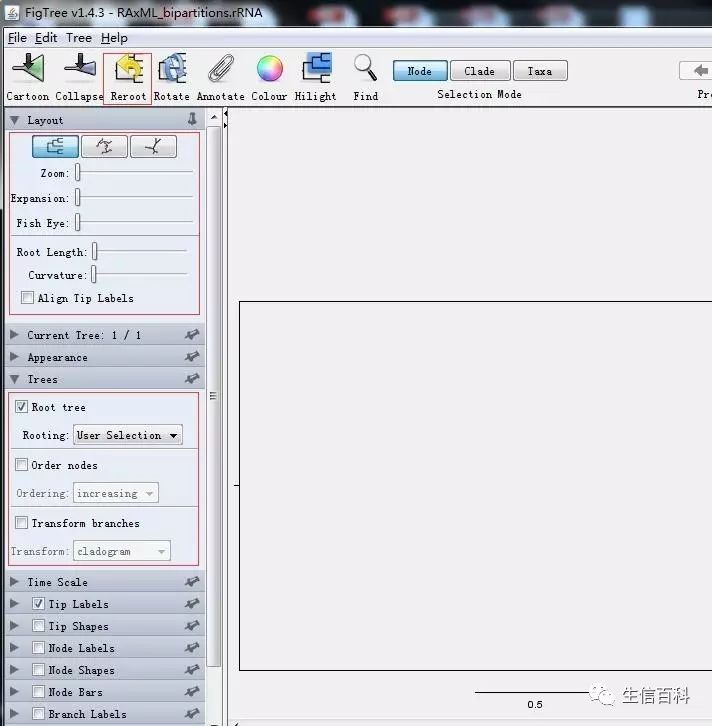
第四步:显示Bootstrap值
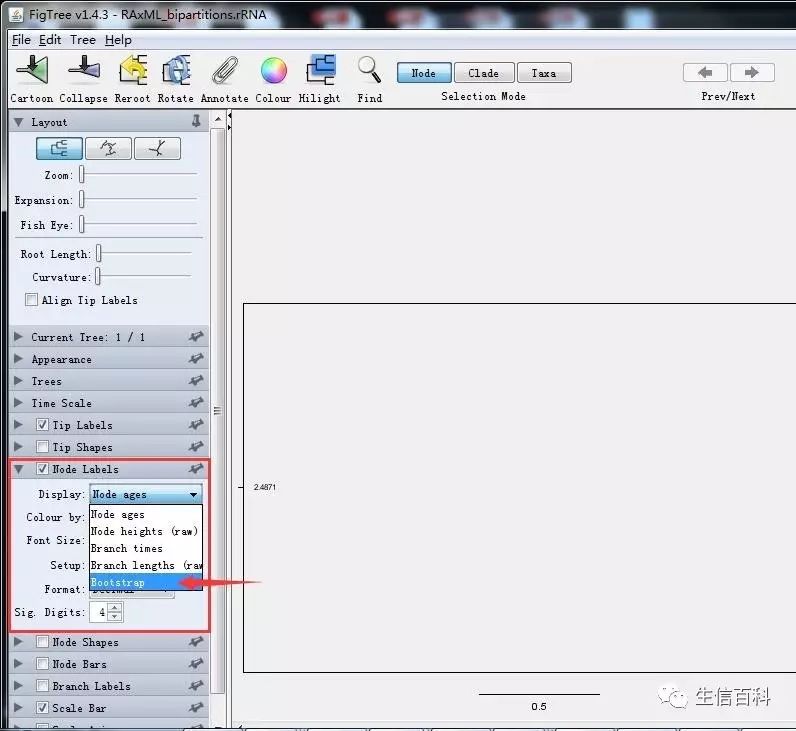
到这里一个基本的建树流程就这走完啦!希望可以帮助到在建树路上越走越远的你!
软件安装
paup的安装
无须编译,下载后解压并赋予可执行权限就可以使用。
wget http://people.sc.fsu.edu/~dswofford/paup_test/paup4a154_centos64.gz
gunzip paup4a154_centos64.gz
chmod 755 paup4a154_centos64
mafft安装参考:















