

进入主题前先跟大家分享两个关于单分子测序技术的小故事。
故事一
Illumina的技术来源于英国的Solexa公司,而Solexa的创始人Balasubramanian 和 Klenerman起家的技术是利用荧光标签核酸分子观测固定在表面的单个DNA分子被聚合酶合成的过程,但这信号实在是太弱了,俩人弄了几年都没成,后来想通了还是得利用扩增的手段把信号放大,在2004年买下了Manteia公司的分子团簇技术(molecular clustering technology),很快就发展出自身的桥式扩增技术,开启了横扫天下的模式。
故事二
现在火热的纳米孔测序技术其实早在上世纪80年代就有人提出来了,图1是纳米孔技术前辈David Deamer在1989年的一份珍藏手稿:利用电泳把一条DNA单链分子穿过一个纳米尺度的孔并记录其穿孔的电流信号从而推断出序列信息。很快David Deamer,Daniel Branton,和George Church等大神就写了专利,并指出这是个很有前途的方向,剩下的细节就交给大家去完成吧,接下来就是艰苦卓绝的30年,不断被二代技术弯道超车且不断跳票,到今天终现曙光,虽然仍有重重疑虑。

图1. 纳米孔测序方法原创手稿,来源1
纵观这两段历史,难免会有这样的疑问:
单分子测序技术这么大一个“坑
”,为什么大家伙还那么执着去做这件事情?
其实原因也很简单,因为第二代测序技术存在着难以解决的技术瓶颈。主要有:
1.读长短,
这是由于待测分子生化反应不同步所带来的“移相”问题,很多区域譬如长重复片段、平衡易位等就测不了;
2.需要扩增,带来了
GC偏好性等问题;
3.速度慢,每个碱基判定循环都需要冲
洗
换液拍
照
。
鉴于此,我们判断一个单分子测序技术是否可行,关键就要看这个技术是否能在这几个瓶颈有所突破,当然这得在保证足够准确率和通量,以及可接受的成本的基础上,类似有一些利用电镜做测序的技术基本就不用考虑了。
单分子测序技术最核心最困难的一点就是要有效提高单分子信号的信噪比,这一方面可以通过物理、化学和酶促等手段放大信号,另一方面就是降低背景噪音。
基本上我们会看到现在比较成功或者有希望成功的单分子测序技术在这点上都有其独到的解决方案。目前市场上已经商业化的单分子测序技术主要是美国Pacific Biosciences(PacBio)的Single-moleculeReal-time Sequencing技术以及英国Oxford Nanopore Technologies(ONT)的StrandSequencing技术。
基本原理如图2所示:单个聚合酶及文库DNA被固定在零模波导孔(ZMW)结构底部,带荧光标签的dNTP随机扩散到ZMW底部并与对应模板的碱基配对,同时在激光照射下该荧光标签分子产生荧光信号并被检测到,该碱基聚合反应结束后其磷酸上的荧光标签分子被切断并离开探测区域,接下来下一个dNTP继续合成反应,实现没有中断的实时测序。
这里面两个关键的技术点:
★ZMW有效的限制了激光的激发区域仅仅在ZMW底部以上几十nm的空间,大大降低了游离态的荧光标签分子所带来的背景噪音;
★荧光标签结合在磷酸根上,使得连续合成得以实现,而且能有效地保持聚合酶的活性并大大延长读长。
当然除了这两点,还有很多很重要的技术突破,譬如环状循环模板测序,将光波导和影像传感器集成到测序芯片上等等,才有了今天Sequel这款出色的单分子测序机器。
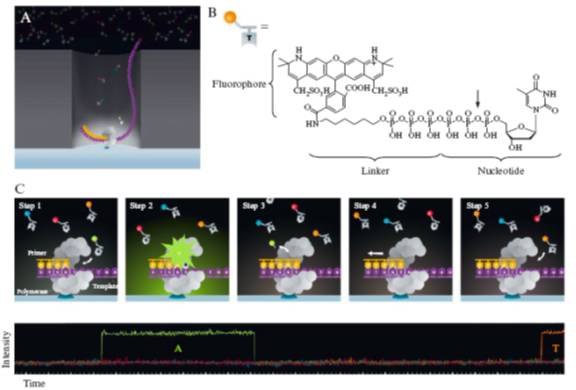
图2. PacBio的单分子实时测序方法原理介绍,来源2
基本原理更加简单,如图3所示:在电场力作用下,溶液中的带电离子可以穿越蛋白质纳米孔并产生离子电流信号,待测DNA单链分子加入溶液后会在电泳作用下顺次穿孔
并阻断部分离子的穿越而造成离子电流突变减小。不同的碱基会带来不同的离子阻断电流,从而可以根据离子阻断电流大小来识别碱基。
这里面有两个关键的技术点:
★找到最小内径只允许DNA单链通过的蛋白质纳米孔,且其最敏感区域所
容纳
的碱基越少越好
(理想情况只容纳1个碱基),
这样的纳米孔才能有最大的信号区分度,
事实上,很难找到只容纳一个碱基的超薄纳米孔,通常在最敏感区域内是一段碱基序列,但根据离子阻断电流大小的差异同样可以推断出不同的序列组合。
目前Illumina的MspA(4个碱基)和ONT的CsgG(4-6个碱基,不确定)是最优代表;
★利用了机动蛋白(聚合酶或者解旋酶)在单碱基精度上有效控制DNA分子的穿孔过程,
很大程度上解决了穿
孔速度过快所导致的信号分辨率问题。
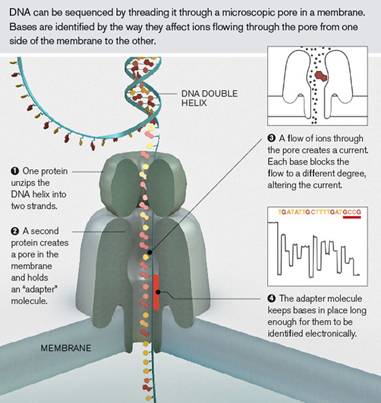
图3. 纳米孔Strand Sequencing原理,来源3
表格1是PacBio的Sequel和ONT的一系列产品的参数比较.需要指出的是,ONT的指标一般都是最优指标,需要有更多的使用者共享数据,所以目前仅供参考。

PacBio有2个比较难解决的问题:
1.
单次准确率较低
:错误率主要来自于生化反应,包括聚合酶的错误,非配对碱基产生信号,荧光标签失效等等,这么多年的改进中这个基本没有大的变化;虽然这种错误是随机的,因此PacBio可以用环状模板循环测序以达到高共有准确率(consensus accuracy),但必须指出这牺牲了读长的优势,因为聚合酶结合在模板上面的总时间是有限的,太长的环状模板对提高覆盖度无用。
2
.价格昂贵
:单分子光学检测所需要的设备便宜不了,而把光学部分集成在芯片上面的话芯片本身的成本又上去了。
说到这里顺带提提Helicos公司,它的tSMS(truesingle-molecule sequencing)技术首次实现了单分子测序,并在2009年用于测出其创始人Stephen Quake的个人全基因组。当初是很受追捧的一家公司,但回过头来看看,该技术作为一种短读长非实时的单分子测序技术,和二代技术比拼准确率、通量和成本是
,应该
很难成功的。继承了其技术
衣钵
的瀚海基因能否在短读长靶向测序方向占得一席之地并不明朗。
ONT技术面临的最大困难仍然是
准确率较低
,主要体现在三个方面:
1.虽然电场力和机动蛋白在一定程度上控制了DNA分子在纳米孔内的随机热运动,但这里面仍然存在极限;
2.此外任何依赖于酶的读取过程都会受到酶本身错误率的影响;
3.最后就是如何解决homopolymer长链的测序,
非官方渠道沟通已经能轻松超越10mer
,有待验证
。
尽管如此,ONT这几年来在准确率方面的飞速提升十分惊人,目前单次准确率和两次准确率已经对外宣称达到92%和96%,这
一方面来自于蛋白改造的进步,另一方面就是大量数据加上深度学习所带来的算法优化
。此外再考虑到
ONT的设备如此低廉,而且其测序芯片的价格还有空间下降,拥有便携式等特有优势,
PacBio的
前途蒙上了阴影。
|
指标
|
Sequel
(PacBio)
|
MinION
(ONT)
|
GridION
(ONT)
|
PromethION
(ONT)
|
|
样品制备
|
时间
|
6hr
|
10min – 1hr
|
10min – 1hr
|
/
|
|
进样量
|
10ng - 10ug
|
10ng – 10ug
|
/
|
/
|
|
准确率
|
单次精度
|
86%
|
92%
|
92%
|
/
|
|
共有精度
|
99.99%@30X
99.999%@60X
|
99.5% @30X
4
|





