文章介绍了Helicobacter pylori Genome Project(HpGP)的研究,揭示了全球范围内H. pylori种群的详细结构。通过对来自50个国家的1011个临床菌株的基因组测序和分析,研究发现了H. pylori的子种群与宿主地理来源和胃病风险密切相关。文章通过fineSTRUCTURE、染色体绘图、共享核心基因特征的网络分析以及主成分判别分析(DAPC)等方法,对HpGP数据集的种群结构进行了深入探究,并揭示了不同地理区域如欧亚、非洲、北美的H. pylori种群的祖先贡献。此外,文章还重点关注了中亚、非洲和拉丁美洲的H. pylori基因组特点,并介绍了北美的克隆种群和美洲土著H. pylori的祖先贡献。
文章介绍了HpGP的背景、目的和方法,包括收集来自全球的临床菌株、生成高质量基因组序列、分析核心基因组多样性和种群结构等。
文章通过数据分析揭示了不同地理区域如欧亚、非洲、北美的H. pylori种群的祖先贡献和分化情况。
文章通过fineSTRUCTURE分析、染色体绘图和网络分析等方法,对中亚地区的H. pylori基因组特点进行了深入研究。
文章分析了非洲和非洲后裔的H. pylori基因组的遗传背景和混合情况,并探讨了与欧洲和其他地区的差异。
文章对拉丁美洲的H. pylori种群进行了深入研究,发现其遗传背景比欧洲和非洲基因组更加混合。
文章通过数据分析揭示了北美存在的深克隆种群的特点和演化历史。
❝The Helicobacter pylori Genome Project: insights into H. pylori population structure from analysis of a worldwide collection of complete genomes
❞
摘要
幽门螺旋杆菌(Helicobacter pylori)[^2]是胃部微生物群的重要成员,且与人类共同进化,形成了不同的基因群体。研究表明,H. pylori的子种群与宿主的地理来源和不同的胃病风险密切相关。该研究为幽门螺旋杆菌(Helicobacter pylori)基因组项目(HpGP)的一部分,旨在揭示H. pylori的致病机制并寻找新的治疗靶点。通过收集来自「50个国家的1011个临床菌株」并生成高质量的基因组序列,分析了核心基因组多样性和种群结构。
- 通过对HpGP数据集和255个全球参考基因组的分析,研究揭示了不同地理区域(如欧亚、非洲、美国)的H. pylori种群的祖先贡献。
- 北欧H. pylori菌株中,来自北欧地区的hpNorthAsia种群和HspUral亚种群有显著贡献。
- 北美和南美的原住民H. pylori菌株有所不同,「北美的菌株与北亚的H. pylori更相似」,而「南美的菌株则与东亚的菌株有更高的相似性」。
- 在北美,存在一个高度克隆化但地理分布广泛的子种群,且其「缺失了cag致病岛」,这一亚群在7%的美国基因组中被检测到[^1]。研究的结果提供了幽门螺旋杆菌(Helicobacter pylori)种群的详细结构,并有助于进一步的基因组学研究。HpGP数据集和相应的菌株预计将成为H. pylori基因组学研究的重要资源。
介绍
研究背景
幽门螺旋杆菌(Helicobacter pylori)与人类存在漫长的共进化过程,其地理分布十分广泛,分为多个亚群[^3] [^4] [^5]。
科学问题
然而,之前的研究未对其整个基因组区域进行测序,使得对H. pylori 种群结构的理解存在局限。H. pylori感染导致的「疾病风险在不同地区差异显著」,因此需要通过对人类和H. pylori 基因组的研究,识别影响这一风险的因素。
HpGP是一个国际多学科合作项目,旨在通过收集全球范围内的H. pylori菌株,进行基因组测序,并绘制H. pylori 的种群结构图。通过将HpGP数据集与已知的种群分配参考数据进行比对,研究能够「高精度地量化H. pylori 亚种群的祖先来源,并探讨亚种群之间的近期和持续的基因流动」。
数据集特点
HpGP数据集包含了来自50个国家的临床菌株,其中包括12个此前没有发布过H. pylori 基因组序列的国家。在1011个基因组中,除了7个外,所有基因组都已经完全环状化。为研究 HpGP 数据集的种群结构,采用了 fineSTRUCTURE(FS)、染色体绘图、共享核心基因特征的网络分析以及主成分判别分析(DAPC)等方法。数据集中的分析还包括了255个已知种群分配的参考基因组,代表了17个全球亚种群。HpGP 数据集的核心基因组由1227个基因组成,涵盖了>95%的基因组序列。包括 HpGP26695参考基因组和255个全球参考基因组。通过fineSTRUCTURE全球分析,研究发现H. pylori存在四个主要的种群簇:
- 「北亚、中亚和东亚以及美洲土著群体」这四个簇包含了17个主要的亚种群。
网络分析和 DAPC 分析也支持上述的种群结构,但显示出六个主要的分化簇。「南非」(DAPC 组4,fineSTRUCTURE 中的 hpAfrica2)和来自「澳大利亚/新几内亚」的参考基因组(DAPC 组6,fineSTRUCTURE 中的「hpSahul」)与其他种群存在明显的差异。分析表明,剩余的群体中有两个群体明显分化:
- 由非洲和美洲来源的菌株组成(DAPC组2,fineSTRUCTURE簇III)
- 包含中亚/东亚及美洲土著菌株的群体(DAPC 组5,fineSTRUCTURE 簇 IV) 剩下的群体(组1和组3)较为相似,分别代表了南欧/东北非和欧亚/中亚及美洲。
hpEurope 亚群横跨大西洋沿岸至南亚
在 fineSTRUCTURE 分析中,研究发现了三大主要的欧洲/欧亚子种群:
- 「hspEurasia」(欧亚地区) 研究提出了 hspEurasia 子种群,其中包括之前描述过的 hspCEurope、hspSEurope 和 hspMiddleEast 子种群。
- 「hspNEurope的东-西分化」:在「hspNEurope」中,存在东西分化的趋势,「拉脱维亚」、「立陶宛」和「俄罗斯」的菌株形成东部分支,与来自「英国」、「瑞典」、「冰岛」和「加拿大」的菌株分布在西部。
- hspEurasia1:德国、波兰、立陶宛、拉脱维亚、土耳其和俄罗斯。
- hspEurasia2:更加南方,涵盖法国、意大利、希腊、约旦和伊朗等地。
- hspEurasia3:包括印度、孟加拉国的菌株,也包括希腊的菌株,虽然有所分离,但仍属于 hspEurasia 子种群。
- 通过对比当前数据集中的基因组,研究推断了不同子种群的祖先成分。祖先的来源包括 hpAfrica2、hpNEAfrica、hspAfrica1WAfrica、hpAsia2、hspUral、hpNorthAsia 和 hspEAsia 等。
- 沿南北轴线,hspEurasia1和 hspNEurope 种群中,亚洲祖先成分逐渐增多,而非洲祖先成分则逐渐减少。
- hspSWEurope 有较高比例的 hspAfrica1WAfrica 祖先成分,而 hspEurasia2与 hspSWEurope 相比,来自 hpNEAfrica 的贡献相似。
- hspUral 作为亚洲祖先,对所有 hpEurope 子种群的贡献显著高于 hpNorthAsia 和 hspEAsia,后两者贡献较为均衡,除了在 hspNEurope 中,hpNorthAsia 的贡献略高。
- 这些遗传关系通过网络分析得到了进一步的支持,进一步验证了各子种群之间的分化和基因流动模式。
 图1| HpGP 菌株起源和种群分配的世界地图。
图1| HpGP 菌株起源和种群分配的世界地图。
中亚地区代表性仍然不足
fineSTRUCTURE分析将中亚的菌株分为三大类群:
- 「hpAsia2」:作为H. pylori的主要祖先种群之一,尽管已有研究,但相对缺乏足够的关注。
- 「hpNorthAsia」:一个主要的西伯利亚种群,早期通过多位点序列分型(MLST)确立。
- 「hspUral」:被认为是hpAsia2的一个南部中亚子种群。hpAsia2的分布和研究:
「hpAsia2的分布」:HpGP数据集中,hpAsia2的基因组主要来自「印度」、「孟加拉国」、「缅甸」和「尼泊尔」。其中,「尼泊尔」的菌株与其他地区的菌株有所分离。
「与hpEurope hspEurasia3的共存」:在除「缅甸」外的所有这些国家,hpAsia2与hpEurope的hspEurasia3种群共存,而在「缅甸」仅存在hpAsia2菌株。
「DAPC分析」:使用k=6时,hpAsia2与hspNEurope和hspEurasia没有明显区分。
「hpNorthAsia」:作为一个西伯利亚主要种群,hpNorthAsia及其子种群「hspAltai」和「hspSiberia1」,主要分布在中东和东部的西伯利亚。在HpGP数据集中,「哈萨克斯坦」和「吉尔吉斯斯坦」的基因组也与这一群体相关联。
「hspUral的分布」:hspUral被认为是hpAsia2的一个南部中亚子种群。该群体在「哈萨克斯坦」、「吉尔吉斯斯坦」、「印度尼西亚」和「日本」等地有较广泛的地理分布。
「染色体绘图分析」:主染色体绘图分析显示,hspUral群体内部有两个子分支,其染色体绘图特征差异明显,这一结果也得到了DAPC和网络分析的支持。
在中亚基因组的祖先贡献分析中,hspUral群体没有明显的hspUral参考基因组贡献,但却显示出较高的「hpAsia2」、「hpNorthAsia」和「hspEAsia」的贡献。群体内的遗传变异较大,表明该群体可能并不是一个纯粹的子种群,而是由多个HpAsia子种群的代表组成。
尽管对于中亚种群的研究分辨率已大幅提高,但仍然存在一些区域未被充分代表。「hspUral群体显示出遗传结构的复杂性,可能代表了多个HpAsia子种群的混合」,这需要进一步的研究以更清晰地揭示其遗传演化和地理分布关系。
 图2|对所研究的幽门螺杆菌菌株核心基因组进行距离网络分析。
图2|对所研究的幽门螺杆菌菌株核心基因组进行距离网络分析。
非洲和非洲人后裔的 hp基因组
- 「HpGP数据集的非洲基因组」:该数据集包括来自较少研究的非洲国家的基因组,如「阿尔及利亚」、「刚果民主共和国(DRC)」、「加纳」和「尼日利亚」,以及来自「冈比亚」和「南非」的已知基因组。
- 通过fineSTRUCTURE分析,确认了非洲大陆上四个主要的H. pylori种群。
- hspAfrica1SAfrica:分布至「刚果」民主共和国。
- hspAfrica1WAfrica:代表「冈比亚」,此前已有研究描述。
- 这些基因组与更多混合的 hspAfrica1NorthAmerica 和 hspAfrica1MiscAmericas 种群相似,并且通过染色体绘图与来自美国、波多黎各(美国领土)、多米尼加共和国、哥伦比亚和巴西的基因组无法区分。这一现象很可能与「西非的跨大西洋奴隶贸易有关」,表明这些基因组的混合起源。
东非基因组的分群:来自苏丹和埃塞俄比亚的东非参考基因组与 hspSWEurope 的欧洲群体相关,但与欧洲 SWEurope 群体不同,形成了一个与北美基因组相似的簇。PCA 分析:在 fineSTRUCTURE 的 PCA 图中,hpNEAfrica 和美国 HpGP 基因组明显分为两组,尤其在 PC10和 PC11上,除了一个马来西亚基因组和一个瑞士基因组外,它们与其他基因组群体明显区分(可以在互动图中查看)。DAPC 和网络分析:「这两种分析都支持 hpAfrica1与 hpNEAfrica 的分离」,后者与来自南欧和伊比利亚的基因组相互交织。阿尔及利亚的基因组:「阿尔及利亚的基因组没有与其他非洲基因组聚集」,而是与来自以色列和哥伦比亚的基因组一起,形成了一个我们称之为 SWEurope2的簇。遗传成分分析:尽管SWEurope2的基因组显示出略高的西非和东北非祖先成分,以及较低的亚洲成分,但分析确认,「北非的H. pylori与伊比利亚和中东的细菌更为相似,而不是与非洲其他地区的H. pylori 相似」。 图3|推断祖先基因组对欧亚 HpGP 基因组的贡献。
图3|推断祖先基因组对欧亚 HpGP 基因组的贡献。
北美的克隆种群
- 「基因组数据」:HpGP数据集包含来自美国大陆的68个基因组,这些基因组的广泛地理代表性使得研究人员能够识别出一个新的子种群:「15个美国菌株」,它们在「fineSTRUCTURE分析」中与「hpNEAfrica」祖先的基因组高度相似,并且「没有携带cag致病岛(cagPAI)」。
- 通常,H. pylori的菌株在基因序列上差异较大,而这一新发现的美国菌株几乎所有基因都有序列差异。
- 在 HpGP 数据集中,超过64%的菌株对比显示在1040个基因上存在「完全的序列差异」,即使是「同一国家的菌株也有34%的基因存在完全差异」。仅有0.15%的菌株对显示出超过1%的基因相似性。
- 在这些相似的菌株中,有66对菌株来自12个美国菌株,基因相似度在0.83到0.94之间,「表现为“深克隆”关系」。这些菌株在「同一祖先下进化」,但通过同源重组(homologous recombination, HR)在基因组的大部分区域发生了分化。
- 这些深克隆群体的菌株采自美国的多个州,包括加利福尼亚、威斯康星、田纳西、阿肯色、乔治亚和德克萨斯,这些菌株共占美国 HpGP 基因组的五分之一。
- 通过 Kmer 聚类分析,发现来自俄亥俄和路易斯安那的另外五个公共基因组与深克隆群体密切相关。
- 使用「ClonalFrameML 工具估算基因组」之间的关系,假设「每年每个位点的突变率为1.38×10^–5」,估计该「群体的共同祖先大约在175年前存在」(95%的置信区间为107–227年),而大多数「内部节点的估算时间不到50年」。
 图4|深入分析全球幽门螺杆菌数据集中的克隆关系。
图4|深入分析全球幽门螺杆菌数据集中的克隆关系。
拉丁美洲的种群
大约三分之一的拉丁美洲基因组与非拉丁美洲种群相似,主要是 hspAfrica1SAfrica 和 hspSWEurope,还有一些 hspEAsia 和 hspEurasia 基因组分别来自阿根廷、巴西和智利。这些发现表明,拉丁美洲基因组的遗传背景比欧洲和非洲基因组更加混合。大多数智利菌株聚集在一个独立的群体 hspSWEuropeChile 中,类似于先前描述的哥伦比亚菌株群体 hspSWEuropeColombia。这些菌株与 hspSWEurope Latin America 和 hspSWEurope 有较近的关系,特别是在 fineSTRUCTURE PCA 中的 PC1和 PC7成分中表现突出。智利菌株的聚类和自绘图特征:在 DAPC 和网络分析中,这些菌株虽然分散,但仍靠近 hspSWEurope 群体,并且在染色体绘图分析中显示出很高的自绘图比例,支持它们与 hspSWEurope 群体的关系。美洲土著H. pylori 的祖先贡献:hspIndigenousAmerica 群体:fineSTRUCTURE 分析确认了 hspIndigenousAmerica 群体。该群体包括来自混合人类血统的城市地区和土著社区的菌株。hspIndigenousAmerica 可以细分为两个子群体:
- hspIndigenousNAmerica:由「北美土著社区(美国和加拿大)的菌株组成」。
- hspIndigenousSAmerica:主要包含来自「拉丁美洲地区的菌株,尤其是在智利、墨西哥、秘鲁、西班牙和美国的观察结果」。根据祖先染色体绘图和网络分析,hspIndigenousSAmerica 与 hspEAsia 有较高的相似性,而 hspIndigenousNAmerica 则表现出较高的土著祖先成分,并与 hpNorthAsia 更为接近,在网络分析中与 hspIndigenousSAmerica 存在一定距离。
 图5 |种群分类摘要。
图5 |种群分类摘要。
尾注
[^2]: 简称为H. pylori。
[^3]: Falush, D. et al. Traces of human migrations in Helicobacter pylori populations. Science 299, 1582–1585 (2003).
[^4]: Linz, B. et al. An African origin for the intimate association between humans and Helicobacter pylori. Nature 445, 915–918 (2007).
[^5]: Moodley, Y. et al. Age of the association between Helicobacter pylori and man. PLoS Pathog. 8, e1002693 (2012).
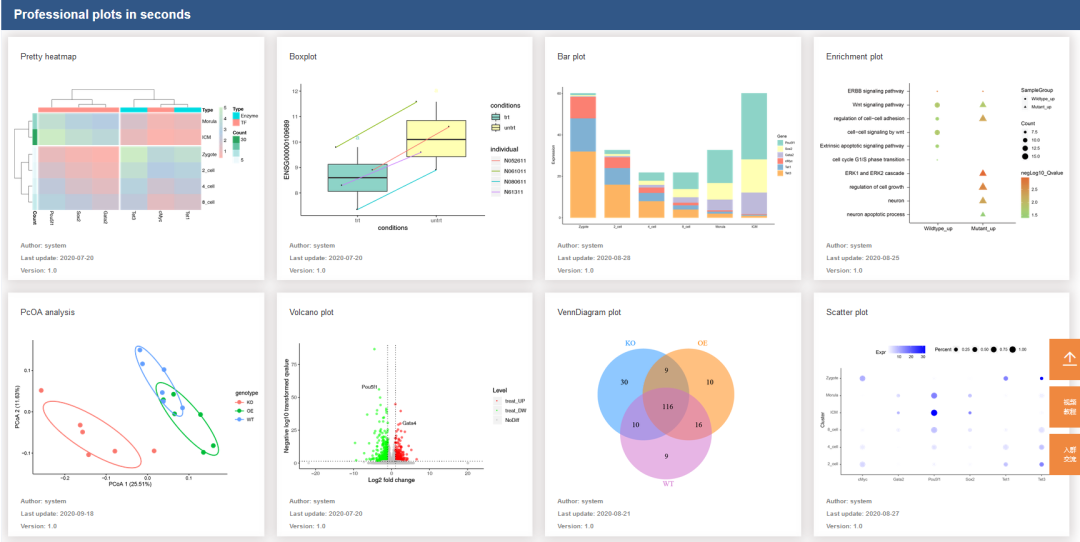
高颜值免费 SCI 在线绘图(点击图片直达)
最全植物基因组数据库IMP (点击图片直达)
往期精品(点击图片直达文字对应教程)
机器学习