
理想条件下,我们希望一个物种有多少染色体,结果最好就只有多少个contig。当然对于二代测序而言,这绝对属于妄想,只希望得到的contig文件中,每个contig都能足够的长,能够有一个完整的基因结构,归纳一下就是3C原则:
-
连续性(Contiguity): 得到的contig要足够的长
-
正确性(Correctness): 组装的contig错误率要低
-
完整性(Completeness):尽可能包含整个原始序列
这三条原则也比较定性,我们需要更加定量的数值衡量,比如说contig数, 组装的总长度等, N50等。问题来了,什么叫做N50呢,
小故事,当初我刚学生信的时候,老板给我一个项目,让我继续组装一个初步组装的contigs。我刚入门啥都不懂,于是就去请教一个师兄,他当时问我你的基因组N50是多少呀?我一脸懵逼,茫然四顾,后来他又问了我几个问题,给了我几个小建议,这些我都已经忘记了,唯独记得N50。后来,老板让我单独去请教帮我们初步组装的那个人,当然他还找了一个会议记录,我还是啥都不懂,场面很尴尬,我最后做的事情就是把原始数据拷到硬盘里,那个数据拷贝后,我就没有碰过。再后来,还是这个物种,老板带着我和师姐一起去找他们讨论。这下稍微好一点,当然不是因为我懂得多了,是因为老板和他们聊八卦比较开心。那么多年过去了,很多人和事都已经随风而去,但是N50却一致挥之不去。
N50定义比较绕口,有一种只可意会不可言传的感觉,所以索性看图
 N50和NG50
N50和NG50
假设一个基因组的大小为10,但是这个值只有神知道,你得到的信息就是组装后有3个contig,长度分别为"3,4,1,1",所以组装总长度为9。为了计算N50,我们需要先把contig从大到小排列,也就是"4,3,1"。然后先看最大的contig,长度是4,他的长度是不是超过组装总大小的一半了吗?如果是,那么N50=4, 4 < 4.5, 不是。 那么在此基础上加上第二长的contig,也就是4+3=7, 是不是超过一半了?7>4.5, 那么N50=3. 因此,N50的定义可以表述为"使得累加后长度超过组装总长度一半的contig的长度就是N50"。为了方便管理和使用软件,建议建立如下几个文件夹
N50是基于一个未知的基因组得到得结果,如果基因组测序比较完整,那么就可以计算NG50,也就是"使得累加后长度超过基因组总长度一半的contig的长度就是NG50"。NA50比较稍微复杂,需要将组装结果进一步比对到参考基因组上,以contig实际和基因组匹配的长度进行排序计算。
说完N50,我们介绍两款工具,QUAST和BUSCO。
QUAST
使用质量标准(quality metrics)来评估不同组装工具和不同参数的组装效果,无论是否有基因组都可以使用。我们分别以有参和无参两种模式比较
Minia
,
IDBA
和
SPAdes
三个组装的运行结果
# without reference
quast.py -o compare idba_ud/contig.fa minia/minia.contigs.fa spades/contigs.fasta
# with reference
quast.py -R ../genome/Saureus.fna -o compare idba_ud/contig.fa minia/minia.contigs.fa spades/contigs.fasta
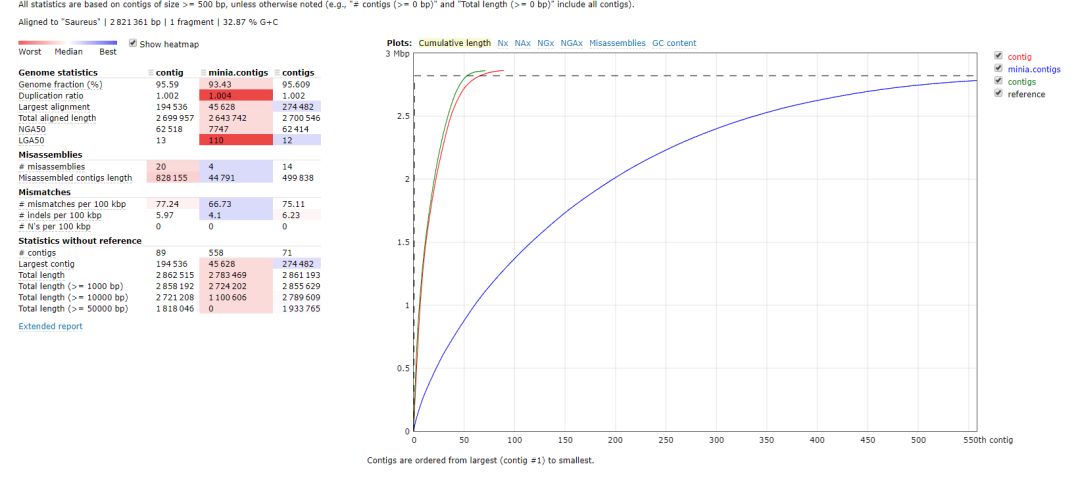 quast运行结果
quast运行结果
这个结果非常直观的告诉我们一个事实就是
spades
组装的contigs`各方面表现都很优秀,minia由于内存使用率最低,所以组装效果一般也是可以理解。
BUSCO
通过同源基因数据库从基因完整度来评价基因组组装结果。BUSCO首先构建了不同物种的最小基因集,然后使用HMMER,BLAST,Augustus等工具分析组装结果中的同源基因,从而定量评估组装是否完整。
busco -i assembly/spades/contigs.fasta -o result -l /home/wangjw/db/busco/bacteria_odb9 -m genome -f
运行结果会在当前目录下的
run_result
生成一些列文件,其中的
short_summary_result
.
txt
内容如下
# Summarized benchmarking in BUSCO notation for file assembly/spades/contigs.fasta
# BUSCO was run in mode: genome
C:98.6%[S:98.6%,D:0.0%],F:0.0%,M:1.4%,n:148
146 Complete BUSCOs (C)
146 Complete and single-copy BUSCOs (S)
0 Complete and duplicated BUSCOs (D)
0 Fragmented BUSCOs (F)
2 Missing BUSCOs (M)
C值表示和BUSCO集相比的完整度,M值表示可能缺少的基因数,D则是重复数。正所谓没有比较,就没有伤害,我们拿之前QUAST对比中表现比较差的minia结果作为对比。
C:85.1%[S:85.1%,D:0.0%],F:2.7%,M:12.2%,n:148
126 Complete BUSCOs (C)
126 Complete and single-copy BUSCOs (S)
0 Complete and duplicated BUSCOs (D)
4 Fragmented BUSCOs (F)
18 Missing BUSCOs (M)
这样子就一目了然了,说明spades不但组装效果号,而且基因完整度也高,从两个维度上证明它的组装
到这里位置,组装的核心部分基本上就结束了,关于contig的进一步搭建scaffold,10X genomics和Chicago技术是如何让二代测序高大上,三代测序如何辅助二代,光学图谱是怎么一回事,这些等到老板愿意花钱让我去测再说。
基因组组装,简单也复杂
到底要不要组装基因组,没有调查就没有发言权
基因组组装之环境准备
顺便说一句,组装这个领域我也是刚进入,所以知识更新比较快,欢迎加入我的知识星球和我讨论






