上期为大家剖析了基因组组装的基本原理,本期将从实战操作的角度带大家逐步实践宏基因组组装的具体步骤。宏基因组组装一般首先逐个对样本进行组装,再将所有样本中未参与组装的reads混合起来进行组装。

1. 配置config文件
根据质控后的fastq文件(sample1.fq1,sample1.fq2,sample2.fq1.sample2.fq2,sample3.fq1,sample3.fq2)配置每个样本的组装输入文件sample1.config,sample2.config,sample3.config;
Config文件内容如下:
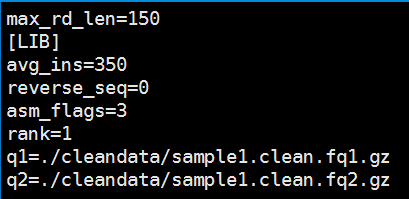
max_rd_len
表示read的最大长度;
[LIB]
表示文库信息标签;
avg_ins
文库中插入片段平均长度
reverse_seq
序列是否需要被反转,0(不反转),1(反转),一般插入片段大于等于2k文库,在建库是会将插入片段进行环化,此时须设置该参数为1;
asm_flags
表示reads用于组装哪个部分,可设为1,2,3, 1表示reads仅用于contig组装,2表示reads仅用于scaffold组装,3表示reads同时用于contig和scaffold组装;
rank
构建scaffold时,不同文库中reads的使用顺序,文库中reads序列越短,级别越高;
q1,q2
用于组装的双端fq文件。
2. 软件
安装SOAPdenovo,下载地址 http://sourceforge.net/projects/soapdenovo2/files/SOAPdenovo2/ ;
安装SOAPaligner,下载地址为http://soap.genomics.org.cn/soapaligner.html
1. 对每个样本单独组装
组装命令:
![]() SOAPdenovo-63mer all –s sample1.config –K 57 –o sample1 -d 1 -M 3 -u –F -p 10
SOAPdenovo-63mer all –s sample1.config –K 57 –o sample1 -d 1 -M 3 -u –F -p 10
SOAPdenovo-63mer all –s sample2.config –K 57 –o sample2 -d 1 -M 3 -u –F -p 10
SOAPdenovo-63mer all –s sample3.config –K 57 –o sample3 -d 1 -M 3 -u –F -p 10
参数说明:
-s config配置文件;
-K k-mer的长度;
-o 输出文件前缀;
-d [INT], kmerFreqCutoff, 去除频数小于等于该值的kmers,默认为0;
-M [INT], mergeLevel连接contigs时, 合并相似序列的等级,默认为1,最小值为0,最大值为3,
-u 构建scaffold前屏蔽过高或过低覆盖度contigs,默认屏蔽;
-F 利用reads对scaffolds的gap进行填补,默认不执行;
-p 需要使用的cpu数目,默认8;
输出结果文件:
Sample1.scafSeq:scaffold的fasta序列文件;
Sample1.scafStatistics: scaffold和conitg的统计文件,分布统计了scaffold和contig的数目,序列的一些统计信息,GC含量,N50及N90值等;
2. 通过re-mapping获取每个样本未参与组装的reads
比对命令:
2bwt-builder sample1.scafSeq #对scafSeq构建索引;
Soap -p 6 -r 2 -m 200 -x 400 -a ./cleandata/sample1.clean.fq1.gz -b ./sample1/sample1.clean.fq2.gz -D sample1.scafSeq.index -o sample1_PE.soap -2 sample1_SE.soap -u sample1_UN.fasta
参数说明:















