
10分的文章可能很多童鞋想都没想过,今天本宫就从ceRNA机制的角度解析一下如何从无到有,设计一篇10分文章的研究思路。
ceRNA机制想必大家已经比较熟悉,先前的微信文章有不少介绍,这里不再赘述,环状RNA作为ceRNA研究套路总结,ceRNA综述、数据库及经典文章。生信作为实验的辅助工具是非常高效的,特别是在肿瘤研究中,大量的芯片以及测序数据可供使用,重复测序显得意义不大。生信+实验双管齐下可以事半功倍地进行科学研究。

lncRNA-miRNA-mRNA这样的ceRNA机制研究是最为常见的,它由miRNA-mRNA和lncRNA-miRNA两部分构成。数据库工具可以帮助我们在茫茫基因海中进行初步筛选,miRNA预测靶基因的数据库工具我们先前已经介绍了很多我好像发现了一个miRNA各种软件的大本营……,lncRNA-miRNA的预测我们介绍过DIANA这一工具如果miRNA神器只能推荐一个,就是这个了!,今天再给大家介绍一个新的——lncRNASNP2(http://bioinfo.life.hust.edu.cn/lncRNASNP2#!/),这个数据库不仅可以预测lncRNA-miRNA的关系,还包含了人类lncRNA的SNP位点信息结合lncRNA/microRNA和SNP的研究,有哪些数据库可以用?。
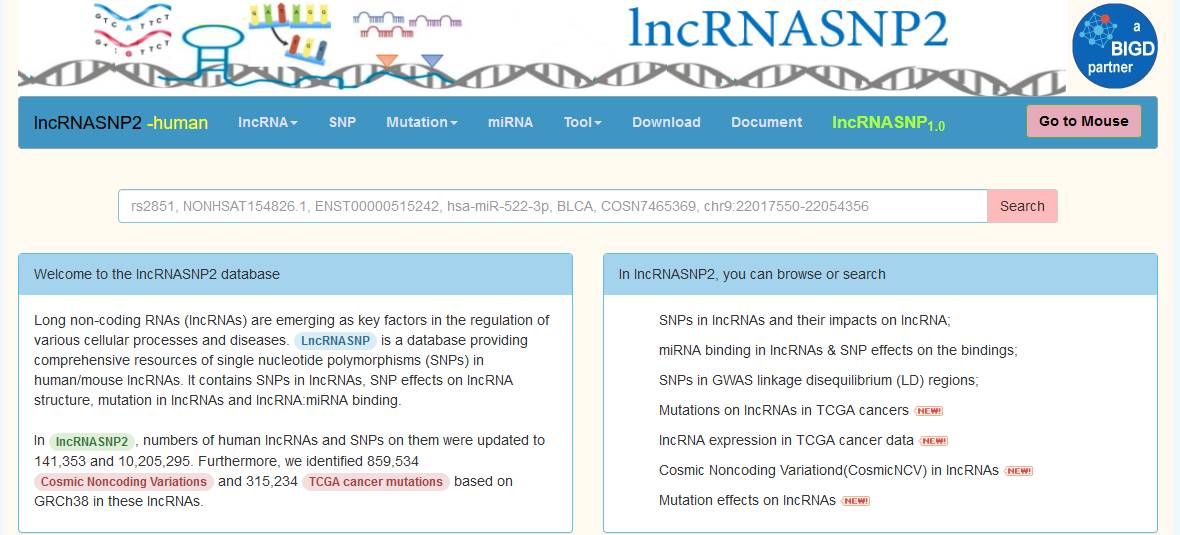
在Tool的Search中我们可以查找lncRNA&SNP&miRNA的相关信息:

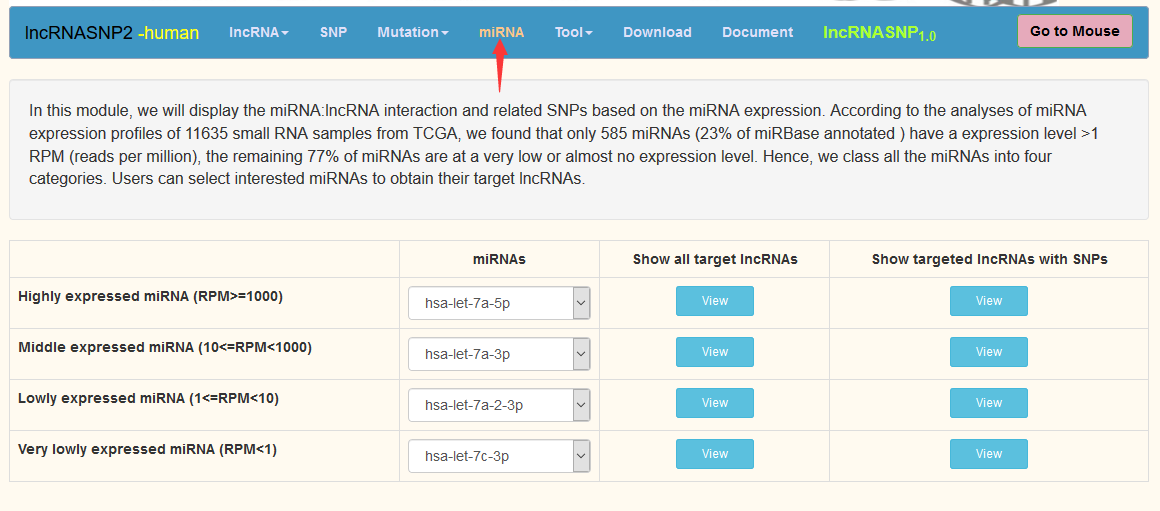
我们也可以直接用miRNA查找相应的lncRNA:
下面我们来看一篇ceRNA相关的纯生信文章Analyzing
the LncRNA, miRNA, and mRNA Regulatory Network in Prostate Cancer with
Bioinformatics Software(Journal of computational biology, IF=1.74)。
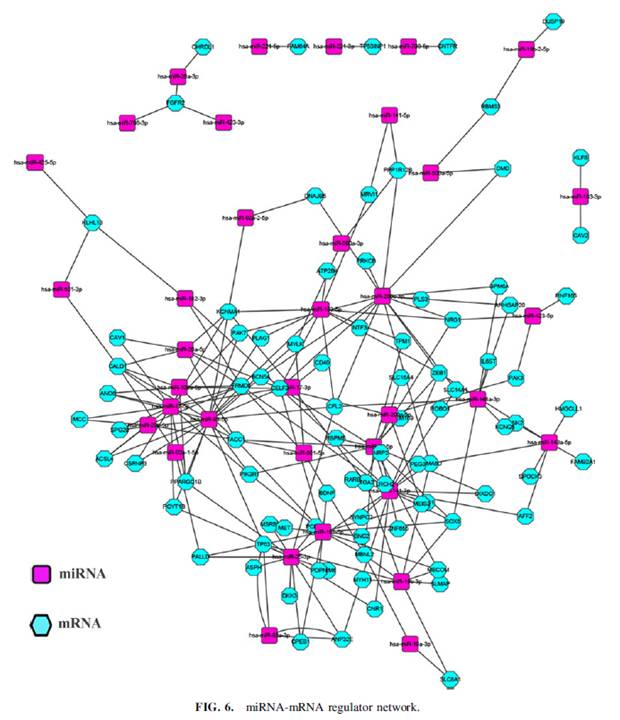
首先作者从TCGA数据库中下载了前列腺癌的mRNA和miRNA的数据,R语言分析筛选差异表达的基因,然后利用TargetScan,microcosm,miRanda,miRDB,和PicTar这五个数据库预测了miRNA和mRNA的作用关系,构建了miRNA-mRNA表达网络。
利用lncRNASNP2数据库预测lncRNA与miRNA的作用关系(这里作者分析的lncRNA已经没有芯片或测序数据的支撑了,有条件的话可以结合lncRNA的芯片数据进行分析),构建lncRNA-miRNA-mRNA的调控网络。
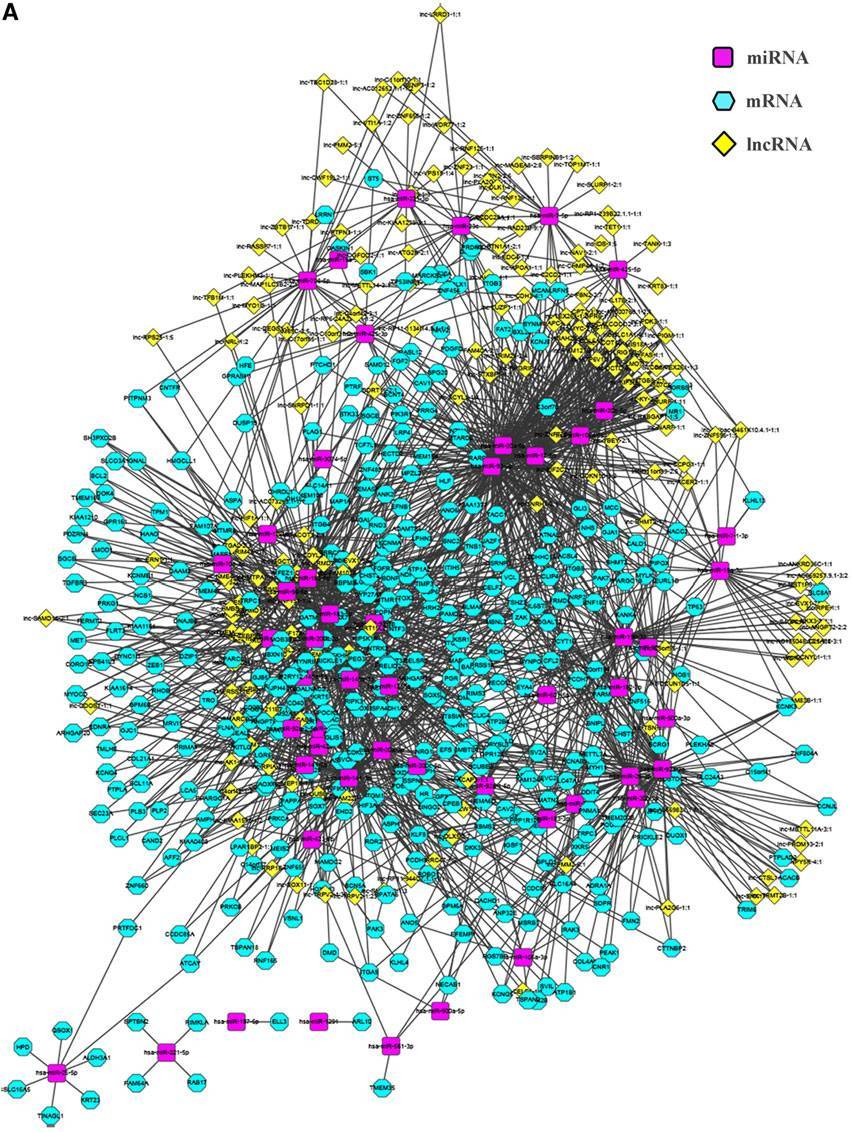
再加上ChIPBase数据库(http://deepbase.sysu.edu.cn/chipbase/)预测的lncRNA的转录因子(TF),整个网络已经相当复杂了。这篇文章的研究到这里就基本结束了,这是一个1.5分左右的研究水平。
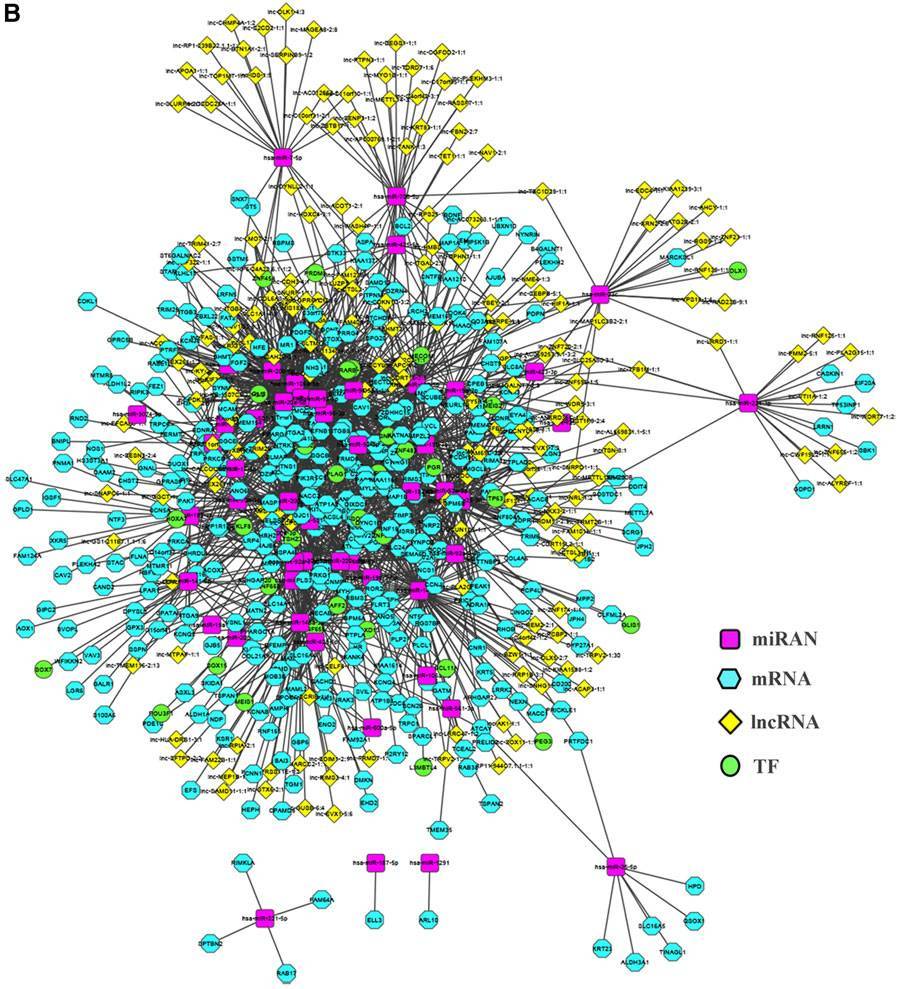

此时我们距离Oncotarget上发表的那些lncRNA的ceRNA机制研究还差些什么呢?

主要差的是后续实验,但是万事开头难,这类研究中如何选定靠谱的lncRNA才是关键,后续的实验验证都是有迹可循,容易模仿的。上图中第一篇文章中的lncRNA H19是文献报道的比较多的一个lncRNA(现在明星lncRNA越来越难做了,做的人太多了),第二篇文章则是一个数据挖掘筛选的结果,而更多的文章则是测序筛选的结果。
现在我们已经有了上面这个庞大的调控网络数据了,如何筛选我们想要的lncRNA呢?
1、网络拓扑结构分析
在之前那篇烧脑的文章中如何愉快地利用网络分析筛选关键基因,我们详细的介绍了如何通过分析节点的中心度(Centrality)来筛选hubgene,以及通过k-core解析来筛选核心网络结构。
2、组织特异性表达
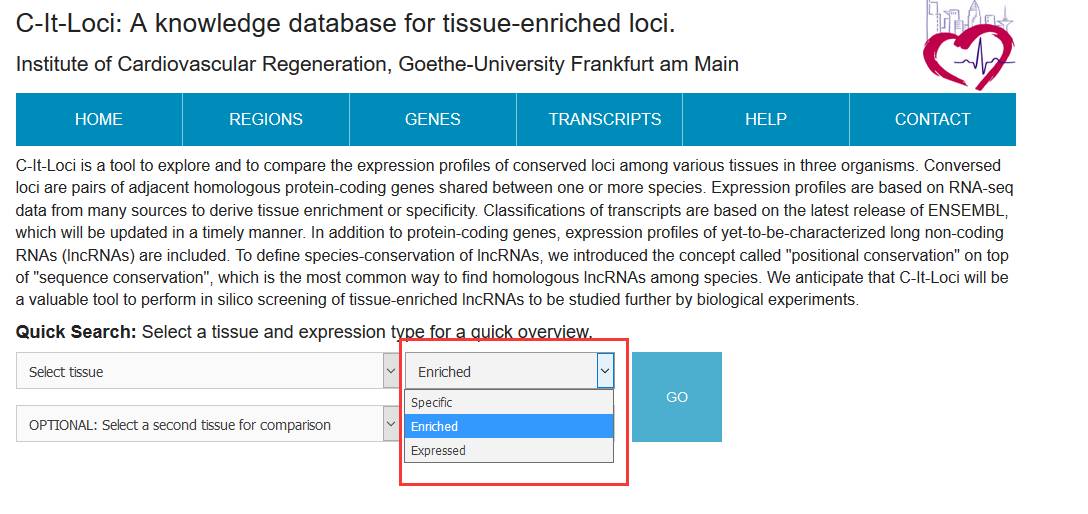
我们可以用c-it-loci(http://c-it-loci.uni-frankfurt.de/)筛选具有组织特异性表达的基因。其中Speci表示只在该组织中表达,Enriched表示在该组织中的表达水平高于总体的平均水平,Expressed表示在该组织中有表达。该数据库还提供了两个组织间基因表达水平的比较。
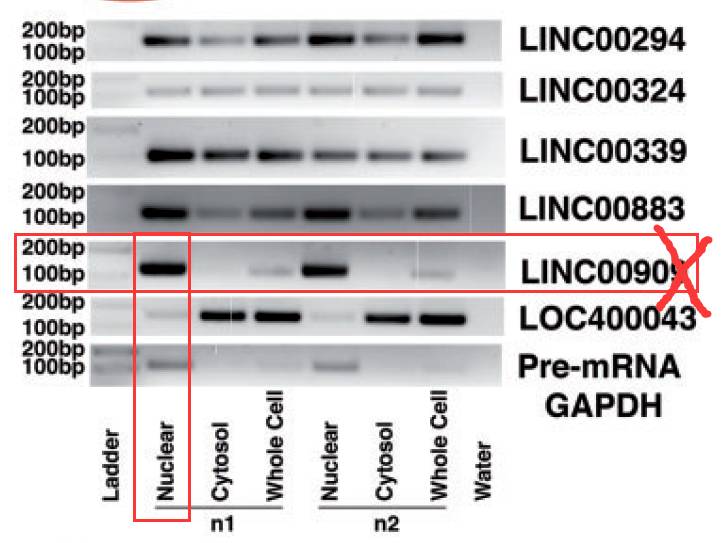
3、lncRNA细胞内定位
可以通过实验的方法直接验证,只在Nuclear里表达的就可以直接X掉了。
也可以通过数据库进行筛选,这里推荐LncATLAS(http://lncatlas.crg.eu/)这个数据库。
选定了想要研究的lncRNA,miRNA和mRNA怎么选?最简单粗暴的方法就是直接敲减后测序,再验证了,省钱点的方法就是再用数据库分析分析结合位点,筛一筛,挑一些验证。后续的ceRNA机制研究,参照上面两篇oncotarget的文章即可。

上面提到的测序虽然贵,但能分析出新的东西以供进一步研究,并且你的文章或许能达到这篇文章Screening and validation of lncRNAs and circRNAs as miRNA sponges(Briefings in Bioinformatics, IF=6.72)的level。
如果想更深入的机制研究,可以看看这篇文章Interferon-alpha competing endogenous RNA network antagonizes microRNA-1270 (Cellular and Molecular Life Sciences, IF=5.62)。
把上面的内容通通整合起来,Nature Communications (IF=11.8)就在向你招手了。

不多说了,大家撸起袖子加油干吧!

关注后获取《科研修炼手册》1、2、3、4、5,基金篇精华合集















