
上一期我们介绍了用MEGA软件构建进化树(
如何用MEGA作进化树(一)
) ,以及用它对树进行简单的调整及美化,但是图的整体还是比较单调,并且只有进化关系。如果你还想对树做精细调整及添加各种注释信息,那不妨试试iTOL(http://itol.embl.de/):
这是一款进化树在线展示及注释工具,操作简单,功能强大到令人发指。
像很多分析网页一样,你需要注册并登录账号(点击右上角的Login),才能使用它的功能。
登录后就可以上传树文件species.nwk
(百度云盘下载链接:https://pan.baidu.com/s/1pL0qTqr 密码: 345u),通过点击Upload tree file或者直接拖拽到页面(后面的注释文件同样可以用拖拽的方式直接载入)。可以看到新生成了一个Sample project叫species.nwk,默认以拖入的文件名命名。
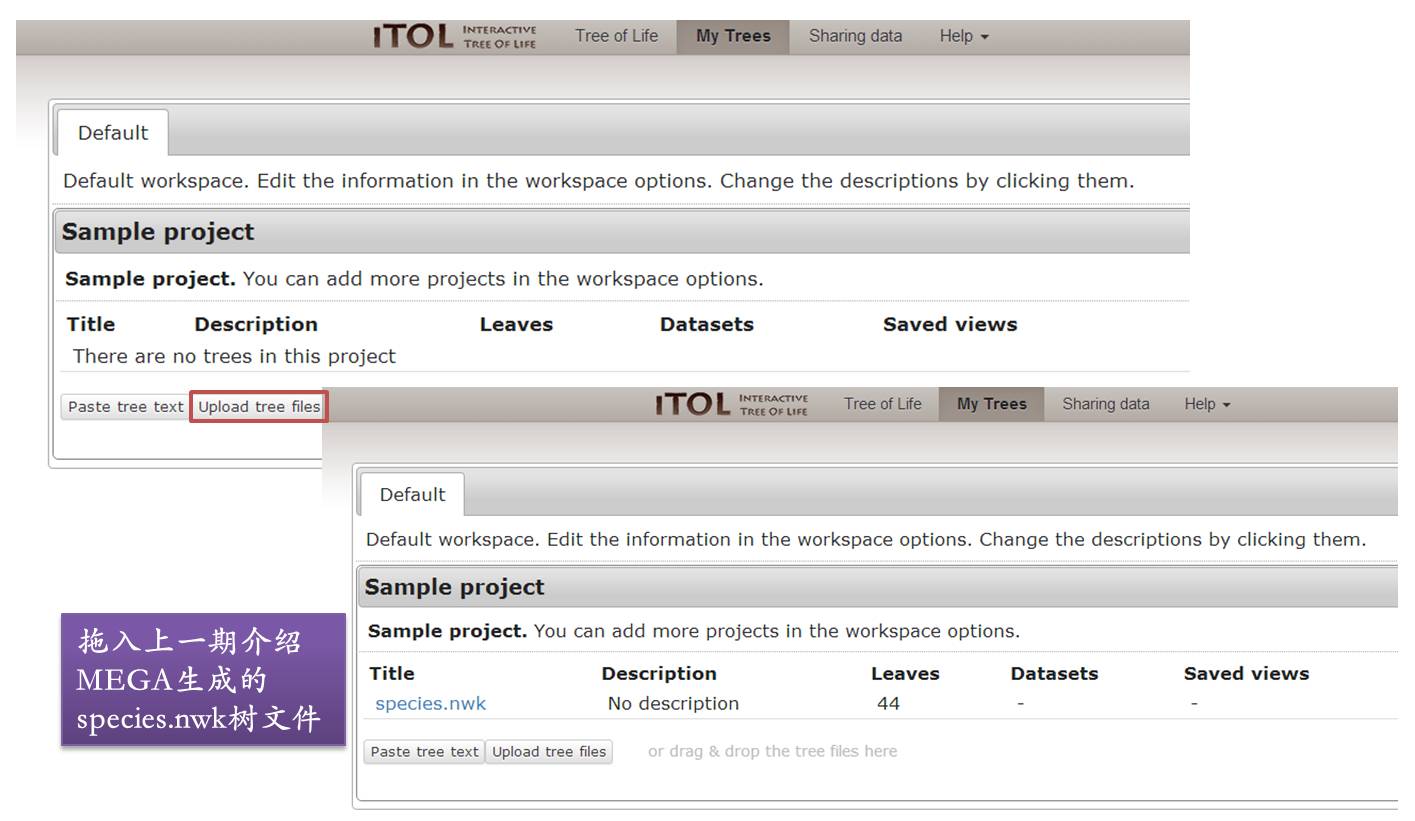
点击蓝底的
species.nwk
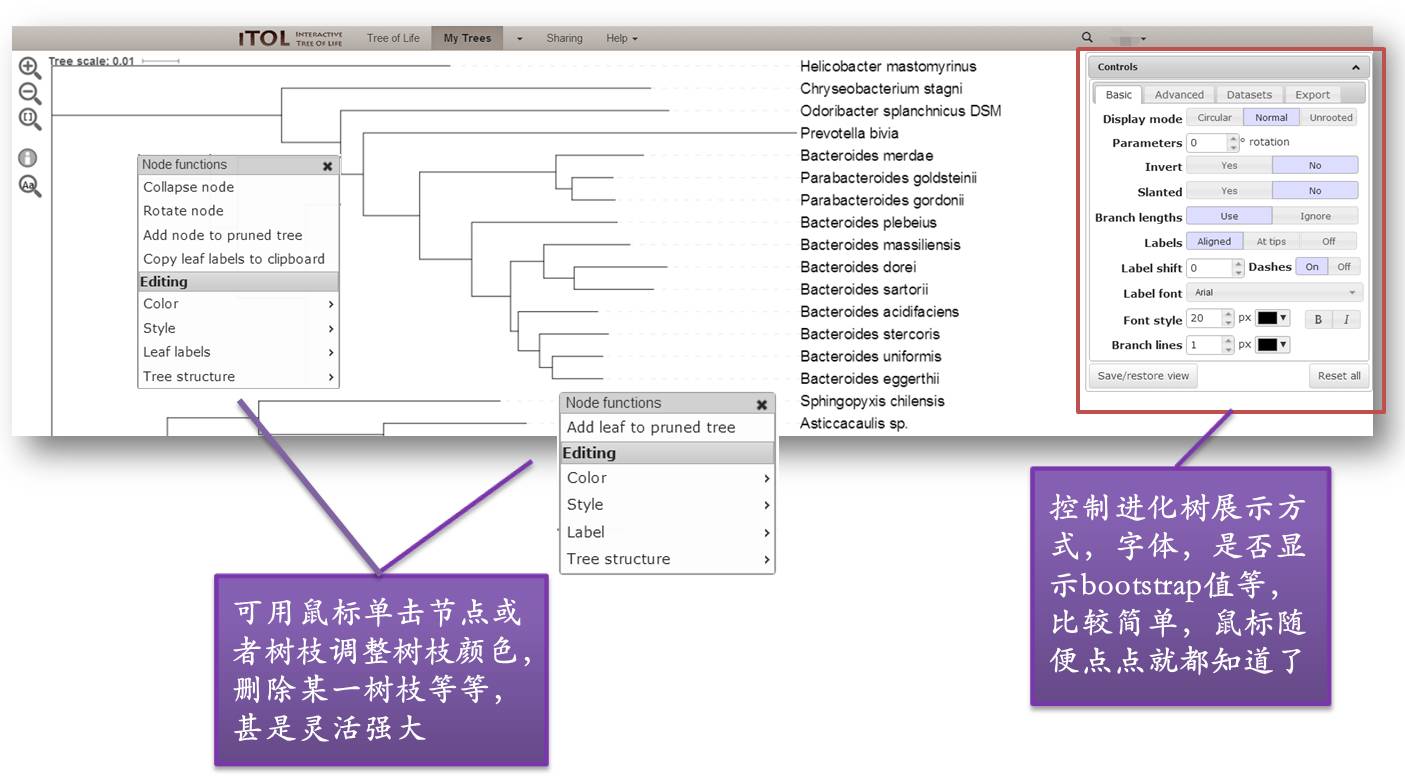
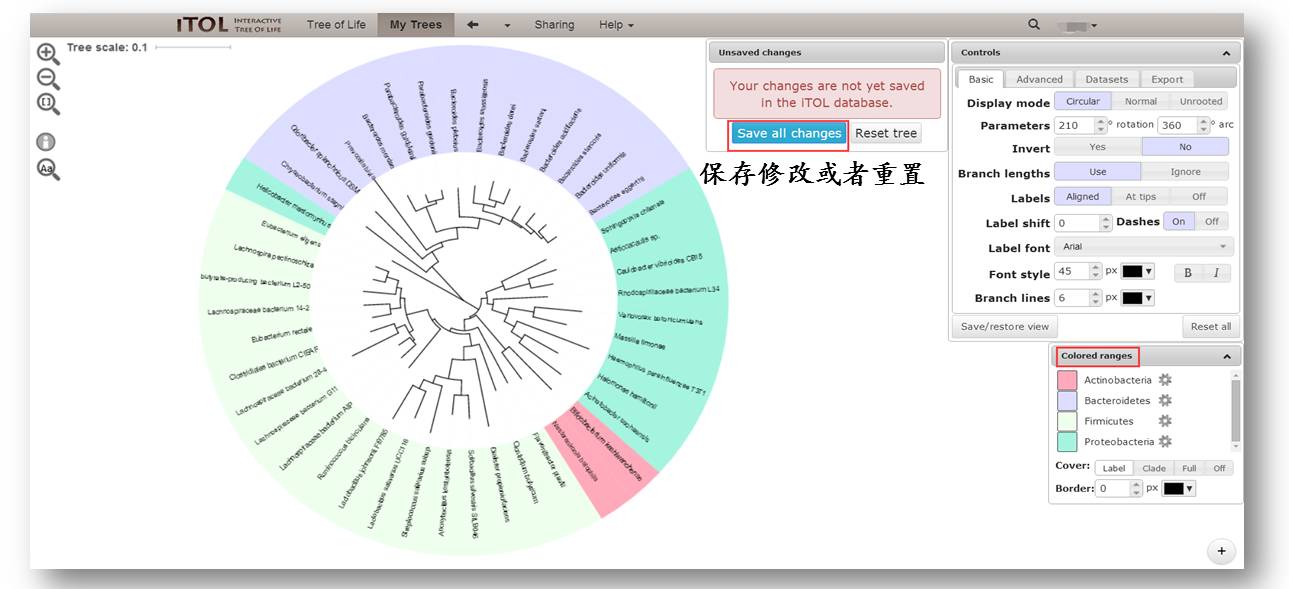
,就可以看到如下的进化树图,这是默认的形式,你可以通过右侧的Controls的工具窗口对它进行调整,还可以直接点击树枝或者节点调节树枝的颜色,隐藏树枝等等。
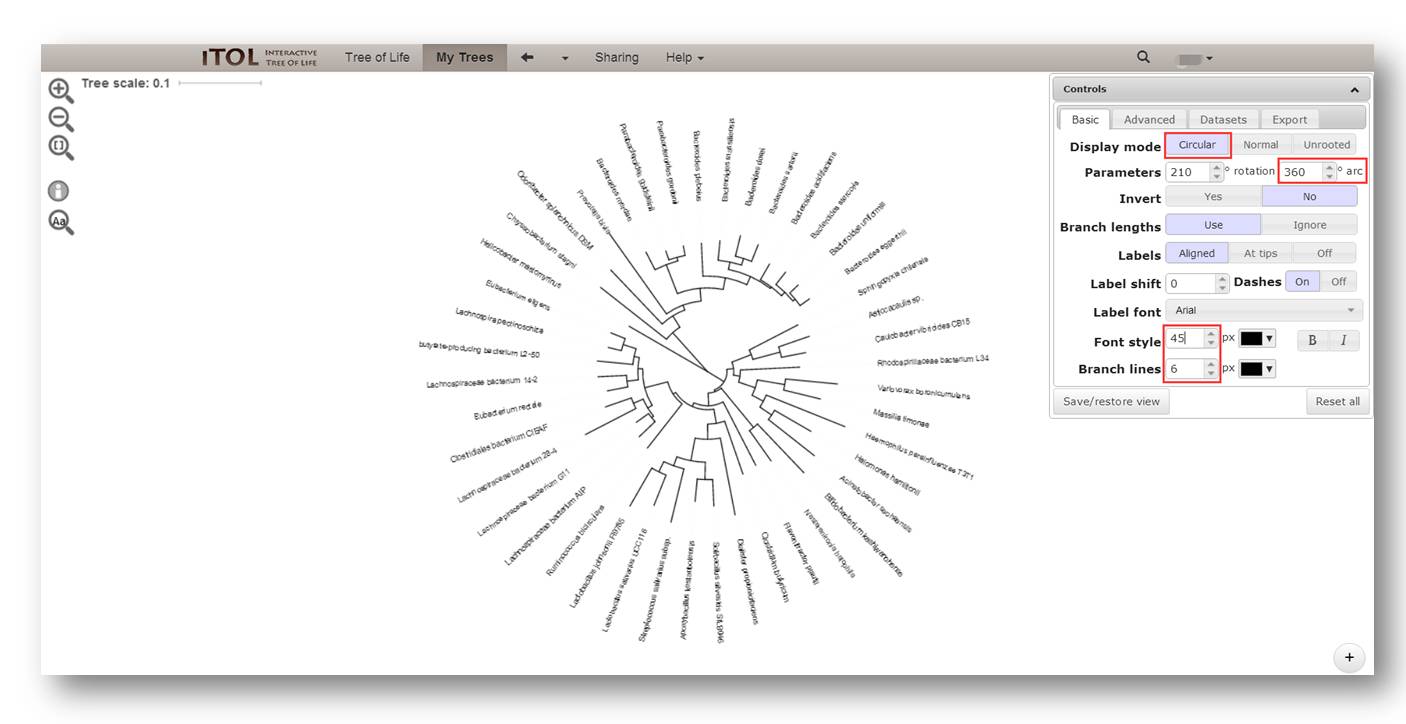
这里我们将树调整成圈图,调大了字体并且加粗了树枝。
然后我们可以直接拖入注释信息range.txt,我们这里是用不同颜色来表示不同的物种,同一个颜色就表示这些树枝所代表的物种是属于同一个门,如浅紫色表示这些物种都属于拟杆菌门(Bacteroidetes)。
这里的注释信息是画此图的唯一难点,我们以range.txt为例稍作介绍,无论什么样的注释文件,最终都要保存为纯文本文件,建议用notepad++编辑保存。

我们还可以在此基础上添加表达量或者物种丰度等信息,以下是我们添加了tol_multibar.txt注释信息。可以看到左上角同时多出了Sample name这样的图例。
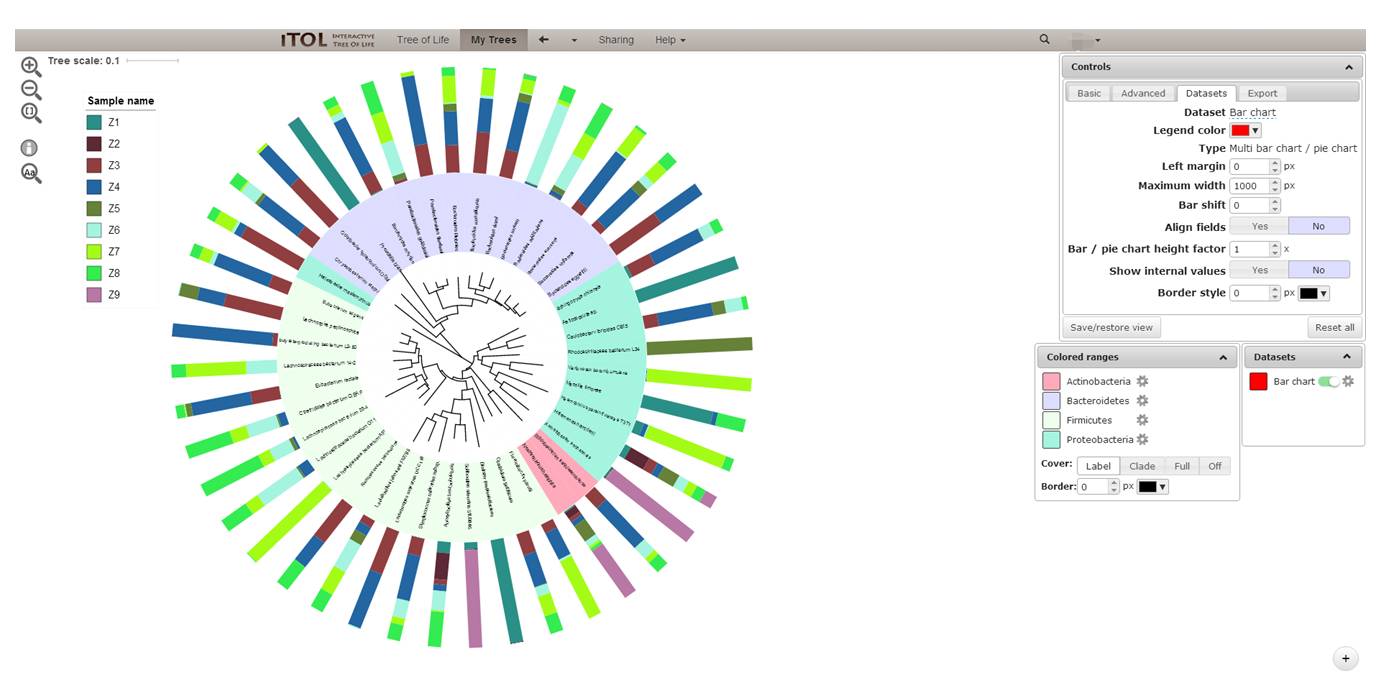

进一步,我们还可以利用右侧的Controls控制窗口对图形进行调整。最后点击Export选框进行如下设置,将生成的图片进行保存。

以上我们主要介绍了两类注释方式,实际对进化树的注释信息还有好多种,而且可以一次注释多种信息。
iTOL的帮助页面(http://itol.embl.de/help.cgi)提供了example_data.zip可供学习参考。我们可以以他们的注释文件为模板来制作我们期望的注释形式。
长按二维码识别关注“小张聊科研”

关注后获取《科研修炼手册》1、2、3、4、5,基金篇、生信特辑





