清华大学清华信息国家实验室及医学院的
张奇伟
教授是国际生物信息学界的权威科学家,在基因发现、非编码区及启动子识别、细胞周期调控、比较基因组学等研究方面有突出成就。
在生物信息飞速的研究下,张奇伟教授同时也注意到通过进阶的光学技术,对基因组物理本质对生命现象研究的重要性。

对此,清华大学张奇伟课题组研究人员联合深圳大学、柠檬数据、香港大学、哲源科技、德州大学达拉斯分校的研究人员通力合作,在深圳大学光电工程学院自主搭建的建的三维随机光学重建显微镜(3D-STORM)平台上,扩展了一种基于统计光学成像的诊断工具,用来原位捕获人基因组中特定非重复的短片段,获得了在复杂细胞核环境背景下、长度仅为2500碱基的DNA序列的3D超分辨图像,
这是迄今为止在细胞核中直接观察到的最短特异DNA序列的图像。
这项工作最近发表于国际高水平科学杂志eLife上[1]。
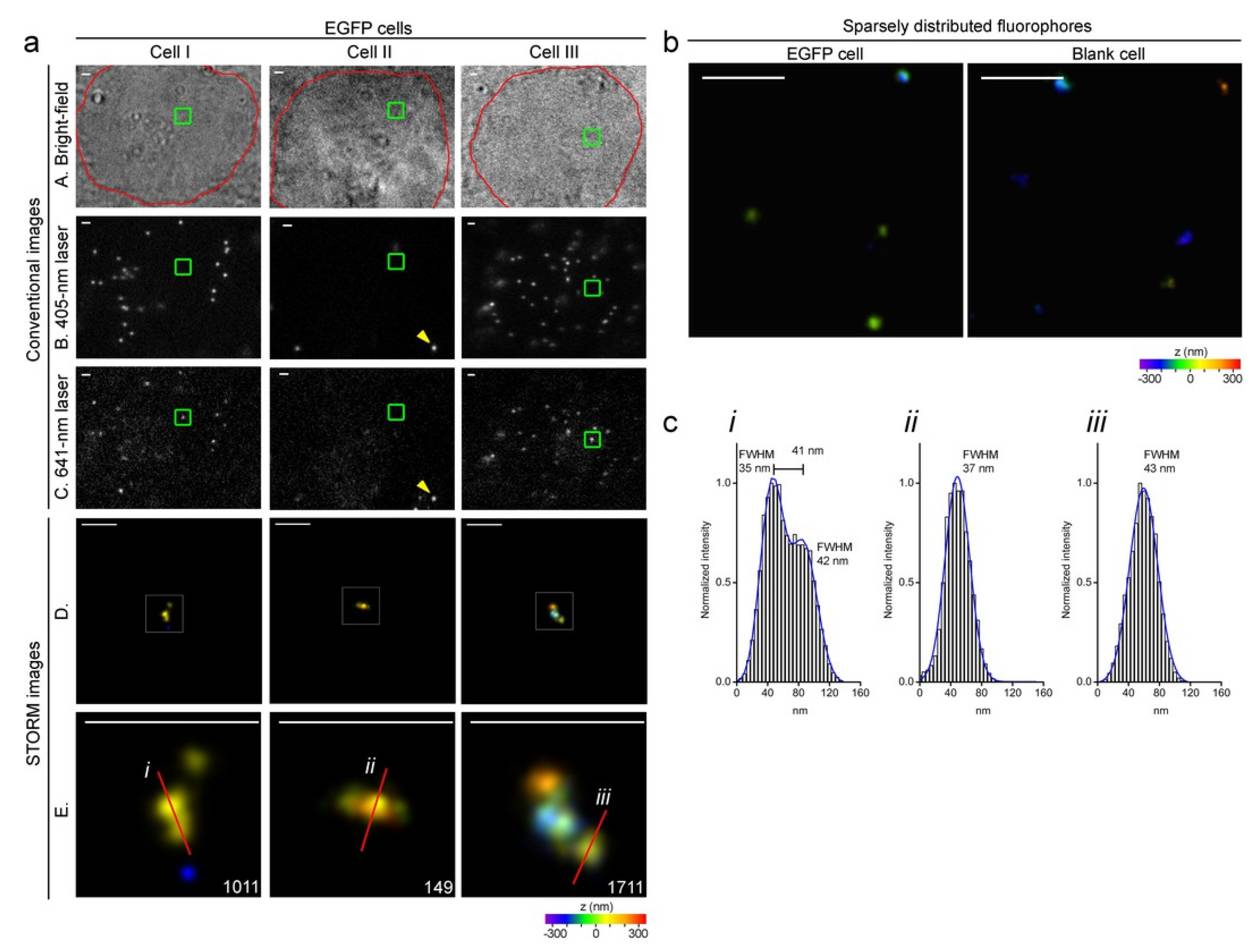
荧光原位杂交(FISH)是用于发现基因或染色体异常的分子诊断技术。首先在20世纪80年代初开发,它包括使用结合染色体特定部位的荧光探针来检测特定DNA序列是否存在。最近发表于eLife上的这项工作推动了FISH技术捕获基因组特定短片段的能力。采用了分子信标(MB)探针的这一新方法(MB-FISH)能够在纳米分辨条件下展现目标DNA小片段在细胞核内三维空间的分布;这种能力类似于在拥挤着超过一百万人的大厅中精确识别出想要找的那个人。
共同通讯作者张奇伟教授说,“
传统的FISH方法受限于各种因素(包括标记能力和光学分辨率)而难以获得基因组中特定短片段的清晰微观图像
。”
2015年,在哈佛大学的Wu Chao-ting教授和庄小威教授实验室联合建立了寡核苷酸探针FISH(Oligopaint-FISH)与STORM相结合的新方法,能够对最短为4900碱基的非重复基因组区域进行超分辨率成像[2]。“虽然这种技术在染色质结构域精细组织方式研究很有前景,但我们希望能够发展一种新方法:
不但进一步提高基因组序列分辨率,而且,很重要的是避免繁琐的探针制备过程对实际应用的限制。通过这种新方法,我们将可以精确地靶向基因增强子和启动子,使其相互作用原位可视化,同时可以用来检测癌症和其他疾病中单细胞内特定DNA小片段及其之间相互作用的变化。
”张奇伟教授说。
为了开发该方法,来自深圳大学的第一作者兼共同通讯作者倪燕翔博士首次把MB概念应用于基因组特异靶序列的标记上。 MB的设计其实已存在多年并广泛应用:它通过形成发夹结构、淬灭未结合或脱靶探针的荧光团,从而大大减少非特异探针的荧光。但是,对特定双链DNA序列的某一单链进行标记实际上非常困难:一方面因为目标序列拷贝数低,另一方面,需要在确保目标双链被打开时,MB探针能够竞争过互补链而稳定结合到目标链上。这不仅需要摸索最适合的标记实验条件,同时因为其单分子超分辨成像的本质决定需要进行大量工作以优化3D-STORM成像条件,因此目前成功使用MB标记基因组位点还属于首次。
“
实际实验效果要好于我的预期,”倪博士说,“MB设计不仅显著降低了未结合和脱靶探针的荧光,而且有效减少探针与基因组其它区域的非特异性结合,后者也非常重要
”。
倪博士补充道:“目前的阶段性进展虽然突显了我们在单细胞纳米分辨尺度上原位成像人类和小鼠基因组中特异短序列的能力;相信我们团队建立的方法在接下来研究人类正常和疾病(比如癌症)细胞中3D基因组结构功能中释放更大的潜力。”
















