生物极客按:
有效的gRNA
设计
工具,
对
我
们进行基因编辑有着重要的意义,来自哈佛大学的
GM Church
课题组在
Acs Synthetic Biology
上,开发的
SgRNA Score2.0
,可以用于包括SaCas9等多种sgRNA的预测,方便人们更好的利用基因组编辑工具。

论
文解
读
:
目前已
经有多个课题组评
估了数百到数千个
sgRNA
的相关的序列特征并且已
经创
建了公开的可用
软
件来帮助研究人
员进
行
sgRNA
选择。来自
GM church
课题组开发的
sgRNA Scorer
1.0
自
2015
年
7
月
发
布以来,共有超
过
7000
个独立用
户
,
87
个不同的国家的研究人
员进
行使用。
然而随着CRISPR
领
域的不断深入,科学家
们
从其他
细
菌物种中
鉴
定
CRISPR
系
统
,同
时
也开
发
了相
应
的真核
细
胞
编辑
的能力
.
然而,如果一个一个不同的cas9
进
行
验证
相
应
的工作量非常的
庞
大。因
为
目前的策略需要需要
为
每个要分析的
CRISPR
蛋白
产
生和
测试
sgRNA
序列文
库
。
因此,科学家
们
开
发
新的工具
试图预测
多种
CRISPR
蛋白
质
中的
sgRNA
活性。
科学家们构建了
支持向量机(SVM)模型
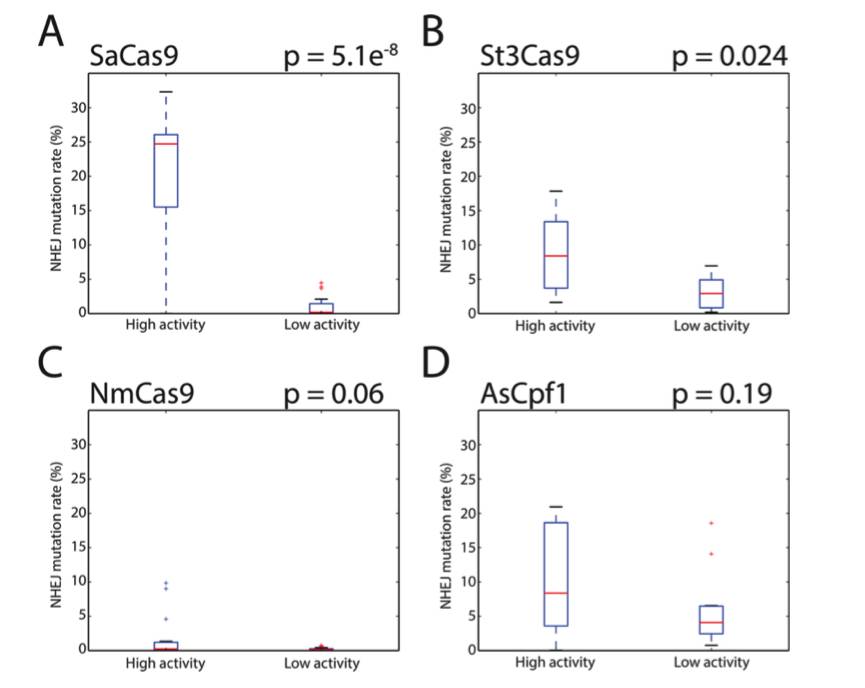
,用来评价其他三个Cas9同源物和一个非Cas9系统----Cpf1的预测能力。
首先科学家们合并了SpCas9(133高,146低)和St1Cas9(82高69低)的高活性和低活性sgRNA序列,产生215个高活性和215种低活性sgRNAs 。随后,将这430个sgRNA序列用作新的SVM的基础。我们的新模型的10倍交叉验证产生了73.7%的精度,72.8%的精度和75.8%的回收率,这与以前的SpCas9和St1Cas9的各个型号相当。科学家们评估新模型预测SpCas9和St1Cas9的sgRNA活性的能力。使用以前生成的靶基因座测序的预测数据,我们重新计算了这些sgRNA,发现我们的新旧模型之间的预测分数与SpCas9的相关性为0.997,St1Cas9的相关性为0.940,表明将来自两种不同的Cas9直向同源物对每个Cas9的预测能力没有不利影响。
1.科学家们大胆推测不同crispr系统之间sgRNA活性的相关性,从而极大的拓展了使用。
2.科学家们使用SVM支持向量机,从而识别活性背后潜在的规律。方便更好的预测。






