作为科研者,如果你到现在还不知道
生物信息学
,那真的太out了,生信有多火?
拿最有名的数据库TCGA(
Cancer Genome Atlas
)来说,
目前已经收录了来自1万多例病人的33种癌症的数据,2.5PB的数据量。
在pubmed中随便搜一个TCGA,就可以发现近年来相关文献发表量在成指数级增长。

这其中,一部分是利用TCGA数据库发表的纯生信文章,比如
2018年4月
发表在Cell上的一篇
“
Comprehensive Characterization
of Cancer Driver Genes and Mutations
”。
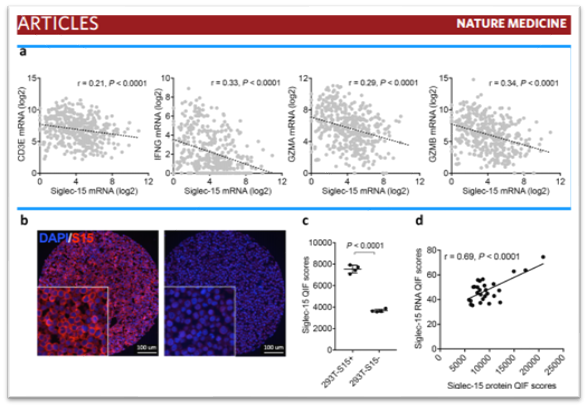
也有很多利用TCGA数据库的图片给文章增色的,
比如
2019年3月4,耶鲁大学
陈列平
教授团队在
Nature Medicine
上发表题为
Siglec-15 as an immune suppressor and potential target for normalization cancer immunotherapy
的文章,里面就利用TCGA数据库分析了Siglec-15 和T cell signature genes (CD3E, IFNG, GZMA and GZMB) 在膀胱癌中的关系。
而这些都不需要额外的湿实验,大大提高了临床医生的科研效率。这也是生信之所以火的原因了。
但你可能要问,如果不会R语言,不会代码,怎么利用利用生信来提高科研效率呢?
或许参加过好几千一次的培训班,但结果就是,课堂上什么都会,一回去自己弄马上就被各种
报错
打败,自学视频,同样是困难重重。

科研界一直需要一个功能强大,不需要代码,可视化分析工具。
科研者之家(Home for Researchers)
上有一个生信零代码版块:

已经有数十种分析模块,整合了
TCGA,GEO,Target,ICGC,CCL
E五大公共数据库的样本数据,
可以直接拿来用
。

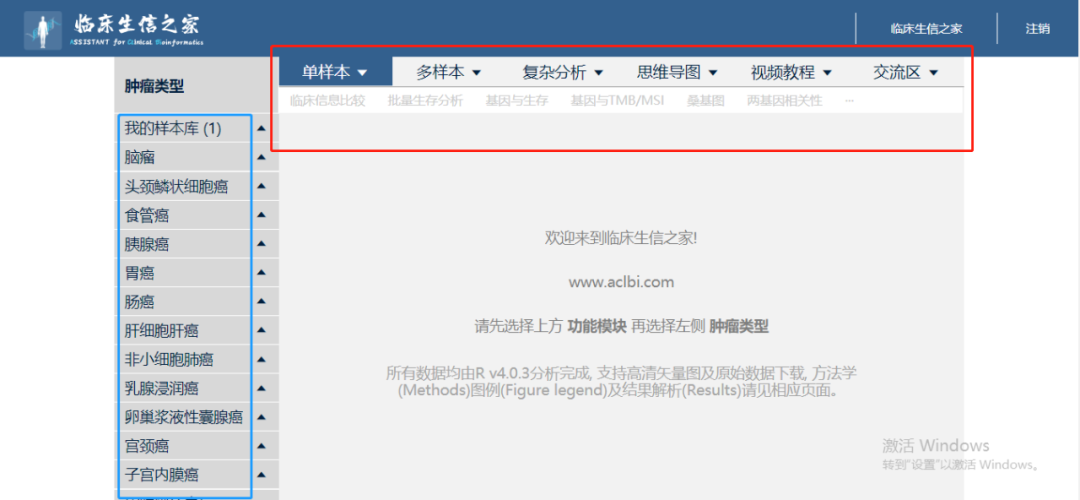
平台是由几个临床医生联合生信大拿一起搭建,界面极其简介。
比如在TCGA模块,
界面极其简介,左侧栏是TCGA上所有33种肿瘤类型,之后第二期会纳入非肿瘤的疾病。
顶部是各种分析模块。
首页特别注明了,所有数据均由R v4.0.3分析完成, 意思就是说在平台上分析的结果和拿R语言敲代码分析的结果是完全一样的,这就太爽了。