
背景介绍
越来越多的科学家用下一代测序
(NGS)
来诊断人类遗传病。
NGS
通常用于罕见病和癌症基因的遗传研究。全基因组,全外显子组和基因
Panel
等
NGS
技术也广泛应用单基因和复杂疾病的临床研究。
NGS
的主要变异类型包括单核苷酸变异和拷贝数变异,存储在一个
VCF
文件中。
VCF
文件通常包含数万个有别于参考基因组的位置
(“
即所谓
VCF
,检出变异列表
”)
。随后
,
通过对
VCF
文件注释,来分析筛选这些
SNPs
和
indels,
例如受影响的基因家族
,
遗传连锁信息
,
人群中频率对蛋白质的影响例如
PolyPhen
,
SIFT
和
SNAP2
,
对所有变异进行筛选
(
二次分析
)
。根据我们感兴趣的遗传模式,筛选条件,最终会留下几十到几百个变异位点,这有助于生成一个
“
中等严重变异列表
”
。
在上述的分析中,对基因序列个体的疾病表型进行了分析。作为引入表型关系的第一步,利用疾病变异的数据库
(
比如
ClinVar
或
COSMIC
)
为了进一步过滤序列变异。
一个典型的基因中有大量新的变异,这些变异尚未证明与疾病或者表型有关。大多数情况下,没有一种算法直接实现变异位点和疾病之间关联。此外
,
研究表明大约每
100
个功能丧失变异大约会导致
20
基因完全失活,频率和预测这些筛选条件也不足以确定致病突变。为应对这些挑战
,
基于基因层面的解释非常必要。这种算法需要为基因寻找与疾病或表型之间的关联,而不是位点。
在本文中,我们描述了
VarElect
中的构成和使用
(ve.genecards.org), VarElect
是一个基于网络的表型关联工具
,
利用
GeneCards
及其附属数据库中的大量信息,为基因和表型直接关联打分。
VarElect
为
NGS
表型关键单词提供了直接检索和间接检索两种方式。得益于
GeneCards
的优秀表现,
VarElect
的间接检索模式通过大量的注释信息,将基因与表型相互联系起来;为基因排序同时提供强大的事实证据,指出基因可能与病人的疾病有关。
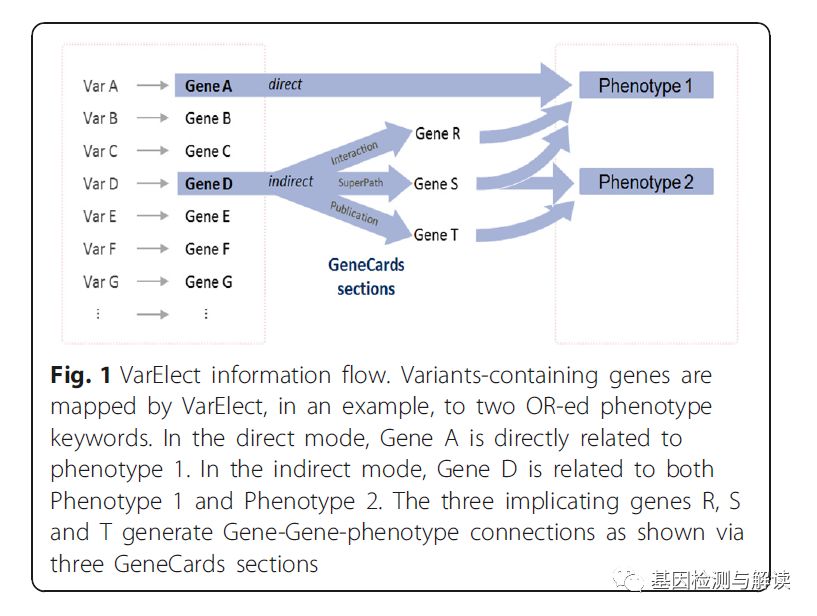
研究方法
得分
VarElect结果中的得分主要来自于Elasticsearch技术。该基因在GeneCard中出现的频率决定了得分。Elastic和Solr是基于Lucene搜索机制,可能会引起各种偏差,例如Solr通过对该主题研究发表的资料进行语义搜索和科学文献搜索。(cf [21]).此时,VarElect界面中,给出的表型关证据链接就具有重大的意义
(例如疾病信息或者疾病功能等)。对于布尔逻辑定义的多词查询,为了确定查询的匹配的基因,具体的查询关键词被分解,以便进行搜索(以单词或者用引号作为词组检索),每一个单词或者词组都会获得一个得分,最终的
VarElect
分数由每一个关键词的得分相加而来。
与其他软件性能比较
为了测试
VarElect
的检出性能
],
我们使用了
34
组平行实验
,
包含了致病基因(探针基因)与这个基因会导致的疾病
/
表型
/
症状。其中,
13
组数据是基于威兹曼研究院实验室的
WES
数据,
9
组数据来自
ToldotDNA
的
WES
数据。
(http://www.toldot-dna.com/ content/about-us),12
组来自发表文献
,
包括在
MalaCards
中的
4
个。在运用
VarElect
检索时,每个探针基因都有干扰基因,这些基因在原始实验时,就已经纳入考虑,另外我们还从
Illumina TruSight WES
捕获试剂当中随机挑选
500
个基因一同来进行筛选。
结果与讨论
VarElect
是用于实现基因和表型关联的在线工具,可以根据从实验数据文件中导入的基因,查找疾病相关的表型和症状。
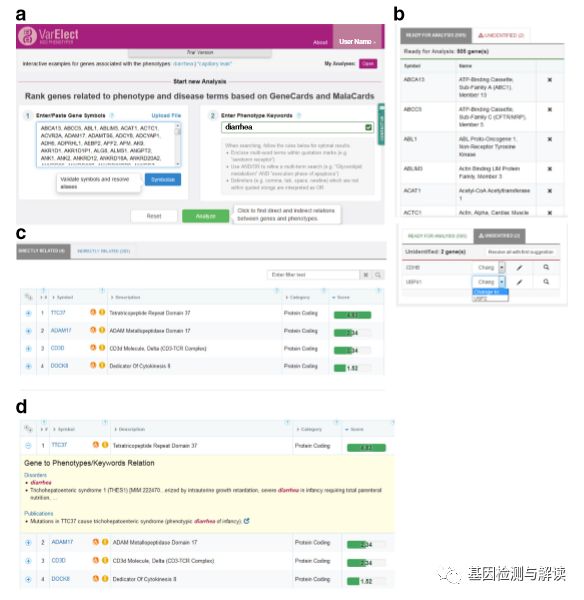
图
2VarElect
界面和直接检索的例子
VarElect
输入
VarElect
不限制基因输入的数量,且提供了一个“
symbolize
”工具,用于验证输入的基因是否是官方名(图
2b
)。通过与用户的交互,非官方名称的基因(即别名)就被翻译成相应的官方名。这个功能是根据
GeneCards
基因综述部分开发的。
VarElect
表型可以实现自由文本输入,没有必须输入专业术语的限制,并且可以实现模糊检索、按照布尔逻辑检索。这样
VarElect
就特别适合用于查找表型相对模糊的遗传病病因。这背后离不开
GeneCards Suite
中提供给
VarElect
的大量信息(参见
VarElect
知识库)
.
。
VarElect
的布尔逻辑在以下情况下非常有益:
第一种情况是患者有许多的同义词(例如“截瘫”,“下肢轻瘫”,“下肢瘫痪”,“下肢瘫痪”)定义的表型。在这种情况下,用户显然希望包含所有可能性,因此建议使用
OR
搜索关键词。第二种情况是综合征,试图找出同时伴有的几种症状(例如“智力低下”,“面部畸形”,“共济失调”),在这种情况下,建议使用
AND
逻辑。
VarElect
结果和评分
在分析阶段,
VarElect
根据输入的表型关键词搜索与
GeneCards
中所有关联的基因。依靠
GeneCards
强大的搜索引擎,自动从
160
多个数据源中挖掘所有关键信息。
GeneCards
知识库中包括人类疾病数据库
MalaCards
,包括来自
71
个数据库的
19,552
种疾病条目
; PathCards
根据基因重叠的内容,将
12
种人类生物通路整合到约
1000
个
SuperPaths
中
[19];
还有
LifeMap Discovery
,其人工筛选组织和细胞基因表达信息,内容涉及胚胎发育和干细胞领域。
VarElect
分析的结果按照基因和表型的关联性由高到低排列,也可以通过表型关键词匹配的次数来排序。结果显示在两个单独的选项卡中,一个用于显示直接关联的基因,另一个用于显示间接关联的基因。后者的分析处于“联合作用”领域,即基因
A
与表型无关,但是(在一个例子中)基因
A
和基因
B
属于相同的通路,而基因
B
与表型相关(图
1
,图
2c
)。基因
B
被定义为基因
A
(有关基因)与表型之间的间接关系的“有关基因”。
“
MiniCards
”会在分析界面提供证据,这一功能和
GeneCards
中的功能类似。它显示的是与表型关键词相关的
GeneCards
或者
MalaCards
数据库中概述内容,并且提供一键直连。对于直接模式,有单层
MiniCards
显示。间接模式有两层证据:第一层(图
3a
)显示每个相关基因有
5
个相关基因,第二层(图
3c
)显示
MiniCards
与
5
个相关基因的表型关系。
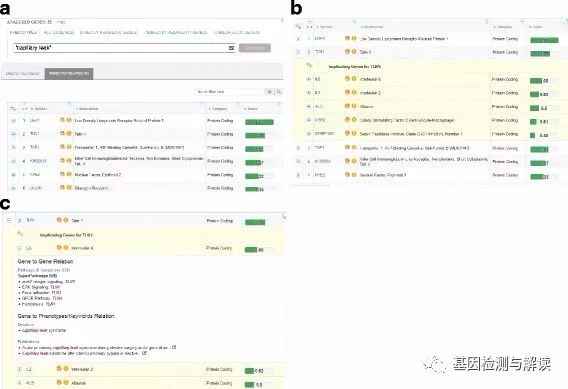
图
3VarElect
信息显示和间接模式
VarElect
报告和集成
Excel
报告
VarElect
的搜索结果可生成
Excel
文件,文件中将得分基因列表分成直接和间接结果。在间接结果的
Excel
中,每个基因,与有关的基因,还有间接关联的分数都会列出。不仅可以在网页上使用
MiniCards
,所有列的信息还会体现在
Excel
中。
TGex
和
VarAnnot
对于喜欢用
VCF
文件分析的用户,可以有两种方法更好的使用
VarElect
:第一个是
TGex
平台。
TGex
会实现
VCF
文件的注释,
VarElect
表型关联,还可以提供可视化的分析流程,帮助生成高度定制的自动化报告,完美的解决了
VCF
数据到临床应用的难题。第二个是
VarAnnot
插件,对
VCF
执行注释后,
VarAnnot
将表型分数输出到新的列中。
VarElect
可以整合到任意一个工具里。
VarElect
优点
依据表型排序解读
VarElect
最核心的优势在于:依据表型与基因之间的关联进行打分排序。大多
NGS
解读都是根据一些网络数据库的注释结果来进行的。常用的数据库比如说
COSMIC
,
ClinVar
等等,这些数据库,有的是疾病数据库,有的是位点数据库。这些数据库并不能满足
NGS
的实际应用需求:一方面,并不是所有的疾病都有明确的病名,这些疾病通常会有很复杂的表型,这些表型可能对应很多种疾病;另外一方面就是有的基因上面可能有很多新发的变异并没有相关报道,检出变异正好就是致病位点的可能性极低。
VarElect
的自由文本检索功能,可以快速的以基因为中心,在后台强大的数据源当中找到疾病和基因位点的关联。
针对基因、位点实现区分
VarElect
的一个优点尽管他的算法是基于基因的,但是他仍然很清楚的区分了分析是疾病和基因,疾病与位点之间的关联。假设我们分析的
VCF
当中,每一个基因都有多个变异位点,很多位点都对蛋白功能产生影响,在使用
TGex
时,软件会帮助我们初步滤除低质量、低影响的变异,最终只列出可信度高的位点,并给出相应的注释信息。
随后我们可以按照分数的高低,来查看相关基因的
VarElect
结果。如果一个基因有多个高影响的位点,那么我们就可以查看
TGex
界面当中这个位点的其他信息。(译者注:其他信息包括变异所在区域,位点的等位基因频率等等)
提高效率
传统的
NGS
解读方法需要遗传学家耗费大量时间去寻找基因和表型之间的关系。
.
我们需要查看每一个发生变异的基因,在基因数据库(比如
GeneCards
)当中的基因描述,然后还需要根据表型找出怀疑的疾病。有了
VarElect
,可以一键查找基因
-
表型之间的关联。
使用灵活
VarElect
允许使用者根据自己的分析逻辑对变异位点进行筛选,并其针对
VCF
当中包含的基因位点数目,没有上限。
数据全面
由
GeneCards
旗下一系列的数据提供证据支持,
VarElect
阐述了基因和表型之间的无数联系,这些关联是对于解读至关重要证据。
搜索引擎强大
VarElect
使用的弹性搜索引擎,提高了查找表型相关基因的速度、效力和准确性。
自由文本检索
VarElec
对使用的表型关键词没有任何限制,相比限定的术语,
VarElect
可以利用更广泛的关键词搜索相关基因。(译者注:
VarElect
现已可以使用中文关键词进行检索,
欢迎各位登录
tgex.genecards.cn
试用这一功能
)
和其他工具的比较
Phenolyzer
34
组实验数据同时上传到
VarElect
和
Phenolyzer
用于比较分析结果。我们使用的是添加了
Mentha
蛋白质交互数据库的
Phenolyzer
网页版。目标基因的排序结果将作为检出性能的判断依据。(图
4a
)结果显示目标基因的排序在
VarElect
中比
Phenolyzer
靠前。这一结果在那些复杂表型的案例当中,更为明显。导致这一现象的原因可能是这两个工具在功能上的一些差异。第一个不同之处在于,当需要采用严格的
AND
筛选逻辑,只有同时匹配上所有关键词的结果才会出现在列表中。为了演示这种检索逻辑下的筛选强度,一些关键词在
VarElect
当中分别用
OR
和
AND
的逻辑进行检索,我们发现后者的排序等级提高了。
(Additional file 2: TableS1).
第二个不同之处在于,我们发现
VarElect
提供的是自由文本检索,而
Phenolyzer
的输入关键词只能从
HPO
、
OMIM
当中提取。结果表明,
VarElect
的检索逻辑检出性能更好。
(Additional file 2: TableS1).
Exomiser
Exomiser
用针对全外数据,实现表型和位点关联的工具。
Exomiser-hiPHIVE
算法综合考虑了位点的稀有性,预测的致病性以及人类疾病和动物模型(小鼠、斑马鱼)的相似性来对每个基因进行排序。我们同样是利用探针基因进行平行实验,来对
VarElect
和
Exomiser
进行比较。我们发现,无论是哪种检索逻辑,
VarElect
的表型均优于
Exomiser
。
Ingenuity variant analysis (IVA)
IVA
利用“
Biological Context filter”(BCF)
来寻找基因和表型之间的关系,限定了必须使用
IPA
当中的表型术语。那就要求用户需要知道变异
/
基因和表型之间的关联,随后用户需要到生成的“通路和表型”图当中
(
部分类似于
VarElect
的间接模式
)
找到相关性的证据。
IVA
工具没有具体数值来表示变异
/
基因和表型直接的关联性。
VarElect
的自由文本检索和表型关联打分工具,可以直接按照分值高低进行表型关联,这一点
IVA
并没有一个打分工具,因此,没办法按照关联性高低来排序。另外一点
VarElect
表现优于
IVA
的就是,在涉及基因和基因之间的关联导致表型的发生,需要分析不同通路彼此间相互影响的时候,这个时候对系统自动化要求更高,点击不需要用户的更多操作,
VarElect
就可以将所有信息呈现。我们同样采取平行对照试验,无论哪种搜索模式,
VarElect
的表现均超过
IVA
。
Phevor
这个工具是
Omica
根据表型术语来开发的表型关联工具,它整合了
HPO
,
MPO,DO,GO
等数据库。用户在
Phevor
当中直接比对到的关键词类似于
VarElect
的直接检索模式,而如果这些关键词被包含在一些扩展性的内容当中,然后需要根据匹配的关键词,来评估基因,这个就类似于
VarElect
的间接检索模式。
因此,在不知道疾病(表型)和基因关联的情况下,
VarElect
和
Phevor
的检出能力可以做一个比较。

















