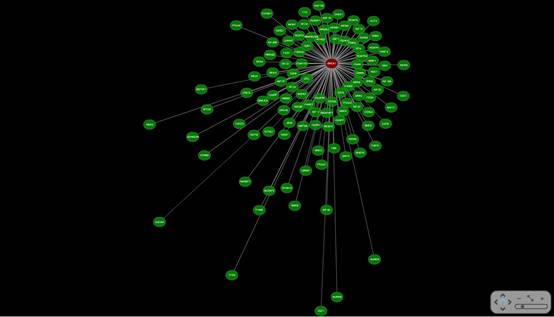
分子生物网络展现了分子之间的相互作用,可较为深入地反映基因间的表达调控关系,在组学机理的研究中尤为重要。
基因共表达网络分析(
Gene Co-expression Network Analysis
)
是基于基因间表达数据的相似性而构建的网络图,图中的节点代表基因,具有相似表达谱的基因被连接起来形成网络。
Network propagation: a universal amplifier of genetic associations. GENETICS

今天给大家介绍一个共表达关系网络分析以及可视化的
在线
工具
Coexpedia
(
http://www.coexpedia.org/
)
。
Coexpedia
搜集了
GEO
数据库中的测序数据(人和小鼠一共
900
多个数据集),对每个数据集单独进行了共表达分析,然后将所得的共表达关系汇总构建成数据库。通常我们利用R语言做共表达分析只针对单个数据集,相比而言
,Coexpedia的数据来源就要广的多,结果的可信度理论上会更高一些。

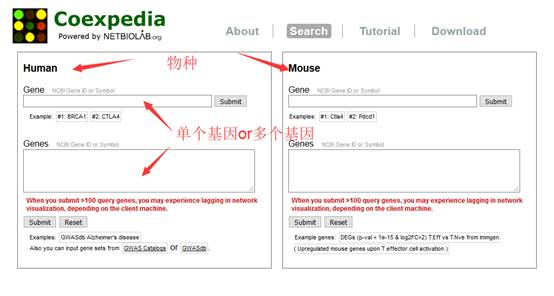
点击
Search
即可进入共表达关系查询页面。物种分人类和小鼠,毕竟
GEO
上还是这两个物种的数据居多,其它的数据太少。
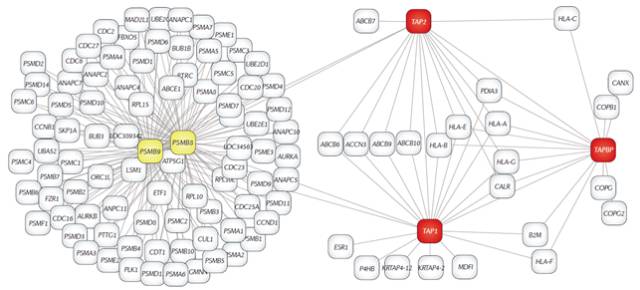
我们先来看一下单个基因的查询结果:
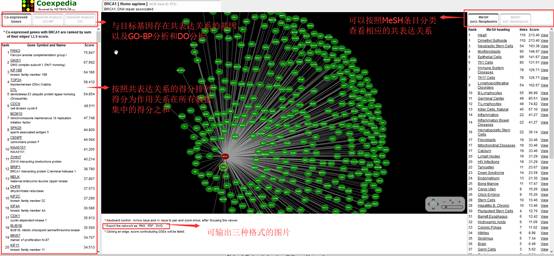
左侧显示
的是与目标基因存在共表达关系的基因列表,依照共表达关系的得分排序,这一得分是该
pair
在所有数据集中的得分之和。数据库中还提供了
GO-BP
(
Gene Ontology - Biological Process
)注释和
DO
(
DiseaseOntology
)注释。
右侧显示
的是按照不同
MeSH
(
Medical subject heading
)条目
进行分类的结果,比如选中
Heart
这一条目,我们即可查看在
Heart
相关研究中得到的共表达关系(黄色描边显示)。
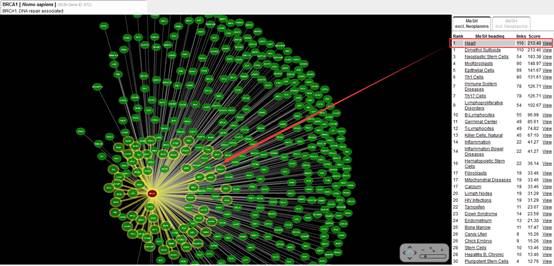
点击最右侧的
view
后可以生成一个新的网络: