题记:这篇读书笔记介绍线性模型中的高级特征选择方法:包括领回归、Lasso回归和弹性网络。本文参考Cory Lesmeister博士主编《Mastering Machine Learning with R》(第2版)一书,这是一本机器学习的入门级教材。数据来自R语言ElemStatLearn包自带prostate数据集和MASS包自带biopsy数据集,代码来自《Mastering Machine Learning with R》这本书配套的代码,笔者对部分代码进行修改。本文是个人的读书笔记,仅用于学习交流使用,我只是一个知识的搬运工。
1.背景知识
随着要处理的数不断增大,最优子集和逐步特征选择这样的技术时间成本越来越高——即使使用高速计算机,在很多情况下,要得到一个最优子集的解需要花费数小时。这种情况下,是否有更好的解决方式?本章讨论正则化方法是解决办法之一。正则化会对系数进行限制,甚至将其缩减到0。现在有很多种算法或算法组合可以实现正则化,我们会集中讨论岭回归和Lasso回归,最后讨论弹性网络,它将前面两种算法的优点合二为一。
2.正则化简介
线性模型形式为Y=B0+B1
x
1+…+Bn
x
n+e,最佳拟合试图最小化RSS。RSS是实际值减去估计值的差的平方和,可以表示为
e1^
2+e2^
2+…+en^2
。通过正则化,我们会在RSS的最小化过程中加入一个新参数,称之为收缩惩罚项。这个惩罚项包含了一个希腊字母λ以及对β系数和权重的规范化结果。不同的技术对权重的规范化方法都不尽相同。简言之,我们在模型用RSS+λ(规范化后的系数)代替RSS。我们对λ进行选择,在模型构建过程中,λ被称为调优参数。如果λ = 0,模型就等价于OLS,因为规范化项目都被抵消。
那么,正则化对我们来说意味着什么?首先,正则化方法在计算上非常有效。如果使用最优子集法,我们需要在一个大数据集上测试2^p个模型,这肯定是不可行的。如果使用正则化方法,对于每个λ值,我们只需拟合一个模型,因此效率会有极大提升。其次,是偏差/方差权衡问题。在线性模型中,响应变量和预测变量之间的关系接近于线性,最小二乘估计接近于无偏,但可能有很高的方差。这意味着,训练集中的微小变动会导致最小二乘系数估计结果的巨大变动(James,2013)。正则化通过恰当地选择λ和规范化,可以使偏差/方差权衡达到最优,从而提高模型拟合的效果。最后,系数的正则化还可以用来解决多重共线性引起的过拟合问题。
3.岭回归
我们先研究什么是岭回归,以及它可以做什么和不能做什么。在岭回归中,规范化项是所有系数的平方和,称为L2-norm(L2范数)。在我们的模型中就是试图最小化RSS+λ(sumβj^2)。当λ增加时,系数会缩小,趋向于0但永远不会为0。岭回归的优点是可以提高预测准确度,但因为它不能使任何一个特征的系数为0,所以在模型解释性上会有些问题。为了解决这个问题,我们使用LASSO回归。
4.LASSO回归
区别于岭回归中的L2-norm,LASSO回归使用L1-norm,即所有特征权重的绝对值之和,也就是要最小化RSS+λ(sum|βj|)。这个收缩惩罚项确实可以使特征权重收缩到0,相对于岭回归,这是一个明显的优势,因为可以极大地提高模型的解释性。如果LASSO这么好,那还要岭回归做什么?当存在高度共线性或高度两两相关的情况下,LASSO回归可能会将某个预测特征强制删除,这会损失模型的预测能力。举例来说,如果特征A和B都应该存在于模型之中,那么LASSO可能会将其中一个的系数缩减到0。可见岭回归与Lasso回归应该是互为补充的关系。
5.弹性网络
弹性网络的优势在于,它既能做到岭回归不能做的特征提取,又能实现LASSO不能做的特征分组。重申一下,LASSO倾向于在一组相关的特征中选择一个,忽略其他。弹性网络包含了一个混合参数α,它和λ同时起作用。α是一个0和1之间的数,λ和前面一样,用来调节惩罚项的大小。请注意,当α等于0时,弹性网络等价于岭回归;当α等于1时,弹性网络等价于LASSO。实质上,我们通过对β系数的二次项引入一个第二调优参数,将L1惩罚项和L2惩罚项混合在一起。通过最小化(RSS + λ[(1 - α)(sum|βj|^2)/2 + α(sum|βj|)]/N)完成目标。
下面我们就使用leaps、glmnet和caret包在下面的案例中选择合适的特征并生成合适的模型。
6.案例解析
本章案例是一个前列腺癌数据。虽然这个数据集比较小,只有97个观测和9个变量,但通过与传统技术的比较,足以让我们掌握正则化技术。斯坦福大学医疗中心提供了97个病人的前列腺特异性抗原(PSA)数据,这些病人均接受前列腺根治术。我们的目标是,通过临床检测提供的数据建立一个预测模型预测患者术后PSA水平。对于患者在手术后能够或应该恢复到什么程度,与其他指标相比,PSA可能是一个更有效的预后指标。手术之后,医生会在各个时间区间检查患者的PSA水平,并通过各种公式确定患者是否康复。术前预测模型和术后数据(这里没有提供)互相配合,就可能提高前列腺癌诊疗水平,改善其预后。
收集自97位男性的数据集保存在一个含10个变量的数据框中,如下所示:
lcavol:肿瘤体积的对数值 lweight:前列腺重量的对数值 age:患者年龄(以年计) lbph:良性前列腺增生(BPH)量的对数值,非癌症性质的前列腺增生。 svi:精囊是否受侵,一个指标变量,表示癌细胞是否已经透过前列腺壁侵入精囊腺(1=是,0=否)。 lcp:包膜穿透度的对数值,表示癌细胞扩散到前列腺包膜之外的程度。 gleason:患者的Gleason评分;由病理学家进行活体检查后给出(2~10),表示癌细胞的变异程度——评分越高,程度越危险。 pgg45:Gleason评分为4或5所占的百分比。 lpsa:PSA值的对数值,响应变量。 train:一个逻辑向量(TRUE或FALSE,用来区分训练数据和测试数据)。
这个数据集包含在ElemStatLearn这个R包内。加载所需的程序包和数据框之后,查看变量以及变量之间可能存在的联系,如下所示:
library(ElemStatLearn) #contains the data
library(car) #package to calculate Variance Inflation Factor
## Loading required package: carData
library(corrplot) #correlation plots
## corrplot 0.84 loaded
library(leaps) #best subsets regression
library(glmnet) #allows ridge regression, LASSO and elastic net
## Loading required package: Matrix
## Loading required package: foreach
## Loaded glmnet 2.0-16
library(caret) #this will help identify the appropriate parameters
## Loading required package: lattice
## Loading required package: ggplot2
加载程序包之后,调出prostate数据集,查看数据结构,如下所示:
data(prostate)
str(prostate)
## 'data.frame': 97 obs. of 10 variables:
## $ lcavol : num -0.58 -0.994 -0.511 -1.204 0.751 ...
## $ lweight: num 2.77 3.32 2.69 3.28 3.43 ...
## $ age : int 50 58 74 58 62 50 64 58 47 63 ...
## $ lbph : num -1.39 -1.39 -1.39 -1.39 -1.39 ...
## $ svi : int 0 0 0 0 0 0 0 0 0 0 ...
## $ lcp : num -1.39 -1.39 -1.39 -1.39 -1.39 ...
## $ gleason: int 6 6 7 6 6 6 6 6 6 6 ...
## $ pgg45 : int 0 0 20 0 0 0 0 0 0 0 ...
## $ lpsa : num -0.431 -0.163 -0.163 -0.163 0.372 ...
## $ train : logi TRUE TRUE TRUE TRUE TRUE TRUE ...
检查数据结构会发现几个问题,需要特别注意。检查数据集的特征就会发现,svi、lcp、gleason和pgg45的前10个观测值都具有相同的数值,只有一个例外——gleason的第三个观测值。为了保证这些特征作为输入特征是确实可行的,我们可以做出统计图或表格来理解数据。使用下面的plot()命令,将整个数据框作为输入值,即可建立散点图矩阵,如下所示:
plot(prostate)
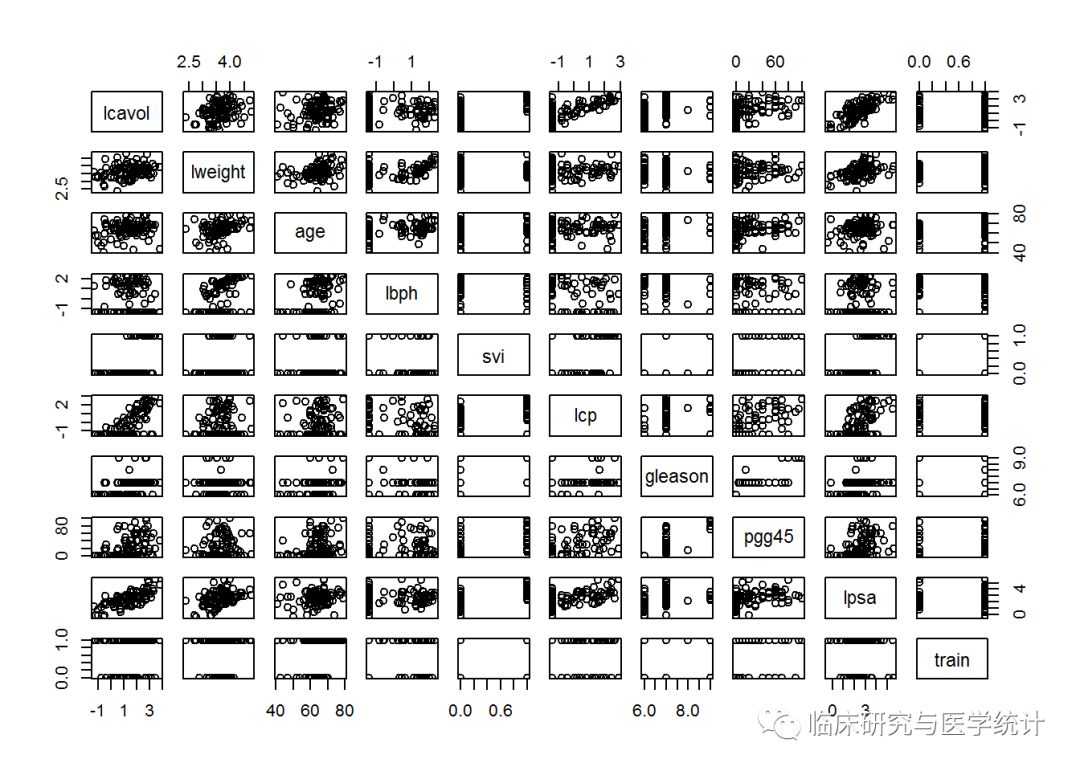
这么多变量放在一张图上确实有点让人搞不清状况,所以我们循序渐进。可以看出,结果变量lpsa和预测变量lcavol之间确实存在明显的线性关系。还可以看出,这些特征变量的离散度是比较合适的,而且在训练集和测试集之间的分布也比较平衡,可能的例外只有Gleason评分这个特征。请注意,这个数据集中,Gleason评分只有4个值。如果看一下位于train和gleason相交点的那个图,就会发现,有一个Gleason评分值既不属于测试集,也不输入训练集。这可能会使我们的分析过程产生问题,需要进行数据转换。所以,我们专门为这个特征建立一个统计图,如下所示:
plot(prostate$gleason, ylab = "Gleason Score")
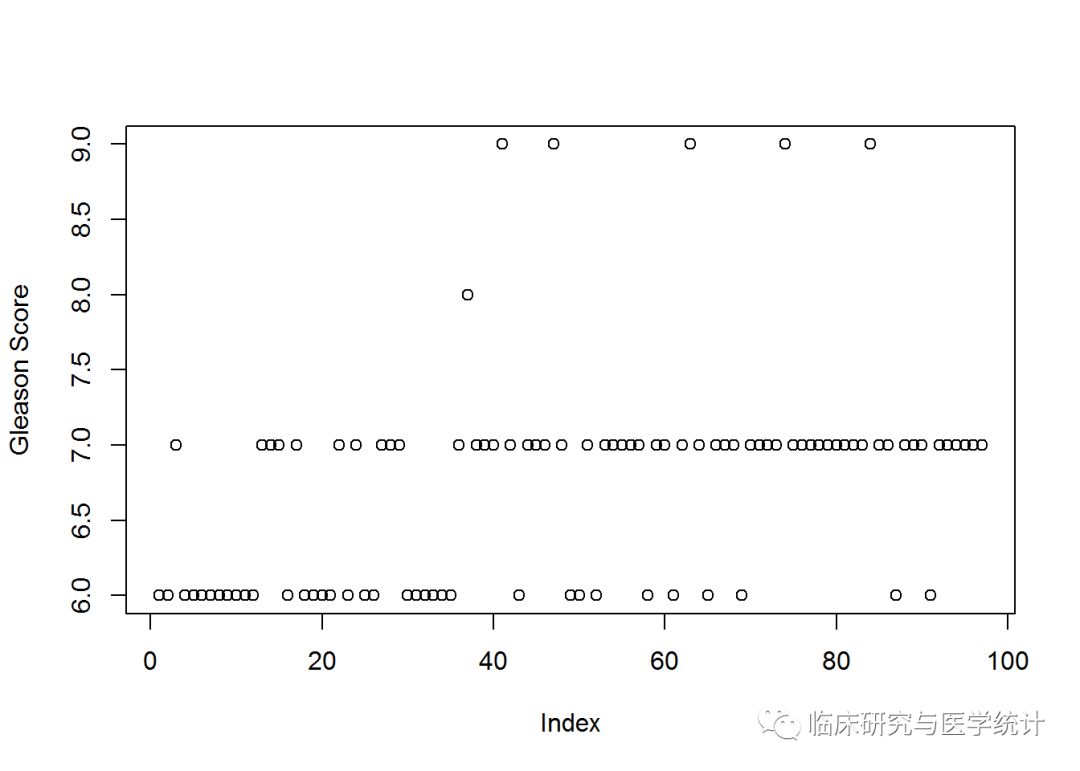
现在就看出问题了。每个点代表一个观测,X轴是数据框中观测的编号。只有1个观测的Gleason评分是8.0,只有5个观测的Gleason评分是9.0。你可以建立一个特征值表格来查看确切的数量。如下所示:
table(prostate$gleason)
##
## 6 7 8 9
## 35 56 1 5
怎么办呢?我们有以下3种选择:
(1)完全删除这个特征; (2)仅删除值为8.0和9.0的那些评分; (3)对特征重新编码,建立一个指标变量。
如果建立一个横轴为Gleason Score,纵轴为Log of PSA 的箱线图,会对我们的选择有所帮助。如下所示:
boxplot(prostate$lpsa ~ prostate$gleason, xlab = "Gleason Score",
ylab = "Log of PSA")
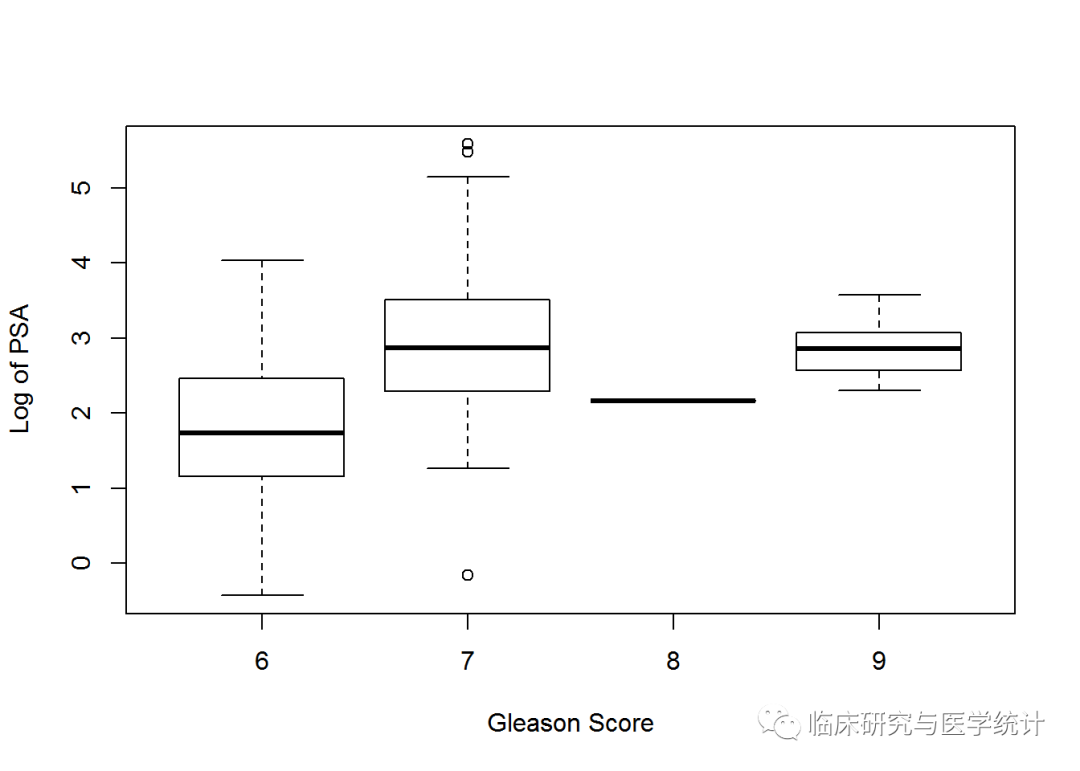
看了上面的图,最好的选择是将这个特征转换为一个二分类变量,0表示评分为6,1表示评分为7或更高。删除特征可能会损失模型的预测能力。缺失值也可能会在我们将要使用的glmnet包中引起问题。你可以非常简单地实现对指标变量的编码,通过一行代码即可。使用ifelse()命令,指定你想在数据框中转换的列,然后按照这个规则转换:如果观测值的特征值为x,则将其编码为y,否则将其编码为z。
prostate$gleason 6, 0, 1)
table(prostate$gleason)
##
## 0 1
## 35 62
散点图矩阵比较难以理解,我们可以试试相关性统计图,它可以表示特征之间是否存在相关性或依赖。先用cor()函数建立一个相关性对象,然后利用corrplot包中的corrplot.mixed()函数做出相关性统计图,如下所示:
p.cor = cor(prostate)
corrplot.mixed(p.cor)
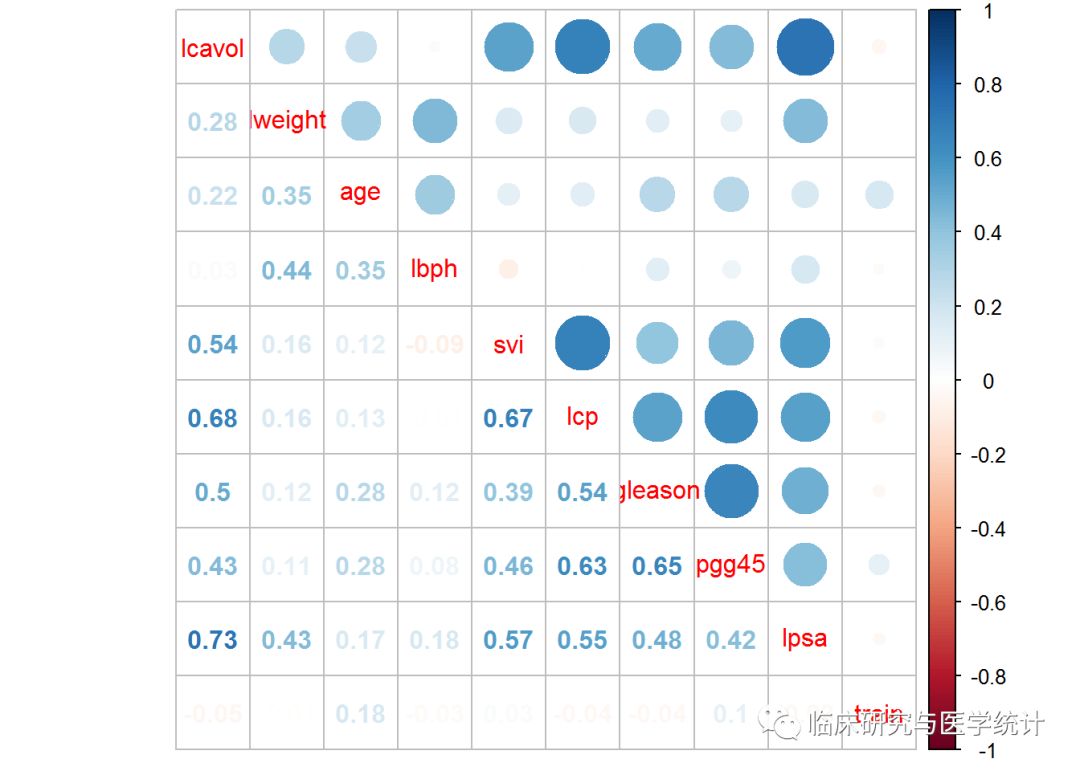
这样又有两个新问题。首先,PSA和肿瘤体积的对数(lcavol)高度相关。回想一下,在散点图矩阵中,它们表现出很强的线性相关关系。其次,多重共线性是个问题。例如,肿瘤体积还与包膜穿透相关,而包膜穿透还与精囊是否受侵相关。
首先建立训练数据集和测试数据集。既然观测值中已经有一个特征指明这个观测值是否属于训练集,我们就可以使用subset()命令将train值为TRUE的观测值分到训练集中,将train值为FALSE的观测值分到测试集。将train特征删除也是必须的,因为我们不想把它作为预测特征。如下所示:
train TRUE)[, 1:9]
str(train)
## 'data.frame': 67 obs. of 9 variables:
## $ lcavol : num -0.58 -0.994 -0.511 -1.204 0.751 ...
## $ lweight: num 2.77 3.32 2.69 3.28 3.43 ...
## $ age : int 50 58 74 58 62 50 58 65 63 63 ...
## $ lbph : num -1.39 -1.39 -1.39 -1.39 -1.39 ...
## $ svi : int 0 0 0 0 0 0 0 0 0 0 ...
## $ lcp : num -1.39 -1.39 -1.39 -1.39 -1.39 ...
## $ gleason: num 0 0 1 0 0 0 0 0 0 1 ...
## $ pgg45 : int 0 0 20 0 0 0 0 0 0 30 ...
## $ lpsa : num -0.431 -0.163 -0.163 -0.163 0.372 ...
test = subset(prostate, train==FALSE
)[,1:9]
str(test)
## 'data.frame': 30 obs. of 9 variables:
## $ lcavol : num 0.737 -0.777 0.223 1.206 2.059 ...
## $ lweight: num 3.47 3.54 3.24 3.44 3.5 ...
## $ age : int 64 47 63 57 60 69 68 67 65 54 ...
## $ lbph : num 0.615 -1.386 -1.386 -1.386 1.475 ...
## $ svi : int 0 0 0 0 0 0 0 0 0 0 ...
## $ lcp : num -1.386 -1.386 -1.386 -0.431 1.348 ...
## $ gleason: num 0 0 0 1 1 0 0 1 0 0 ...
## $ pgg45 : int 0 0 0 5 20 0 0 20 0 0 ...
## $ lpsa : num 0.765 1.047 1.047 1.399 1.658 ...
7.模型构建与评价
数据已经准备好,下面我们将开始构建模型。为了进行对比,先用最优子集回归建立一个模型,然后使用正则化技术建立模型。
通过regsubsets()命令建立一个最小子集对象,然后指定训练数据集。选择出的特征随后用在测试集上,通过计算均方误差来评价模型。我们建立模型的语法为lpsa~.,使用“~.”说明,要使用数据框中除响应变量之外的所有变量进行预测。如下所示:
subfit
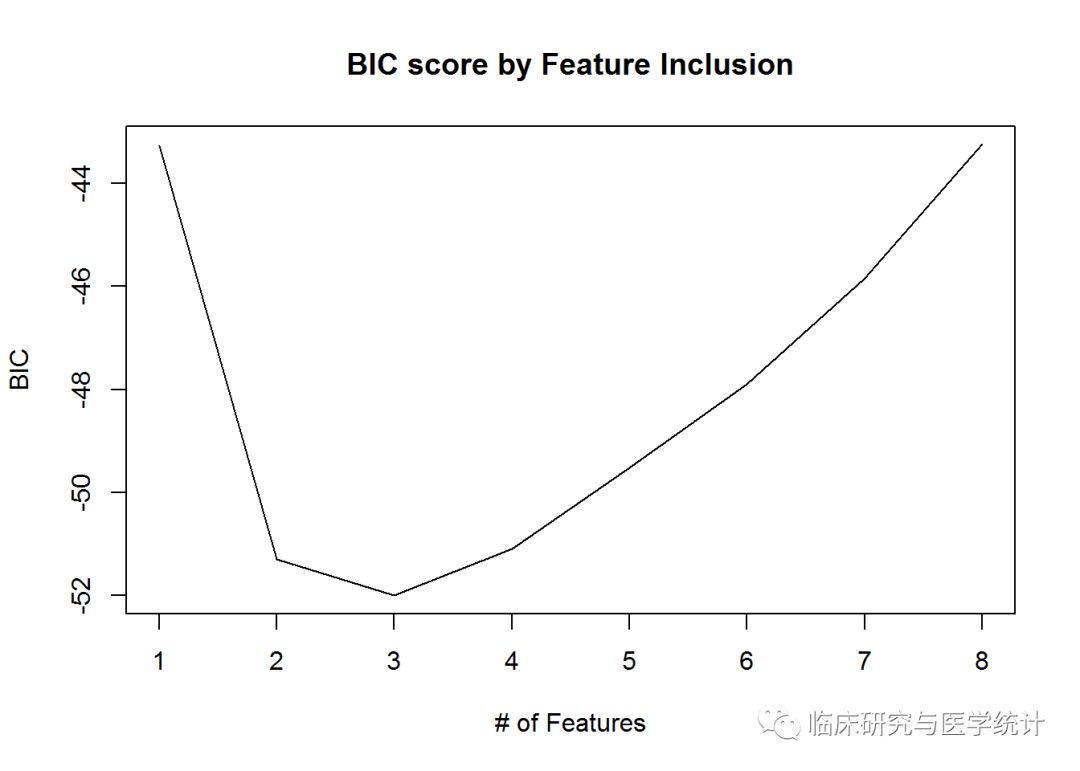
模型建立之后,你可以通过两行代码得到最优子集。第一行代码将subfit模型摘要写入一个新对象b.sum,然后从这个对象提取各个子集,使用which.min()命令确定最优子集。在本例中,我们使用第2章中讨论过的贝叶斯信息准则,如下所示:
b.sum
## [1] 3
结果告诉我们,具有三个特征模型具有最小的BIC值。可以通过一个统计图查看模型性能和子集组合之间的关系,如下所示:
plot(b.sum$bic, type = "l", xlab = "# of Features", ylab = "BIC",
main = "BIC score by Feature Inclusion")
对实际模型做出统计图,可以让我们进行更详细的检查。如下所示:
plot(subfit, scale = "bic", main = "Best Subset Features")
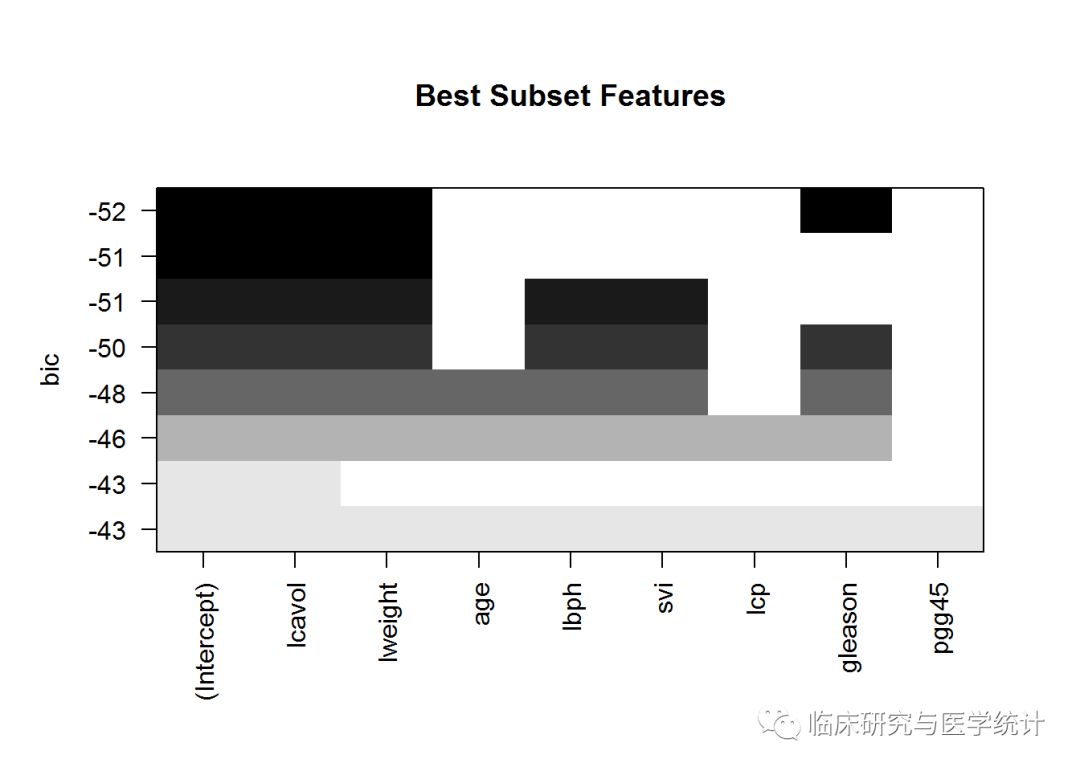
于是,上图告诉我们具有最小BIC值的模型中的3个特征是:lcavol、lweight和gleason。值得注意的是,lcavol包含在所有模型组合之中,这与我们之前的数据探索结果是一致的。现在,可以在测试集上试验模型了,但要先用模型的拟合值与实际值画一张图,看看最终解中的线性关系,并检查同方差性。需要用以上3个变量建立一个线性模型,因为是用OLS建立的模型,所以把它存储在名为ols的对象中。然后,使用OLS拟合出的模型就可以同训练集中的实际值进行对比了。如下所示:
ols "Predicted", ylab = "Actual",
main = "Predicted vs Actual")
abline(0,1,col=2)
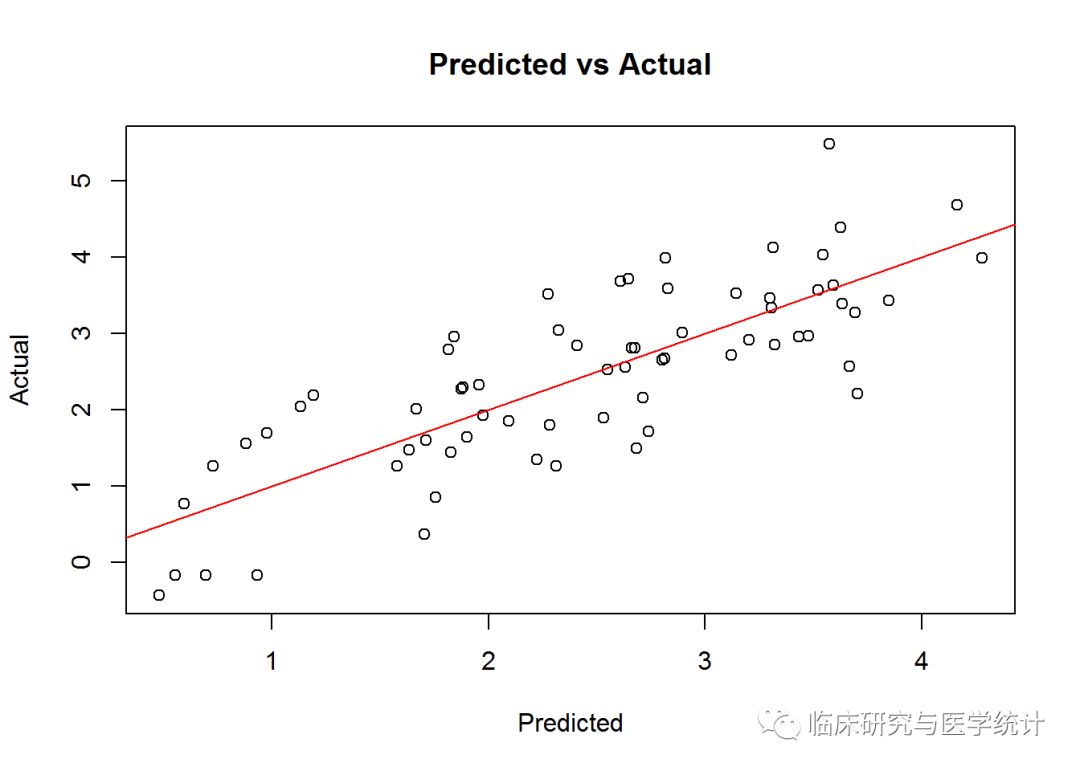
从图中可以看出,在训练集上线性拟合表现得很好,也不存在异方差性。然后看看模型在测试集上的表现,使用predict()函数并指定newdata = test,如下所示:
pred.subfit = predict(ols, newdata=test)
plot(pred.subfit, test$lpsa , xlab = "Predicted",
ylab = "Actual", main = "Predicted vs Actual")
abline(0,1,col=2)
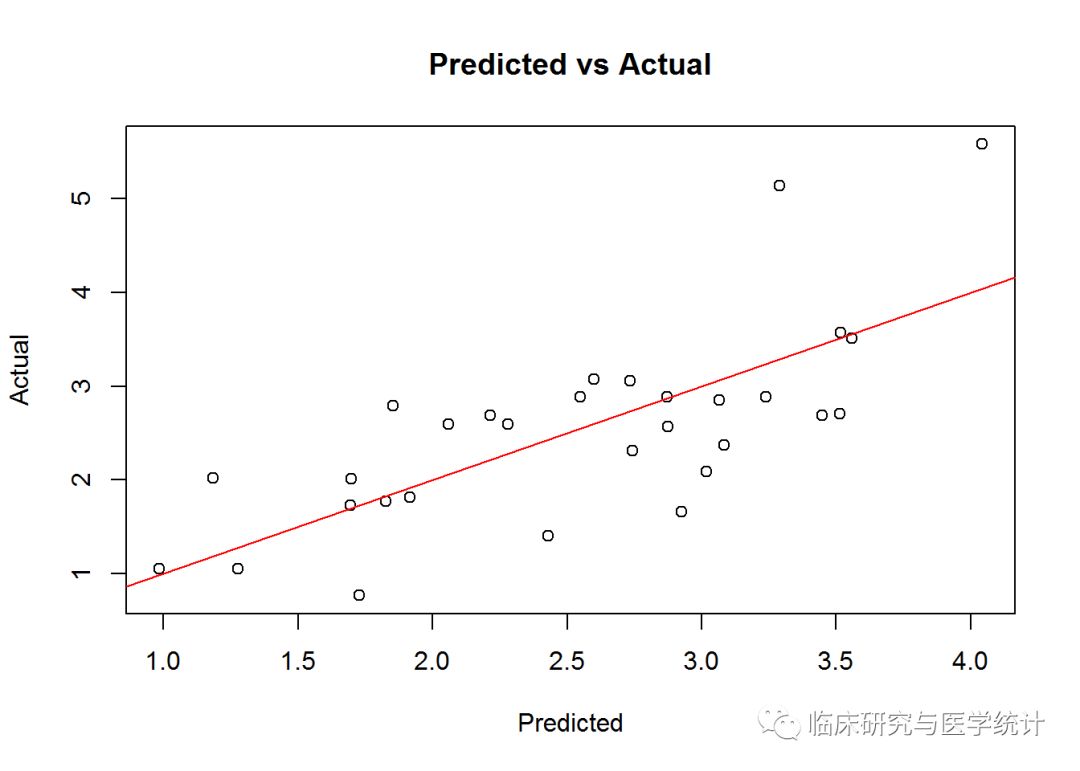
这个图还不是太难看。总体来说,图中呈现一种线性关系,只不过当PSA值比较高时,有两个离群点。ranhou 我们再计算均方误差,以便在不同模型构建技术之间进行比较。这非常简单,只要算出残差并求出残差平方的均值即可,如下所示:
resid.subfit = test$lpsa - pred.subfit
mean(resid.subfit^2
)
## [1] 0.5084126
MSE值为0.508,以此为参照我们继续下面的内容。
8.岭回归建模
在岭回归中,我们的模型会包括全部8个特征,所以岭回归模型与最优子集模型的比较令人期待。我们要使用的程序包glmnet。这个程序包要求输入特征存储在矩阵中,而不是在数据框中。岭回归的命令形式为glmnet(x=输入矩阵,y=响应变量,family=分布函数,alpha=0)。这里的alpha为0时,表示进行岭回归;alpha为1时,表示进行LASSO回归。要准备好供glmnet使用的训练集数据也很容易,使用as.matrix()函数处理输入数据,并建立一个向量作为响应变量,如下所示:
x 1:8])
y 9]
现在可以使用岭回归了,我们把结果保存在一个对象中,可以为对象起一个恰当的名字,比如ridge。这里有一点非常重要,请一定注意:glmnet包会在计算λ值之前首先对输入进行标准化,然后计算非标准化系数。你需要指定响应变量的分布为gaussian,因为它是连续的;还要指定alpha = 0,表示进行岭回归。如下所示:
ridge "gaussian", alpha = 0)
这个对象包含了我们进行模型评价所需的所有信息。首先尝试print()命令,它会展示非0系数的数量,解释偏差百分比以及相应的λ值。程序包中算法默认的计算次数是100,但如果偏差百分比在两个λ值之间的提高不是很显著的话,算法会在100次计算之前停止。也就是说,算法收敛于最优解。为了节省篇幅,我只在下面列出了前5个和后10个λ结果:
print(ridge)
##
## Call: glmnet(x = x, y = y, family = "gaussian", alpha = 0)
##
## Df %Dev Lambda
## [1,] 8 3.801e-36 878.90000
## [2,] 8 5.591e-03 800.80000
## [3,] 8 6.132e-03 729.70000
## [4,] 8 6.725e-03 664.80000
## [5,] 8 7.374e-03 605.80000
## [6,] 8 8.086e-03 552.00000
## [7,] 8 8.865e-03 502.90000
## [8,] 8 9.718e-03 458.20000
## [9,] 8 1.065e-02 417.50000
## [10,] 8 1.168e-02 380.40000
## [11,] 8 1.279e-02 346.60000
## [12,] 8 1.402e-02 315.90000
## [13,] 8 1.536e-02 287.80000
## [14,] 8 1.682e-02 262.20000
## [15,] 8 1.842e-02 238.90000
## [16,] 8 2.017e-02 217.70000
## [17,] 8 2.208e-02 198.40000
## [18,] 8 2.417e-02 180.70000
## [19,] 8 2.644e-02 164.70000
## [20,] 8 2.892e-02 150.10000
## [21,] 8 3.163e-02 136.70000
## [22,] 8 3.457e-02 124.60000
## [23,] 8 3.777e-02 113.50000
## [24,] 8 4.126e-02 103.40000
## [25,] 8 4.504e-02 94.24000
## [26,] 8 4.915e-02 85.87000
## [27,] 8 5.360e-02 78.24000
## [28,] 8 5.842e-02 71.29000
## [29,] 8 6.364e-02 64.96000
## [30,] 8 6.928e-02 59.19000
## [31,] 8 7.536e-02 53.93000
## [32,] 8 8.191e-02 49.14000
## [33,] 8 8.896e-02 44.77000
## [34,] 8 9.652e-02 40.79000
## [35,] 8 1.046e-01 37.17000
## [36,] 8 1.133e-01 33.87000
## [37,] 8 1.225e-01 30.86000
## [38,] 8 1.324e-01 28.12000
## [39,] 8 1.428e-01 25.62000
## [40,] 8 1.539e-01 23.34000
## [41,] 8 1.655e-01 21.27000
## [42,] 8 1.778e-01 19.38000
## [43,] 8 1.907e-01 17.66000
## [44,] 8 2.041e-01 16.09000
## [45,] 8 2.181e-01 14.66000
## [46,] 8 2.327e-01 13.36000
## [47,] 8 2.477e-01 12.17000
## [48,] 8 2.631e-01 11.09000
## [49,] 8 2.790e-01 10.10000
## [50,] 8 2.951e-01 9.20700
## [51,] 8 3.115e-01 8.38900
## [52,] 8 3.281e-01 7.64400
## [53,] 8 3.447e-01 6.96500
## [54,] 8 3.614e-01 6.34600
## [55,] 8 3.780e-01 5.78200
## [56,] 8 3.945e-01 5.26900
## [57,] 8 4.108e-01 4.80100
## [58,] 8 4.268e-01 4.37400
## [59,] 8 4.424e-01 3.98600
## [60,] 8 4.576e-01 3.63200
## [61,] 8 4.724e-01 3.30900
## [62,] 8 4.866e-01 3.01500
## [63,] 8 5.003e-01 2.74700
## [64,] 8 5.134e-01 2.50300
## [65,] 8 5.260e-01 2.28100
## [66,] 8 5.380e-01 2.07800
## [67,] 8 5.493e-01 1.89300
## [68,] 8 5.601e-01 1.72500
## [69,] 8 5.703e-01 1.57200
## [70,] 8 5.800e-01 1.43200
## [71,] 8 5.891e-01 1.30500
## [72,] 8 5.976e-01 1.18900
## [73,] 8 6.057e-01 1.08400
## [74,] 8 6.133e-01 0.98730
## [75,] 8 6.204e-01 0.89960
## [76,] 8 6.270e-01 0.81960
## [77,] 8 6.333e-01 0.74680
## [78,] 8 6.391e-01 0.68050
## [79,] 8 6.445e-01 0.62000
## [80,] 8 6.496e-01 0.56500
## [81,] 8 6.543e-01 0.51480
## [82,] 8 6.587e-01 0.46900
## [83,] 8 6.628e-01 0.42740
## [84,] 8 6.666e-01 0.38940
## [85,] 8 6.701e-01 0.35480
## [86,] 8 6.733e-01 0.32330
## [87,] 8 6.763e-01 0.29460
## [88,] 8 6.790e-01 0.26840
## [89,] 8 6.815e-01 0.24460
## [90,] 8 6.838e-01 0.22280
## [91,] 8 6.859e-01 0.20300
## [92,] 8 6.877e-01 0.18500
## [93,] 8 6.894e-01 0.16860
## [94,] 8 6.909e-01 0.15360
## [95,] 8 6.923e-01 0.13990
## [96,] 8 6.935e-01 0.12750
## [97,] 8 6.946e-01 0.11620
## [98,] 8 6.955e-01 0.10590
## [99,] 8 6.964e-01 0.09646
## [100,] 8 6.971e-01 0.08789
以第100行为例。可以看出非0系数,即模型中包含的特征的数量为8。请记住,在岭回归中,这个数是不变的。还可以看出解释偏差百分比为0.6971,以及这一行的调优系数λ的值为0.08789。此处即可决定选择在测试集上使用哪个λ。这个λ值应该是0.08789,但是为了简单起见,在测试集上可以试一下0.10。此时,一些统计图是非常有用的。我们先看看程序包中默认的统计图,设定label = TRUE可以给曲线加上注释,如下所示:
plot(ridge, label = TRUE)
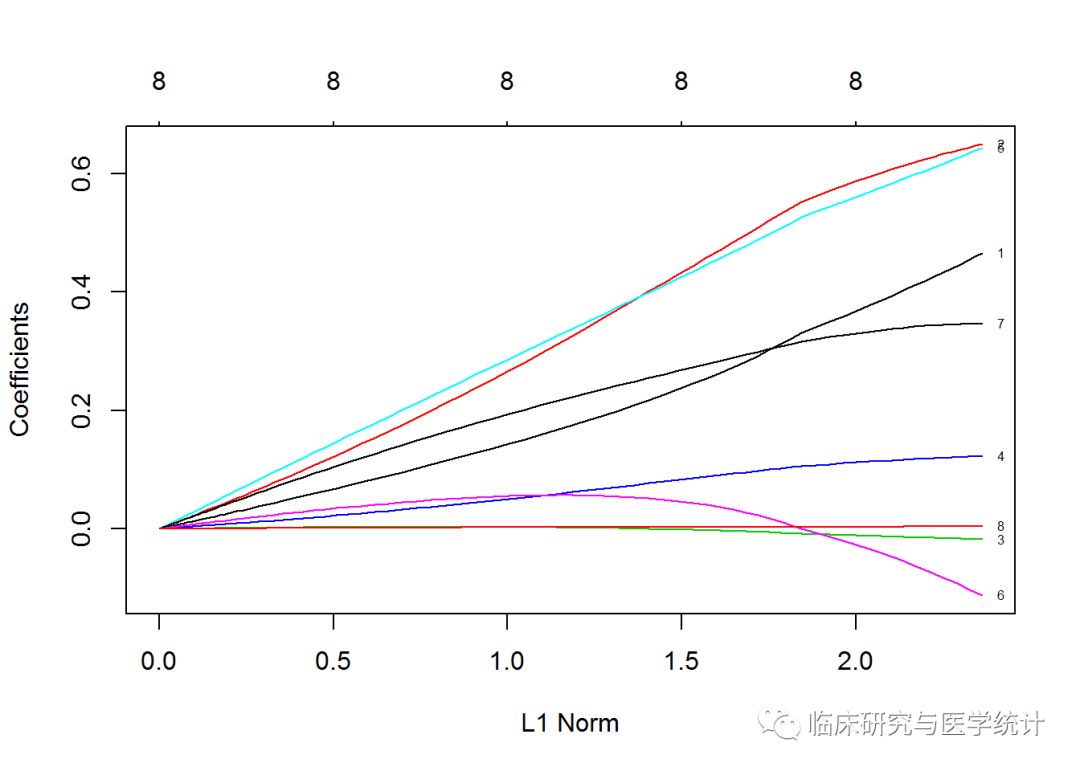
在默认图中,Y轴是回归系数值,X轴是L1范数,图中显示了系数值和L1范数之间的关系。图的上方有另一条X轴,其上的数值表示模型中的特征数。也可以看看系数值如何随着λ的变化而变化。只需在plot()命令中稍稍调整,加上参数xvar=“lambda”即可。另一种选择是,看系数值如何随解释偏差百分比变化,将lamda换成dev即可。
plot(ridge, xvar = "lambda", label = TRUE)
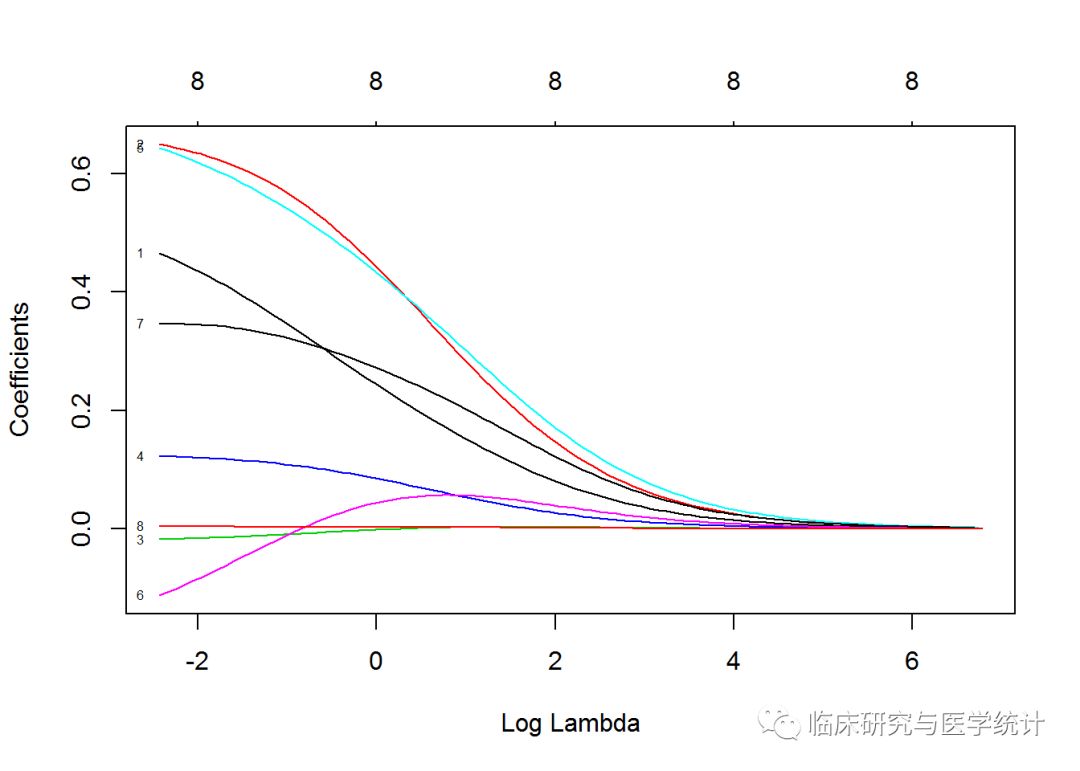
这张图非常有价值,因为它表明,当λ值减小时,压缩参数随之减小,而系数绝对值随之增大。要想看看当λ为一个特定值时的系数值,可以使用predict()命令。现在,看一下当λ为0.1时,系数值是多少。指定参数s=0.1,同时指定参数type = “coefficients”,告诉glmnet在拟合模型时使用具体的λ值,而不是从λ值的两侧选值插入。如下所示:
ridge.coef 0.1, type = "coefficients")
ridge.coef
## 9 x 1 sparse Matrix of class "dgCMatrix"
## 1
## (Intercept) 0.130475478
## lcavol 0.457279371
## lweight 0.645792042
## age -0.017356156
## lbph 0.122497573
## svi 0.636779442
## lcp -0.104712451
## gleason 0.346022979
## pgg45 0.004287179
需要特别注意的是:age、lcp和pgg45的系数非常接近0,但还不是0。别忘了再看一下偏差与系数之间的关系图:
plot(ridge, xvar = "dev", label = TRUE)
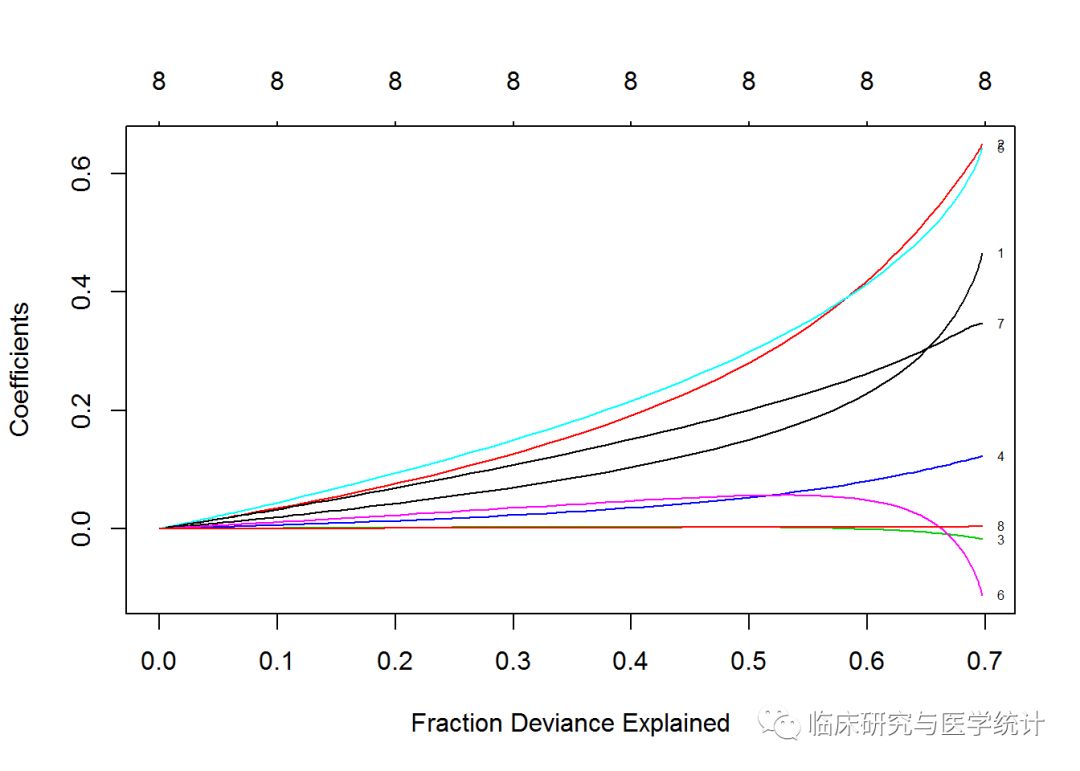
同前两张图相比,我们可以从这张图中看出,当λ减小时,系数会增大,解释偏差百分比也会增大。如果将λ值设为0,就会忽略收缩惩罚,模型将等价于OLS。为了在测试集上证明这一点,需要转换特征,像我们在训练集上做的一样:
newx 1:8])
然后,使用predict()函数建立一个名为ridge.y的对象,指定参数type=“response”以及λ值为0.10,画出表示预测值和实际值关系的统计图,如下所示:
ridge.y = predict(ridge, newx = newx, type = "response", s=0.1)
plot(ridge.y, test$lpsa, xlab = "Predicted",
ylab = "Actual", main = "Ridge Regression")
abline(1,0.1,col=2)
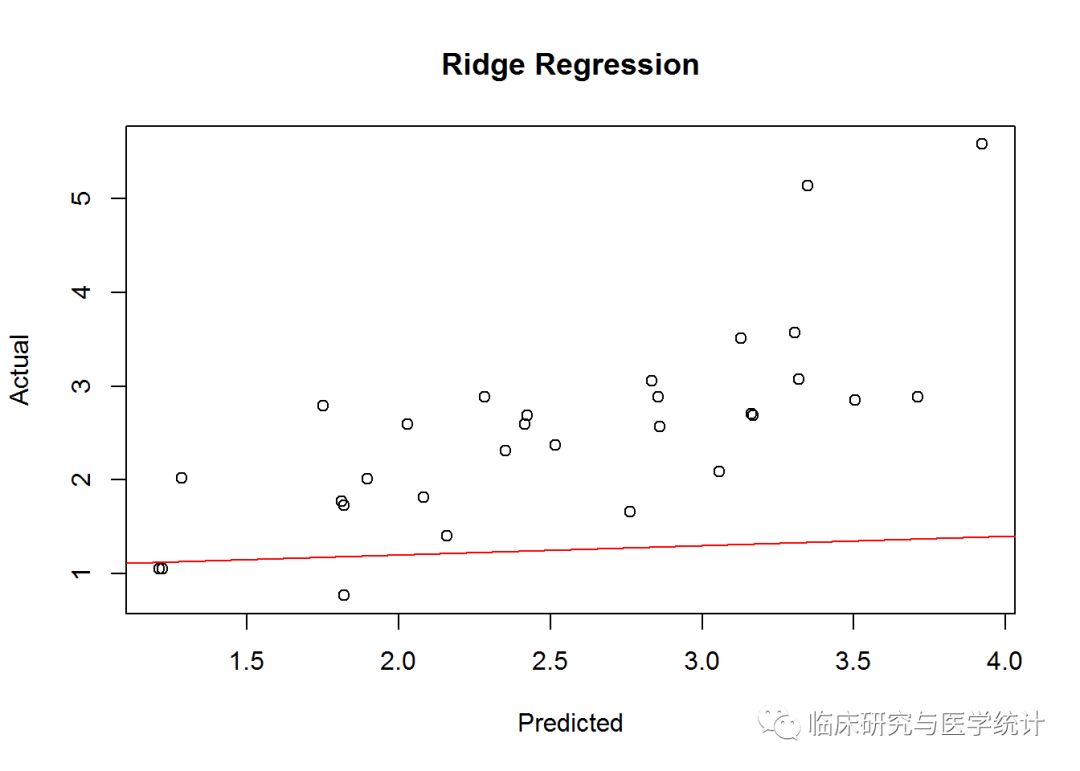
表示岭回归中预测值和实际值关系的统计图看上去与最优子集的非常相似,同样地,在PSA测量结果比较大的一端有两个有趣的离群点。在实际情况下,我建议对离群点进行更深入的研究,搞清楚是它们真的与众不同,还是我们忽略了什么。与MSE基准的比较可能告诉我们一些不同的事。先算出残差,然后算出残差平方的平均值:
ridge.resid 2)
## [1] 0.4783559
岭回归给出的MSE稍好一点。现在是时候检验LASSO了,看看我们能否将误差再减少一些。
9.LASSO回归建模
下面运行LASSO就非常简单了,只要改变岭回归模型的一个参数即可。也就是说,在glmnet()语法中将岭回归中的alpha=0变为alpha=1。运行代码,看看模型的输出,检查前5个和后10个拟合结果:
lasso "gaussian", alpha = 1)
print(lasso)
##
## Call: glmnet(x = x, y = y, family = "gaussian", alpha = 1)
##
## Df %Dev Lambda
## [1,] 0 0.00000 0.878900
## [2,] 1 0.09126 0.800800
## [3,] 1 0.16700 0.729700
## [4,] 1 0.22990 0.664800
## [5,] 1 0.28220 0.605800
## [6,] 1 0.32550 0.552000
## [7,] 1 0.36150 0.502900
## [8,] 1 0.39140 0.458200
## [9,] 2 0.42810 0.417500
## [10,] 2 0.45980 0.380400
## [11,] 3 0.48770 0.346600
## [12,] 3 0.51310 0.315900
## [13,] 4 0.53490 0.287800
## [14,] 4 0.55570 0.262200
## [15,] 4 0.57300 0.238900
## [16,] 4 0.58740 0.217700
## [17,] 4 0.59930 0.198400
## [18,] 5 0.61170 0.180700
## [19,] 5 0.62200 0.164700
## [20,] 5 0.63050 0.150100
## [21,] 5 0.63760 0.136700
## [22,] 5 0.64350 0.124600
## [23,] 5 0.64840 0.113500
## [24,] 5 0.65240 0.103400
## [25,] 6 0.65580 0.094240
## [26,] 6 0.65870 0.085870
## [27,] 6 0.66110 0.078240
## [28,] 6 0.66310 0.071290
## [29,] 7 0.66630 0.064960
## [30,] 7 0.66960 0.059190
## [31,] 7 0.67240 0.053930
## [32,] 7 0.67460 0.049140
## [33,] 7 0.67650 0.044770
## [34,] 8 0.67970 0.040790
## [35,] 8 0.68340 0.037170
## [36,] 8 0.68660 0.033870
## [37,] 8 0.68920 0.030860
## [38,] 8 0.69130 0.028120
## [39,] 8 0.69310 0.025620
## [40,] 8 0.69460 0.023340
## [41,] 8 0.69580 0.021270
## [42,] 8 0.69680 0.019380
## [43,] 8 0.69770 0.017660
## [44,] 8 0.69840 0.016090
## [45,] 8 0.69900 0.014660
## [46,] 8 0.69950 0.013360
## [47,] 8 0.69990 0.012170
## [48,] 8 0.70020 0.011090
## [49,] 8 0.70050 0.010100
## [50,] 8 0.70070 0.009207
## [51,] 8 0.70090 0.008389
## [52,] 8 0.70110 0.007644
## [53,] 8 0.70120 0.006965
## [54,] 8 0.70130 0.006346
## [55,] 8 0.70140 0.005782
## [56,] 8 0.70150 0.005269
## [57,] 8 0.70150 0.004801
## [58,] 8 0.70160 0.004374
## [59,] 8 0.70160 0.003986
## [60,] 8 0.70170 0.003632
## [61,] 8 0.70170 0.003309
## [62,] 8 0.70170 0.003015
## [63,] 8 0.70170 0.002747
## [64,] 8 0.70180 0.002503
## [65,] 8 0.70180 0.002281
## [66,] 8 0.70180 0.002078
## [67,] 8 0.70180 0.001893
## [68,] 8 0.70180 0.001725
## [69,] 8 0.70180 0.001572
请注意,模型构建过程在69步之后停止了,因为解释偏差不再随着λ值的增加而减小。还要注意,Df列现在也随着λ变化。初看上去,当λ值为0.001572时,所有8个特征都应该包括在模型中。然而,出于测试的目的,我们先用更少特征的模型进行测试,比如7特征模型。从下面的结果行中可以看到,λ值大约为0.045时,模型从7个特征变为8个特征。因此,使用测试集评价模型时要使用这个λ值。如下所示:
[31,] 7 0.67240 0.053930 [32,] 7 0.67460 0.049140 [33,] 7 0.67650 0.044770 [34,] 8 0.67970 0.040790 [35,] 8 0.68340 0.037170
和岭回归一样,可以在图中画出结果。如下所示:
plot(lasso, xvar = "lambda", label = TRUE)
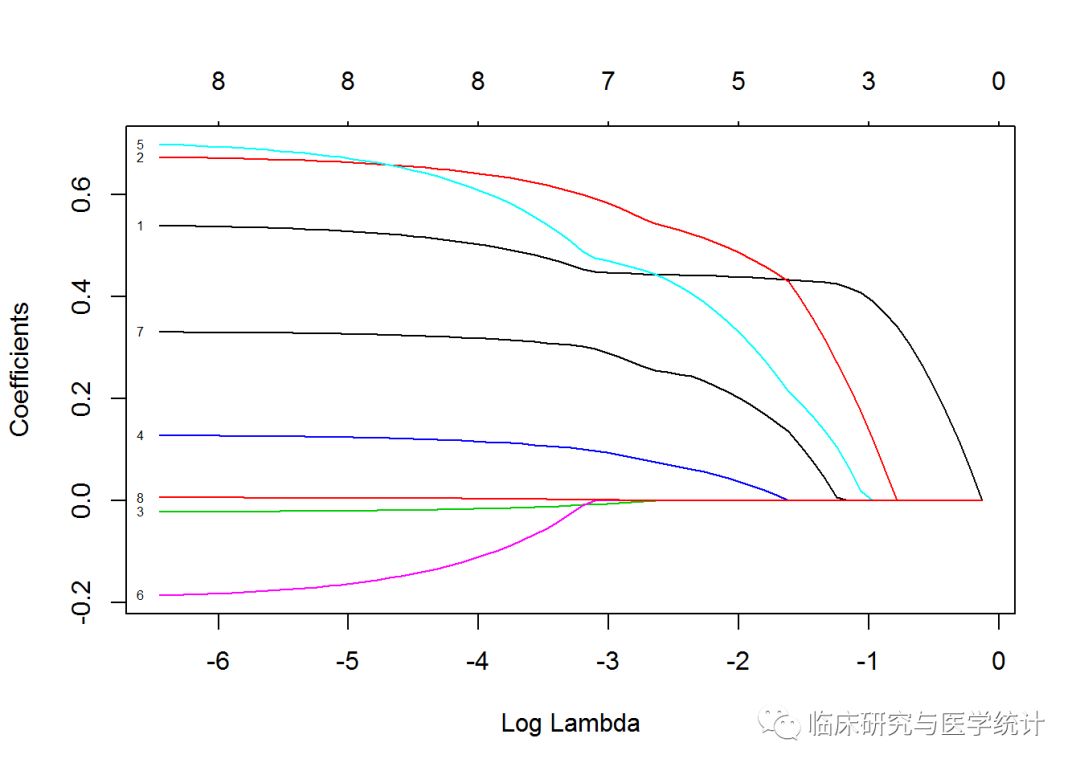
这张图展示了LASSO是如何工作的。请注意观察标号为8、3和6的曲线的表现,这几条曲线分别对应特征pgg45、age和lcp。看上去lcp一直接近于0,直到作为最后一个特征被加入模型。可以通过与岭回归中同样的操作看看7特征模型的系数值,将λ值放入predict()函数,如下所示:
lasso.coef 0.045, type = "coefficients")
lasso.coef
## 9 x 1 sparse Matrix of class "dgCMatrix"
## 1
## (Intercept) -0.1305900670
## lcavol 0.4479592050
## lweight 0.5910476764
## age -0.0073162861
## lbph 0.0974103575
## svi 0.4746790830
## lcp .
## gleason 0.2968768129
## pgg45 0.0009788059
LASSO算法在λ值为0.045时,将lcp的系数归零。下面是LASSO模型在测试集上的表现:
lasso.y "response", s = 0.045)
plot(lasso.y, test$lpsa, xlab = "Predicted", ylab = "Actual",
main = "LASSO")
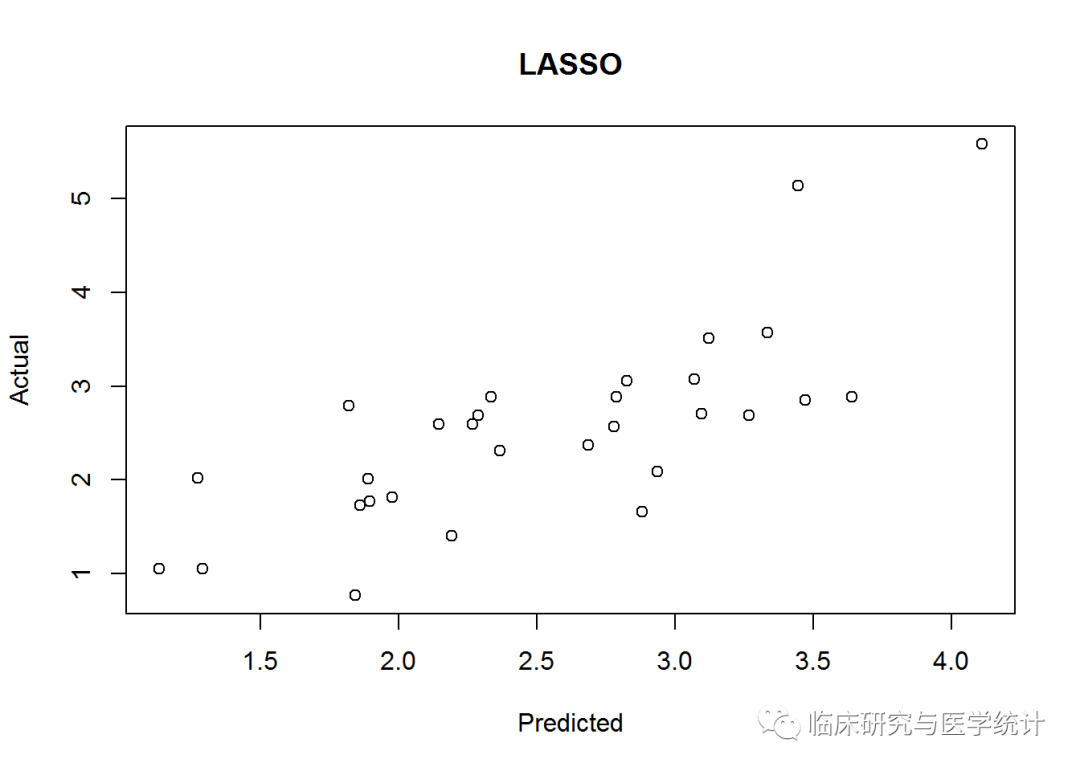
像以前一样,算出MSE的值:
lasso.resid 2)
## [1] 0.4437209
看起来我们的统计图和前面一样,只是MSE值有了一点点改进,重大改进的最后希望只能寄托在弹性网络上面了。要进行弹性网络建模,还可以继续使用glmnet包,要做的调整是不但要解出λ值,还要解出弹性网络参数α。回忆一下,α=0表示岭回归惩罚,α=1表示LASSO回归,弹性网络为0≤α≤1。同时解出两个不同的参数会非常麻烦,令人心生怯意,但是,我们可以求助于R中caret包。
10.弹性网络
caret包旨在解决分类问题和训练回归模型,它配有一个很牛的网站,帮助人们掌握其所有功能:http://topepo.github.io/caret/index.html。这个软件包有很多强大功能可以使用。现在我们的目的集中于找到λ和弹性网络混合参数α的最优组合。可以通过下面3个简单的步骤完成。
(1)使用R基础包中的expand.grid()函数,建立一个向量存储我们要研究的α和λ的所有组合。
(2)使用caret包中的trainControl()函数确定重取样方法,可使用LOOCV。
(3)在caret包的train()函数中使用glmnet()训练模型来选择α和λ。一旦选定参数,我们会像在岭回归和LASSO回归中做的那样,在测试数据上使用它们。
可以按照下面的规则试验这两个超参数。
(1)α从0到1,每次增加0.2;请记住,α被绑定在0和1之间。
(2)λ从0到0.20,每次增加0.02;0.2的λ值是岭回归λ值(λ = 0.1)和LASSOλ值(λ = 0.045)之间的一个中间值。
可以使用expand.grid()函数建立这个向量并生成一系列数值,caret包会自动使用这些数值。caret包通过下列代码生成α值和λ值:
grid 0,1, by=.2),
.lambda = seq(0.00, 0.2, by = 0.02))
使用table()函数,可以看到α和λ的全部66种组合:
table(grid)
## .lambda
## .alpha 0 0.02 0.04 0.06 0.08 0.1 0.12 0.14 0.16 0.18 0.2
## 0 1 1 1 1 1 1 1 1 1 1 1
## 0.2 1 1 1 1 1 1 1 1 1 1 1
## 0.4 1 1 1 1 1 1 1 1 1 1 1
## 0.6 1 1 1 1 1 1 1 1 1 1 1
## 0.8 1 1 1 1 1 1 1 1 1 1 1
## 1 1 1 1 1 1 1 1 1 1 1 1
可以确认这就是我们想要的结果——α值在0和1之间,λ值在0和0.2之间。对于重取样方法,我们要在代码中将method参数指定为LOOCV。还有其他的重取样方法可以选择,比如自助法和K折交叉验证法。trainControl()函数中有更多选择。在trainControl()函数中,你还可以通过selectionFunctioin()函数指定模型选择方法。对于定量型响应变量,使用算法的默认选择均方根误差即可完美实现:
control "LOOCV") #selectionFunction="best"
现在可以使用train()函数确定最优的弹性网络参数了。这个函数和lm()很相似,只需在函数语法中加上method=“glmnet”, trControl=control, tuneGrid=grid。将结果存储在一个名为enet.train的对象中:
set.seed(701) #our random seed
enet.train = train(lpsa ~ ., data = train,
method = "glmnet",
trControl = control,
tuneGrid = grid)
enet.train
## glmnet
##
## 67 samples
## 8 predictor
##
## No pre-processing
## Resampling: Leave-One-Out Cross-Validation
## Summary of sample sizes: 66, 66, 66, 66, 66, 66, ...
## Resampling results across tuning parameters:
##
## alpha lambda RMSE Rsquared MAE
## 0.0 0.00 0.7498311 0.6091434 0.5684548
## 0.0 0.02 0.7498311 0.6091434 0.5684548
## 0.0 0.04 0.7498311 0.6091434 0.5684548
## 0.0 0.06 0.7498311 0.6091434 0.5684548
## 0.0 0.08 0.7498311 0.6091434 0.5684548
## 0.0 0.10 0.7508038 0.6079576 0.5691343
## 0.0 0.12 0.7512303 0.6073484 0.5692774
## 0.0 0.14 0.7518536 0.6066518 0.5694451
## 0.0 0.16 0.7526409 0.6058786 0.5696155
## 0.0 0.18 0.7535502 0.6050559 0.5699154
## 0.0 0.20 0.7545583 0.6041945 0.5709377
## 0.2 0.00 0.7550170 0.6073913 0.5746434
## 0.2 0.02 0.7542648 0.6065770 0.5713633
## 0.2 0.04 0.7550427 0.6045111 0.5738111
## 0.2 0.06 0.7571893 0.6015388 0.5764117
## 0.2 0.08 0.7603394 0.5978544 0.5793641
## 0.2 0.10 0.7642219 0.5936131 0.5846567
## 0.2 0.12 0.7684937 0.5890900 0.5898445
## 0.2 0.14 0.7713716 0.5861699 0.5939008
## 0.2 0.16 0.7714681 0.5864527 0.5939535
## 0.2 0.18 0.7726979 0.5856865 0.5948039
## 0.2 0.20 0.7744041 0.5845281 0.5959671
## 0.4 0.00 0.7551309 0.6072931 0.5746628
## 0.4 0.02 0.7559055 0.6044693 0.5737468
## 0.4 0.04 0.7599608 0.5989266 0.5794120
## 0.4 0.06 0.7667450 0.5911506 0.5875213
## 0.4 0.08 0.7746900 0.5824231 0.5979116
## 0.4 0.10 0.7760605 0.5809784 0.6001763
## 0.4 0.12 0.7784160 0.5788024 0.6024102
## 0.4 0.14 0.7792216 0.5786250 0.6035698
## 0.4 0.16 0.7784433 0.5806388 0.6024696
## 0.4 0.18 0.7779134 0.5828322 0.6014095
## 0.4 0.20 0.7797721 0.5826306 0.6020934
## 0.6 0.00 0.7553016 0.6071317 0.5748038
## 0.6 0.02 0.7579330 0.6020021 0.5766881
## 0.6 0.04 0.7665234 0.5917067 0.5863966
## 0.6 0.06 0.7778600 0.5790948 0.6021629
## 0.6 0.08 0.7801170 0.5765439 0.6049216
## 0.6 0.10 0.7819242 0.5750003 0.6073379
## 0.6 0.12 0.7800315 0.5782365 0.6053700
## 0.6 0.14 0.7813077 0.5785548 0.6059119
## 0.6 0.16 0.7846753 0.5770307 0.6075899
## 0.6 0.18 0.7886388 0.5753101 0.6104210
## 0.6 0.20 0.7931549 0.5734018 0.6140249
## 0.8 0.00 0.7553734 0.6070686 0.5748255
## 0.8 0.02 0.7603679 0.5991567 0.5797001
## 0.8 0.04 0.7747753 0.5827275 0.5975303
## 0.8 0.06 0.7812784 0.5752601 0.6065340
## 0.8 0.08 0.7832103 0.5734172 0.6092662
## 0.8 0.10 0.7811248 0.5769787 0.6071341
## 0.8 0.12 0.7847355 0.5750115 0.6093756
## 0.8 0.14 0.7894184 0.5725728 0.6122536
## 0.8 0.16 0.7951091 0.5696205 0.6158407
## 0.8 0.18 0.8018475 0.5659672 0.6205741
## 0.8 0.20 0.8090352 0.5622252 0.6256726
## 1.0 0.00 0.7554439 0.6070354 0.5749409
## 1.0 0.02 0.7632577 0.5958706 0.5827830
## 1.0 0.04 0.7813519 0.5754986 0.6065914
## 1.0 0.06 0.7843882 0.5718847 0.6103528
## 1.0 0.08 0.7819175 0.5755415 0.6082954
## 1.0 0.10 0.7860004 0.5731009 0.6107923
## 1.0 0.12 0.7921572 0.5692525 0.6146159
## 1.0 0.14 0.7999326 0.5642789 0.6198758
## 1.0 0.16 0.8089248 0.5583637 0.6265797
## 1.0 0.18 0.8185327 0.5521348 0.6343174
## 1.0 0.20 0.8259445 0.5494268 0.6411104
##
## RMSE was used to select the optimal model using the smallest value.
## The final values used for the model were alpha = 0 and lambda = 0.08.
我们选择最优模型的原则是RMSE值最小,模型最后选定的最优参数组合是α=0,λ=0.08。实验设计得到的最优调优参数是α=0和λ=0.08,相当于glmnet中s=0.08的岭回归。R方为61%,真是乏善可陈。在测试集上验证模型的过程和前面一样:
enet "gaussian",
alpha = 0,
lambda = .08)
enet.coef .08, exact = TRUE)
enet.coef
## 9 x 1 sparse Matrix of class "dgCMatrix"
## 1
## (Intercept) 0.137811097
## lcavol 0.470960525
## lweight 0.652088157
## age -0.018257308
## lbph 0.123608113
## svi 0.648209192
## lcp -0.118214386
## gleason 0.345480799
## pgg45 0.004478267
enet.y "response", s= .08)
plot(enet.y, test$lpsa, xlab = "Predicted",
ylab = "Actual", main = "Elastic Net")
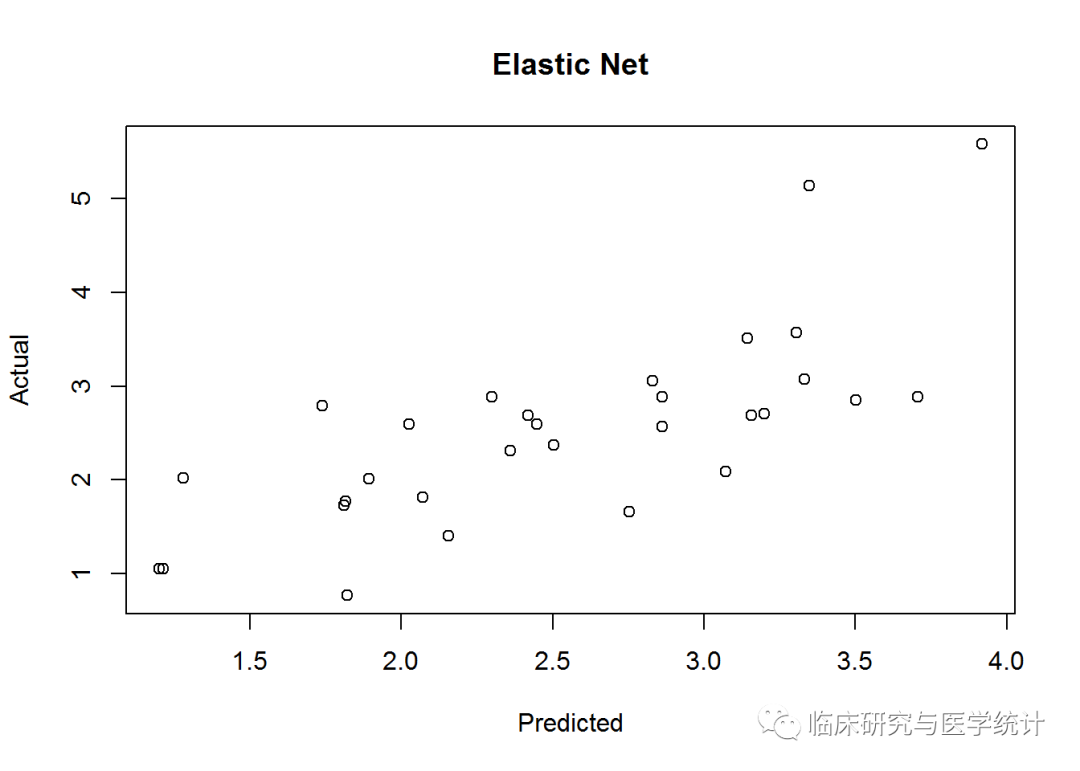
和以前一样,计算MSE:
enet.resid 2)
## [1] 0.4795019
这个模型的误差与岭回归很接近。在测试集上,LASSO模型在误差方面表现最好。模型可能过拟合了!我们的三特征最优子集模型是最容易解释的,但考虑误差的话,却更应该接收另外一种技术得出的模型。可以在glmnet包中使用10折交叉验证来确定哪个模型更好。
11.交叉验证
我们通过caret包使用过LOOCV,现在试试K折交叉验证。glmnet包在使用cv.glmnet()估计λ值时,默认使用10折交叉验证。在K折交叉验证中,数据被划分成k个相同的子集(折),每次使用k-1个子集拟合模型,然后使用剩下的那个子集做测试集,最后将k次拟合的结果综合起来(一般取平均数),确定最后的参数。在这个方法中,每个子集只有一次用作测试集。在glmnet包中使用K折交叉验证非常容易,结果包括每次拟合的λ值和响应的MSE。默认设置为α=1,所以如果你想试试岭回归或弹性网络,必须指定α值。因为我们想看看尽可能少的输入特征的情况,所以还是使用默认设置,但由于训练集中数据量的原因,只分3折:
set.seed(317)
lasso.cv = cv.glmnet(x, y, nfolds = 3)
plot(lasso.cv)
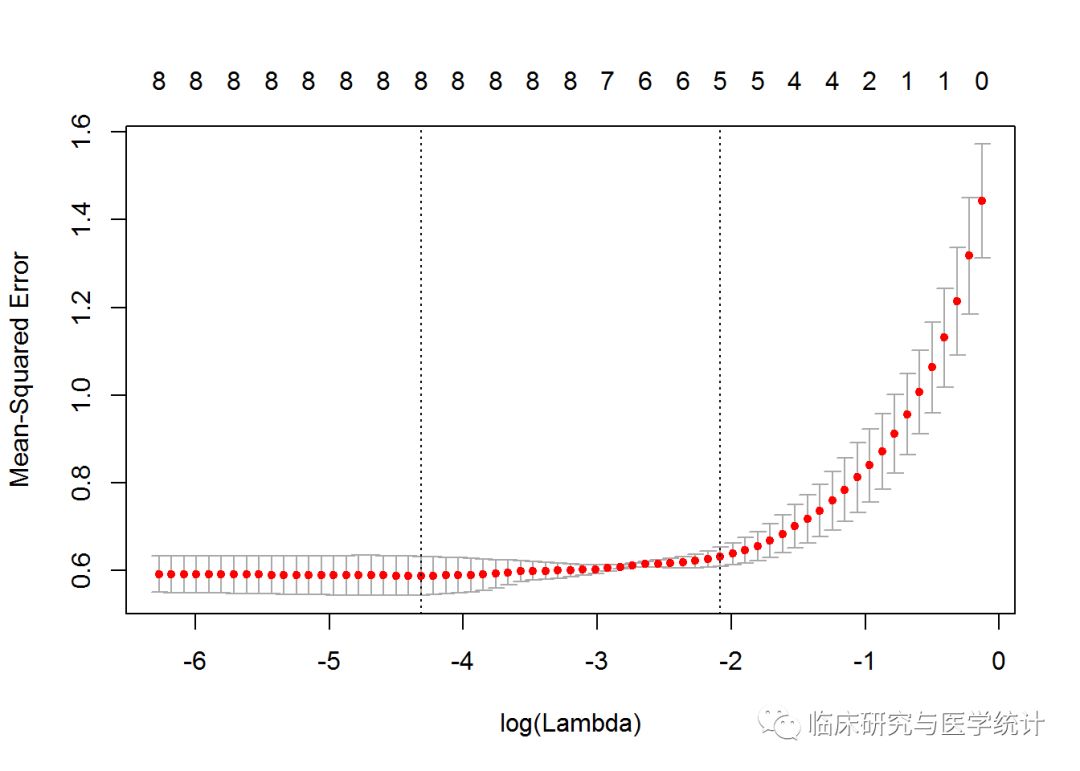
CV统计图和glmnet中其他统计图有很大区别,它表示λ的对数值和均方误差之间的关系,还带有模型中特征的数量。图中两条垂直的虚线表示取得MSE最小值的logλ(左侧虚线)和距离最小值一个标准误差的logλ。如果有过拟合问题,那么距离最小值一个标准误的位置是非常好的解决问题的起点。你还可以得到这两个λ的具体值,如下所示: