bioconda是conda中专门用来管理生物信息相关软件的频道。
绝大部分的用户都是利用bioconda安装软件,绝大部分的教程也都是教大家如何去使用bioconda安装软件。但是bioconda的便利性并不是从天而降的,而是无数的软件开发者持续的不断的贡献和更新一些软件的recipe (菜谱) 的结果。这篇教程就是教大家如何在一个软件没有bioconda的安装方式时,自己动手写一个菜谱。
准备工作
首先,我们在https://github.com/bioconda/bioconda-recipes 上fork该项目到自己的GitHub项目下,然后将其clone到本地
git clone https://github.com/xuzhougeng/bioconda-recipes.git
cd bioconda-recipes
如果一不小心克隆使用了官方的项目或者我的项目,也可以通过如下命令进行修改
git remote rm origin
git remote add origin 你的github地址
如果你之前已经在GitHub上fork了官方的repo,那么你还需要进行同步
git remote add upstream https://github.com/bioconda/bioconda-recipes
git fetch upstream
git merge upstream/master
由于bioconda是建立在conda基础上的频道,因此我们还需要预先安装好 conda,关于conda 的安装、配置和使用可以看我上传到哔哩哔哩的视频。之后安装 conda-build, 用于创建conda软件包的框架。
# 安装conda-build, 后续用于创建模板
conda install conda-build
实际流程
向bioconda贡献一个软件的菜谱大致分为6步:
-
-
-
-
-
-
第一步: 创建分支
我们需要先切换到主分支(master), 并保证你的主分支处于最新状态
git checkout master
git pull upstream master
git push origin master
接下来,我们创建新的分支,用于处理当前的菜谱,避免影响主分支
git checkout -b update_My_recipes
我建议这里的
update_My_recipes
修改成具体的软件名,这样更有标识性。
第二步: 编辑recipe
我们使用
conda skeleton
创建一个软件包框架, 这里pypi指的是软件托管在pypi上,wgdi则是软件名。
# bioconda-recipes目录下
cd recipes
conda skeleton pypi wgdi
因为wgdi存在一些依赖环境,如PAML, MAFFT, MUSCLE, PAL2NAL, 所以我们需要修改
wgdi/meta.yaml
文件,在其中加入这些软件。
run:
- biopython
- matplotlib
- numpy
- pandas >=1.1.0
- python
- scipy
- paml
- mafft
- muscle
- pal2nal - iqtree
❝
如果是直接在原有的meta.yaml进行编辑,那么需要注意修改shasum检验值,conda会计算下载文件的检验值,跟meta.yaml里的校验值进行比较,确保conda下载的文件是完整且可靠。
❞
事实上,conda skeleton 只是做了一小部分事情,我目前的meta.yaml 还不完整,直到后续的递交中修改了数十次才知道出问题的地方以及如何修改。因此建议后还需要查阅 https://docs.conda.io/projects/conda-build/en/latest/resources/define-metadata.html 了解meta.yaml的更多知识。
第三步: 向GitHub上推送更新
这里就是用git命令将我们的修改推送到GitHub上
# bioconda-recipes目录下
git add recipes/wgdi
git commit -m "add wgdi recipes"
git push origin update_My_recipes
第四步: 创建pull 请求
此时,让我们回到GitHub 中我们fork的bioconda-recipes页面。我们可以看到该修改已经被推送到GitHub上。
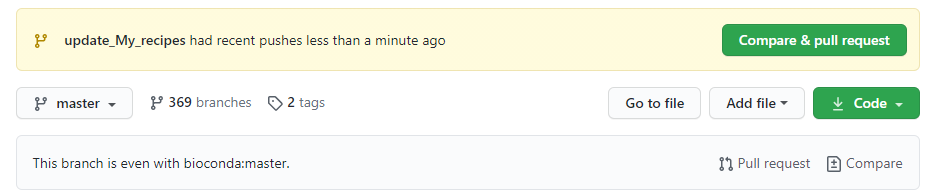
我们点击 Compare & pull request, 接着就会弹出一个页面,介绍如何书写提交申请。
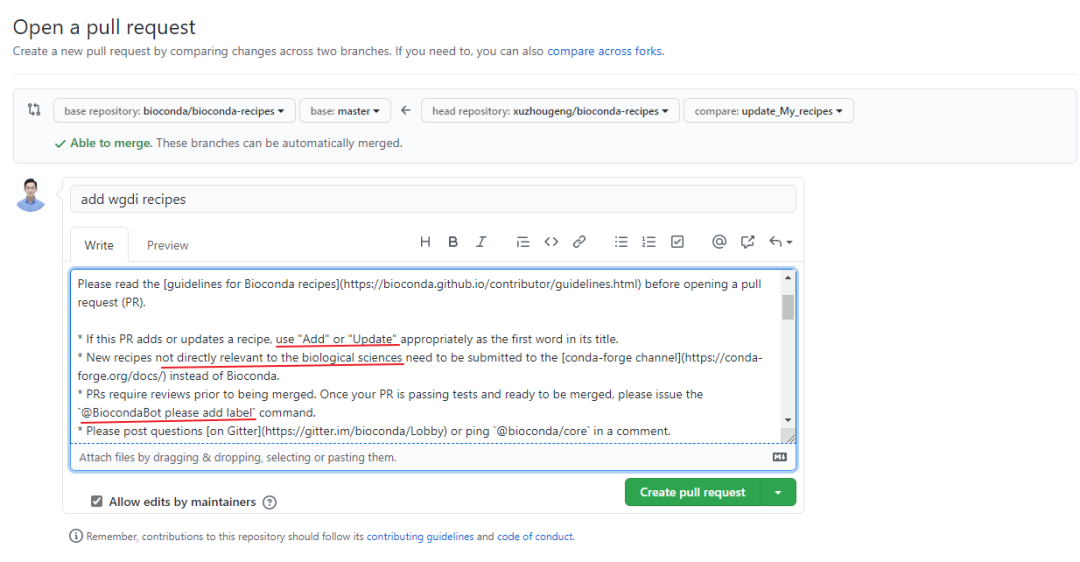
这里写了4点要求,分别为
-
在PR中加入Add或者update 作为用于说明目的,必须是第一个词
-
-
一旦通过了审核,需要在后续的issue中加入
@BiocondaBot please add label
, 或者
@Biocondabot please merge
, 具体区别在后面的命令说明中有提到。我看了已关闭的PR中,通常都是merge。
-
如果有问题,你可以到Gitter或者在评论中用
@bioconda/core
提出自己的问题。
当你提出PR之后,bioconda 的构建系统就会测试我们提交的菜谱。如果通过了测试,我们就可以跳转到下一步,否则需要重复第二~四步,直到通过为止。




