STRING: 一个预测蛋白质间功能相关性的数据库
研究一个基因及其编码的蛋白质,一方面要了解它们的功能,另一方面需研究与此蛋白质相互作用的其他蛋白质的信息。
搜寻已知蛋白质之间和预测蛋白质之间相互作用的生物数据库和网络资源有很多,如:BIND、DIP、IntAct、InterDom、MINT、STRING、HPRD、HPID、MPPI等。其中,STRING数据库是目前应用最广泛的研究蛋白相互作用的数据库。
STRING(检索交互基因/蛋白质的搜索工具)数据库是由CPR,EMBL,KU,SIB,TUD和UZH等学术机构联合开发的,
旨在提供蛋白与蛋白相互作用的关键评估和整合,这种相互作用既包括蛋白质之间直接的物理作用,也包括蛋白质之间间接的功能相关性
。
STRING数据库信息来源广泛,包括实验数据,文章中挖掘的结果,综合其他数据库信息和生物信息学预测的结果。
STRING数据库是免费的,而且定期更新。最新版本涵盖了203
1种生物体的约960万种蛋白质的138亿个相互作用信息。
打开网址
https://string-db.org/
,首先映入眼帘的是这个界面:
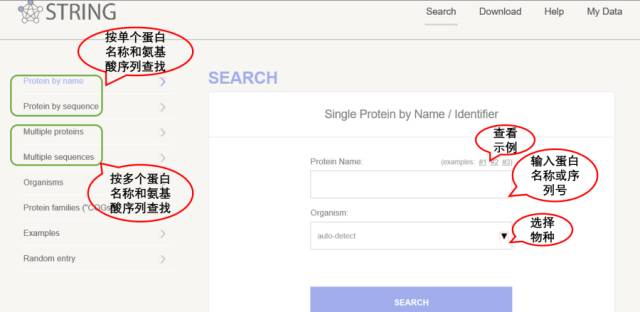
可以根据不同分析起点选择合适的搜索方式。我们可以通过单个蛋白名称、多个蛋白名称和氨基酸序列进行搜索,也可以查看示例和随机搜索。随机搜索是数据库随机选择一个蛋白,该蛋白至少与4个中等信度或者更好的预测蛋白相互作用。Organisms可以搜索你感兴趣的物种在STRING数据库中是否存在。搜索COG(直系同源基团)可以通过蛋白质家族而不是单个生物体中的蛋白质进行搜索。
通常情况下,我们会输入蛋白的名称或者登陆号,然后选择相应的物种。如物种选择auto-detect,则在查询的过程中,如果相关的蛋白质名称出现在几个不同的物种中,则数据库系统会将这些物种全部显示出来,我们可以选择感兴趣的进行下一步的查询。
网络图清晰的展示了预测的与我们感兴趣的蛋白有相互作用的一组蛋白。网络的节点表示蛋白质,点击可查看该蛋白质相关信息
。大的节点表示该蛋白的3D结构已知或能够预测,小的节点表示该蛋白的3D结构未知。节点之间的直线表示蛋白质之间的相互作用关系,点击可查看两蛋白质互作信息。节点之间的直线有3种模
式,evidence模式,confidence模式,action模式。
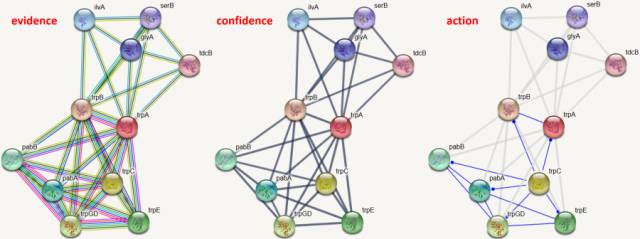
Evidence模式:不同颜色的线代表预测蛋白关联的不同的证据,可多达8种证据。

Confidence模式:线的粗细表示蛋白相互作用的置信度。

action模式:不同的颜色和形状的线表示不同的作用模式。



导航按钮可以帮助我们了解数据的信息,更改参数,并查看支持预测关联的不同类型的证据。

Viewers介绍了STRING数据库信息来源,包括实验数据,文章中挖掘的结果,综合其他数据库信息和生物信息学预测的结果等。

Legend部分显示我们检索的蛋白,与检索蛋白相互作用的蛋白的信息,并按得分进行排序。如果我们输入的基因是两个功能的融合,则两者都会显示。

Settings部分我们根据需要可以进行一些参数设置,请注意,只有您按”update”按钮,设置才会更改成功。

置信度得分中设置最小需要的交互作用得分为阈值,高于阈值的置信度得分才会出现在网络图中,低于这个分数会有更多的假阳性。置信度分数是KEGG数据库中相同代谢图中两种酶之间预测链接存在的近似概率。置信度范围如下:
低置信度-0.15
中等置信度-0.4
高置信度-0.7
最高置信度-0.9
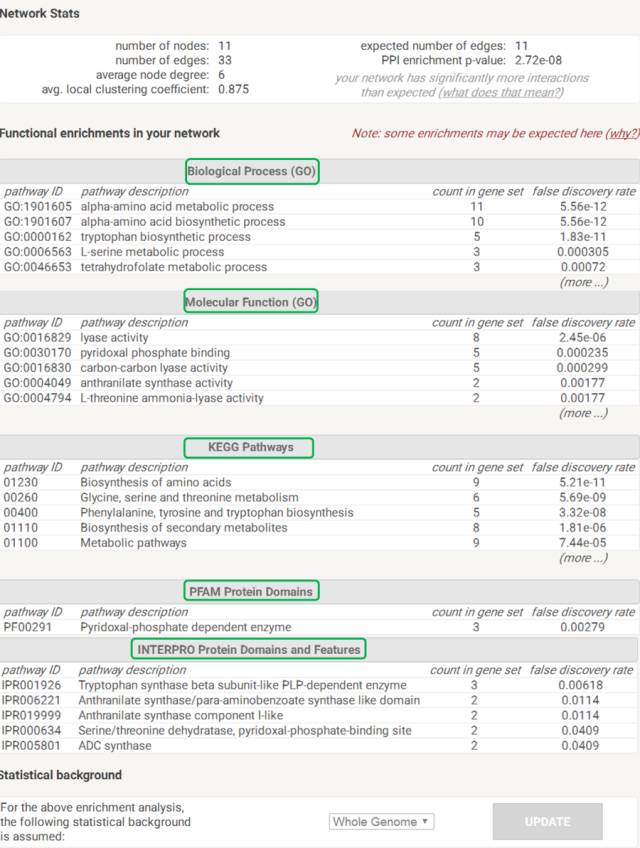
Analysis部分是对网络图的一些简单描述,像网路图中的节点数,互作关系数等。这部分内容里还包括GO富集、KEGG富集和蛋白结构域。总之我们可以从这部分内容中获得网络图中蛋白更多的信息。
Export部分可以选择不同的格式输出结果。







