责编 | 奕梵
N
6
-甲基腺嘌呤
(N
6
-methyladenosine, m
6
A)
是一种广泛存在的RNA修饰,在调控拼接和翻译等方面具有重要的作用
(Wang
et al.
, 2015, Xiao
et al.
, 2016)
。目前基于抗体检测的方法
(m
6
A-seq)
得到广泛使用
(Luo
et al.
, 2014)
,但该方法无法精确到单碱基的水平。m
6
A-CLIP
(Ke
et al.
, 2015)
和miCLIP
(Linder
et al.
, 2015)
可以从单碱基精度检测m
6
A位点,然而由于文库构建流程繁琐,导致应用受限。不依赖于抗体的方法m
6
A-REF-seq
(Zhang
et al.
, 2019)
和MAZTER-seq
(Garcia-Campos
et al.
, 2019)
能从单碱基精度识别和定量ACA基序上的RNA修饰,极大的促进了RNA修饰的研究。但如果想检测ACA基序之外的其它已知的DRACH
(D=G/A/U,R=G/A,H=A/U/C)
修饰还需要开发更为全面和便捷的单碱基水平定量方法。目前牛津纳米孔技术
(ONT)
的直接RNA测序
(DRS)
技术具有检测RNA中碱基修饰信号的潜力,
但目前还缺乏对应的基于DRS直接在单碱基和单转录本水平定量检测m
6
A修饰的方法。
2021年
1月7日,福建农林大学海峡联合研究院林学中心
顾连峰
教授课题组在
Genome Biology
期刊在线发表了题为
Quantitative profiling of N6-methyladenosine at single-base resolution in stem-differentiating xylem of Populus trichocarpa using Nanopore direct RNA sequencing
的研究论文。
该研究提供了一种可在单转录本单碱基水平的分辨率定量m
6
A修饰的方法,为在动植物中的m
6
A修饰研究提供了一种极为有效的检测手段。
利用该方法首次在杨树次生木质部中定量描述选择性多聚腺苷酸化和m
6
A修饰的关联。该研究首先检测人工合成的有m
6
A修饰和无m
6
A修饰位点周围的Nanopore直接RNA测序输出的原始电流信号,提取其信号均值、中值、标准差及宽度等特征构建XGBoost模型,测试数据集的结果表明其m
6
A修饰预测精度达97%。
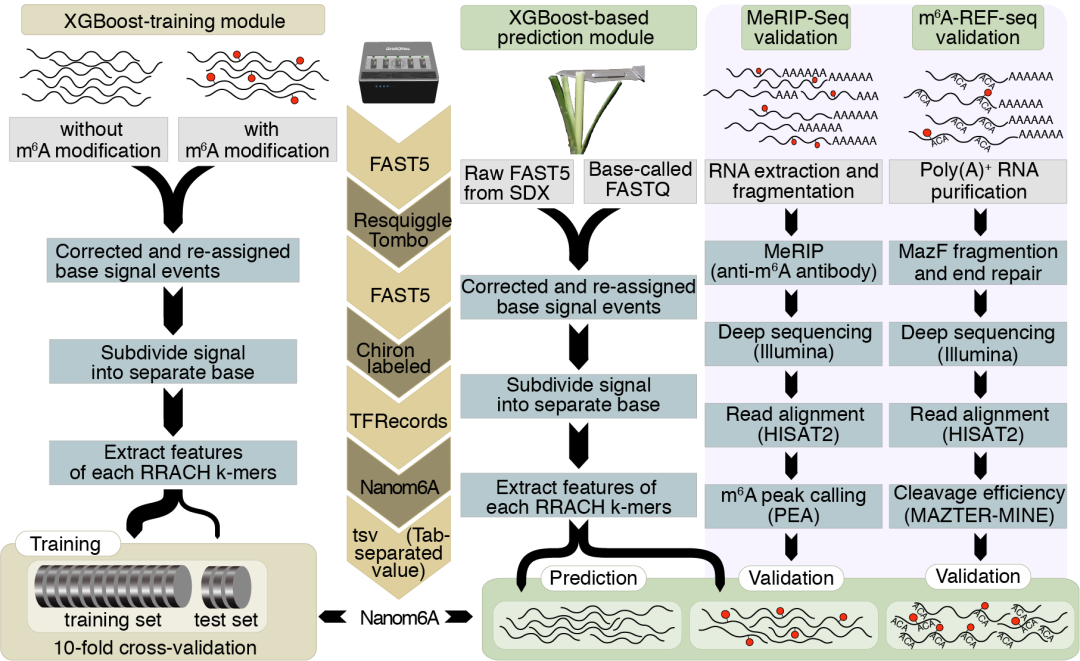
为了验证这种方法的准确性,首先使用该方法对人类HEK293T细胞系和shMETTL3的DRS数据中进行m
6
A位点识别,并和已发表的基于ligation-assisted extraction and thin-layer chromatography
(SCARLET)
(Liu
et al.
, 2013)
技术获得marker基因进行比较。发现可以准确检测到TUG1、TPT1、ACTB及BSG这些RNA上的已知m
6
A修饰位点,这些位点大多位于DRS读段的3’末端区域。基于DRS获得的野生型和METTL3突变体上的m
6
A修饰率,与MeRIP-Seq的结果完全吻合,并且突变体的修饰率整体上均呈现降低趋势,说明该m
6
A定量识别方法可以应用在动物的DRS数据上。
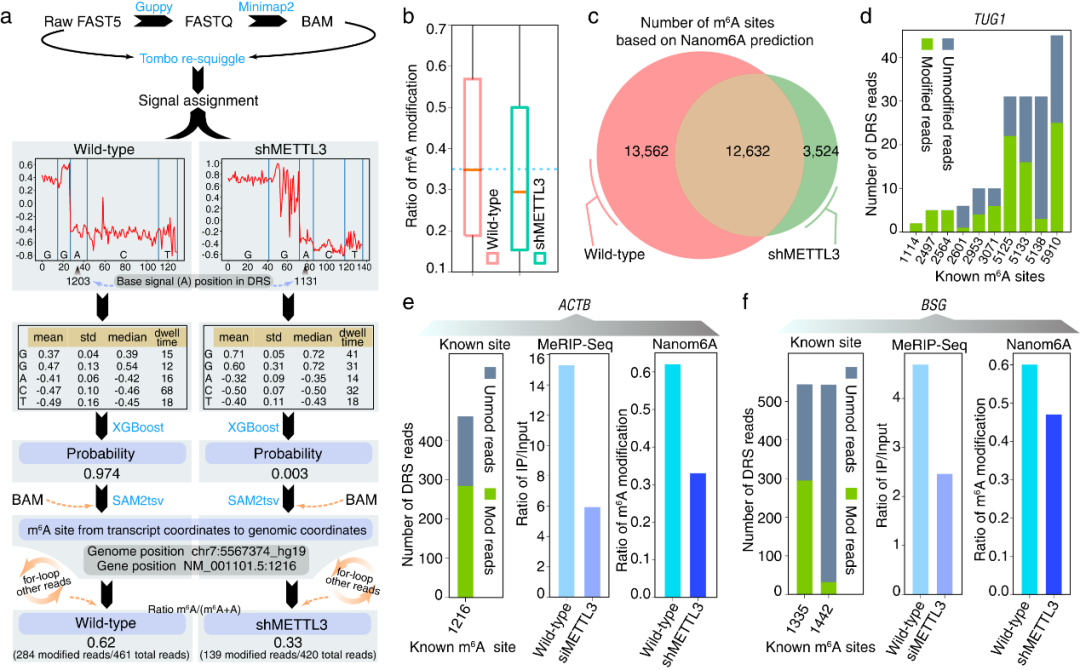
为了进一步验证该方法的通用性,对Parker等报道的拟南芥野生型、
VIRILIZER
(
vir-1
)突变体和VIRc回补株系的DRS数据(Parker
et al.
, 2020)采用本文方法进行m
6
A修饰识别,显示在
vir-1
突变体中m
6
A位点明显减少,从整体分布上看在
vir-1
突变体中m
6
A位点在终止密码子和3’UTR区域明显减少。因此通过植物DRS数据获得修饰信息和遗传学证据一致,说明该m
6
A定量识别方法也可以应用在植物的DRS数据上。
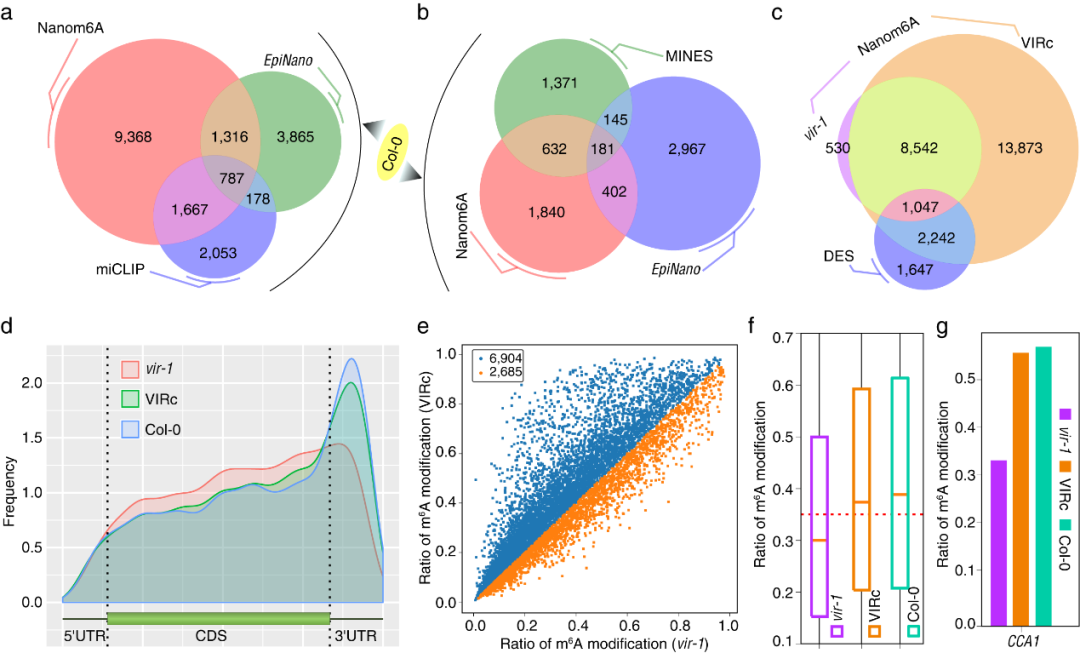
论证了本文方法在动物植物研究中的适用性之后,以毛果杨次生木质部为材料,抽提poly(A)
+
RNA进行Nanopore直接RNA测序。检测到在毛果杨次生木质部中m
6
A位点也富集在转录本的终止密码子或3’UTR上。基因的表达水平和m
6
A修饰水平呈负相关。与木材合成相关的基因修饰率低,可能与维持这些基因在次生木质部的高表达相关。应用毛果杨次生木质部DRS数据,和另外两种只能定性分析m
6
A的软件
(
EpiNano
及MINES)
进行m
6
A位点识别比较,发现本文的方法能预测到另外两种算法无法检测到的低修饰率的m
6
A位点,这些位点大多富集于终止密码子和3’UTR区域。更为重要的是本文的方法除了可以定性识别,还可以进行m
6
A修饰定量分析,这是
EpiNano
及MINES所没有的功能。
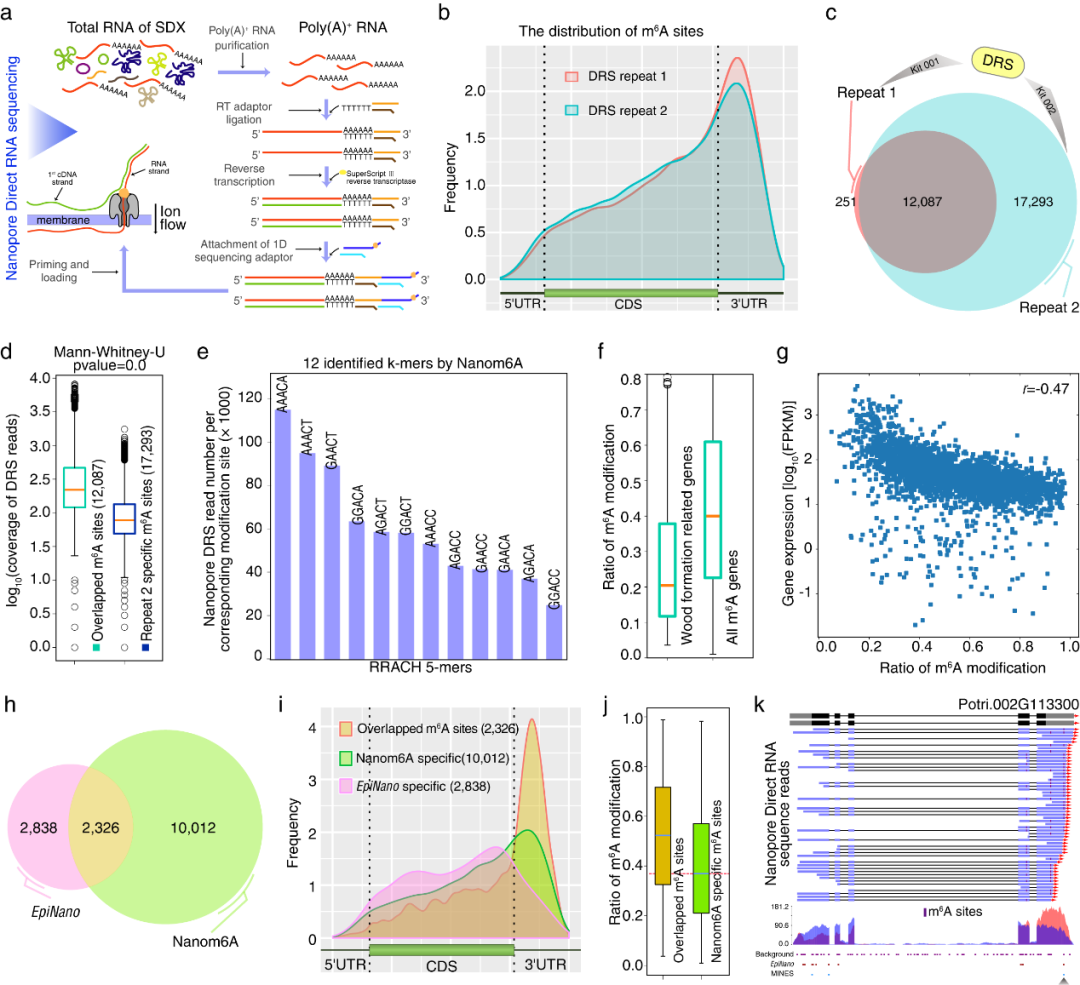
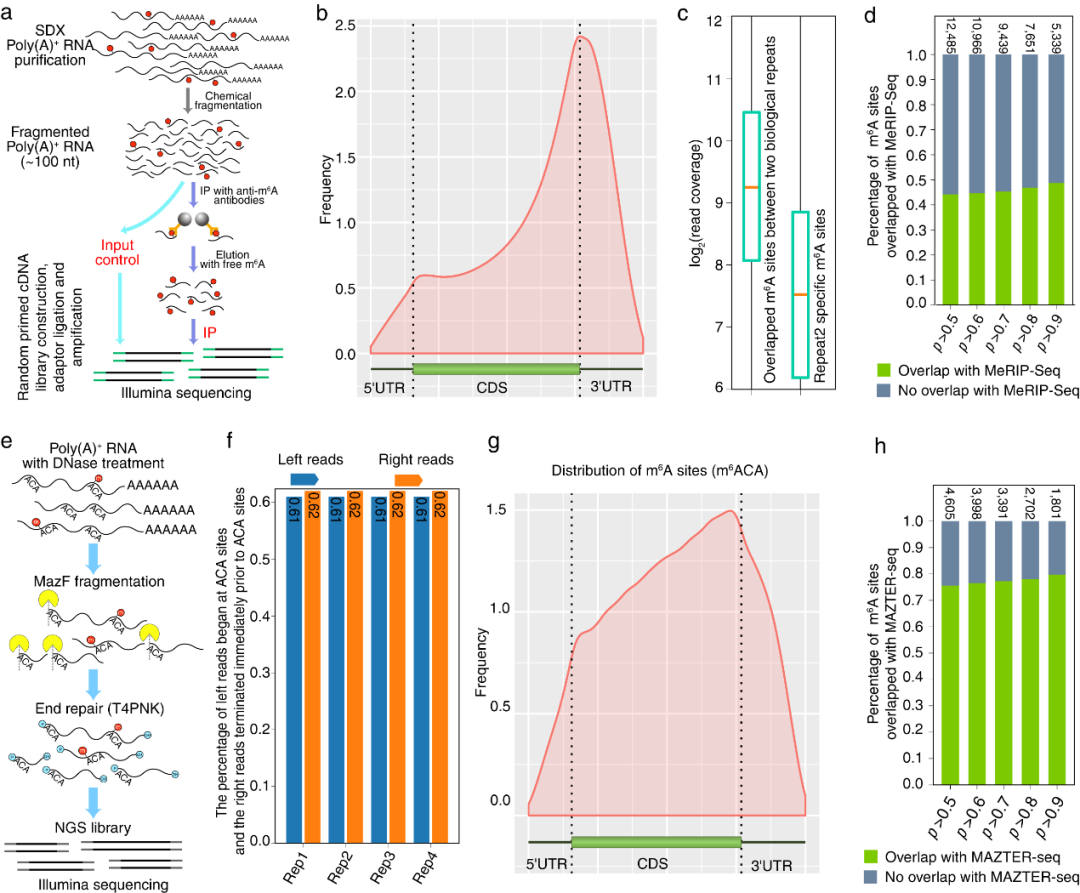
为了验证毛果杨次生木质部中基于DRS数据获得修饰的准确性,进一步运用MeRIP-Seq和m
6
A-REF-seq两种完全不同的技术,在MeRIP-Seq数据中,片段富集于终止密码子和3’UTR区域。DRS中81%的m
6
A修饰基因在MeRIP-Seq中被检测到,49%的基于DRS的m
6
A位点与MeRIP-Seq峰值重叠。在m
6
A-REF-seq结果中,m6ACA基序同样富集于终止密码子区域,更高精度的m
6
A-REF-seq有80%的m
6
A位点与基于DRS的m
6
A位点重叠。这与m
6
A-REF-seq
(Zhang
et al.
, 2019)
能做到单碱基水平相一致。
最后,运用Nanom6A对毛果杨次生木质部具有选择性多聚腺苷酸化位点的远端和近端poly(A)转录本进行m
6
A定量分析,发现远端poly(A)和近端poly(A)的m
6
A修饰比率不同。例如,基因Potri.019G083300,远端poly(A)转录本的m
6
A修饰比率远少于近端poly(A)转录本。该研究为m
6
A修饰与选择性多聚腺苷化之间的调控关系提供了重要的线索。
福建农林大学海峡联合研究院林学中心博士生
高宇帮
和
刘旭庆
为论文共同第一作者,海峡联合研究院
顾连峰
教授为通讯作者。林学中心工程师
吴碧致
、博士生
王慧慧
、
席飞虎
、
Markus V. Kohnen
副教授以及科罗拉多州立大学
Anireddy S. N. Reddy
教授也参与了本项目。东北林业大学
李伟
教授课题组在实验材料上提供大量的帮助,中山大学
骆观正
教授课题组在m
6
A-REF-seq文库构建上给予了详细的技术指导。该研究得到了国家重点研发基金(2016YFD0600106)的支持。
Garcia-Campos, M.A., Edelheit, S., Toth, U., Safra, M., Shachar, R., Viukov, S., Winkler, R., Nir, R., Lasman, L., Brandis, A., Hanna, J.H., Rossmanith, W. and Schwartz, S. (2019) Deciphering the “m6A Code” via Antibody-Independent Quantitative Profiling.
Cell
, 178, 731-747.e716.



















