
童鞋,有没有做过基因敲除实验呢?Crispr/Cas9接触过吗?为了发好文章是不是也开始跃跃欲试了呢?
Crispr/Cas9基因编辑技术带给科研世界的不仅仅是一种高效快速精准的技术,更是一场革命。技术的更新换代太快了,商业化的运作使得技术发展如虎添翼,20世纪需要10~20年才能被有效地利用起来,现在只需要1~2年就可以无孔不入了,在上海,光能搜索得到的Crispr/Cas9编辑公司就不下40家,更何况是哪些看不见的呢,既然林子大了什么鸟都有,那么遇到烂公司就不算是小概率事件了。
那既然技术这么成熟,咱们该自己做呢?还是交给外面的公司做呢?很多有科研情怀、本着技多不压身理念的童鞋会选择自己亲自操刀,尝试Crispr/Cas9基因编辑技术,那开始的时候,咱先从Crispr/Cas9的原理讲起吧。
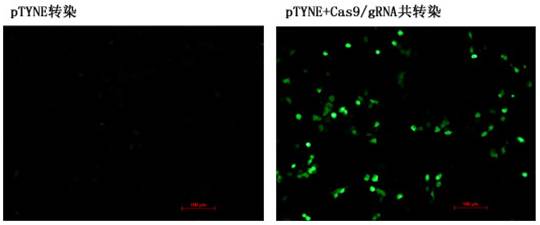
(一) Crispr/Cas9起源于细菌对抗病毒的斗争
免疫系统(Immune system)是个非常强大的防御保护模式,它是抵抗外来侵害的主要手段。人有免疫系统,当结核杆菌侵染到人体血液时,免疫系统就开始工作了,婴儿时期已经被卡介苗免疫过,机体中针对结核杆菌的淋巴B细胞会在一系列的结核杆菌的刺激下,大量增殖,并大量地产生中和抗体,使得结核杆菌被巨噬细胞识别、吞噬,从集体中清理出来。
这种防御模式被科学家应用起来,大家也都非常熟悉的技术,例如大家平常使用的抗体,ELISA试剂盒,免疫组化IHC,ChIP等免疫技术,都是基于这套机体的免疫系统原理:抗体识别抗原。
机体对待入侵者的过程是识别、产生记忆、再次入侵、消除。Crispr/Cas9在微生物中起到的作用就是识别入侵、将病毒的DNA录入自己的基因组,当病毒再次入侵时候,切割病毒DNA、RNA进而消除病毒的感染。
CRISPR(Clustered regularly interspaced short palindromic repeats)是生命进化历史上,细菌和病毒进行斗争产生的免疫武器,简单说就是病毒能把自己的基因整合到细菌,利用细菌的细胞工具为自己的基因复制服务,细菌为了将病毒的外来入侵基因清除,进化出CRISPR系统,利用这个细菌免疫系统,细菌地把病毒基因从自己的染色体上切除,这是细菌特有的免疫系统。
CRISPR/Cas9 系统介导的基因组编辑是由sgRNA引导的,对靶序列的识别是 RNA 与 DNA 的碱基配对过程,然后Cas9核酸酶将DNA给咔嚓一刀切开了,相比蛋白质对 DNA 序列的识别要精确更多,降低了脱靶切割的几率,减低了细胞毒性。CRISPR/Cas9 的构建仅仅需要设计与靶序列互补的 RNA 即可,普通的实验室也可以自行完成构建,这大大提高了基因操作的效率及简便性。
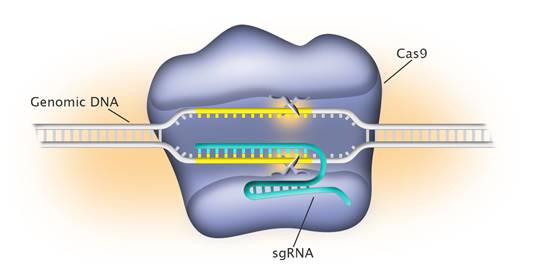
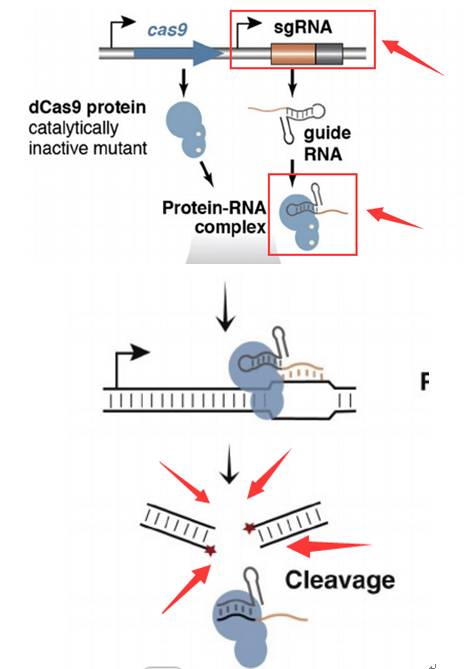
(二)Crispr/Cas9分子机制原理
我们从上面的讲述中知道,Crispr技术有两个分子在参与:Cas9核酸酶+sgRNA(自行设计)。
其中Cas9核酸酶的基因已经被整合到商业化载体中,只要自行设计sgRNA,然后构建Cas9/sgRNA表达载体,转染细胞,就可以行使基因组的定点基因编辑任务。
sgRNA(small-guided RNA)是CRISPR基因敲除敲入系统中重要的组成部分,由两部分组成—tracRNA和crRNA,两部分融合表达后,即sgRNA,与cas9蛋白结合,引导cas9酶靶向基因组DNA进行剪切。
(三)SgRNA的序列特征和设计
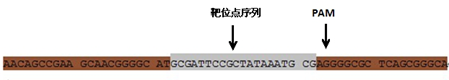
SgRNA 设计靶序列选择:在NCBI找到该基因CDS区,分析相应的基因组结构,明确CDS的外显子部分。
按照基因本身的性质,选择候选的待敲除位点,确定待敲除位点。对于蛋白编码基因,如果该蛋白具重要结构功能域,可考虑将基因敲除位点设计在编码该结构域的外显子。可选择将待敲除位点放在起始密码子ATG后的外显子上。如果被敲除的靶标基因是microRNA或者lncRNA,可以将待敲除位点设计在编码成熟microRNA的外显子或在编码成熟microRNA的外显子的5’和3’侧翼序列。
SgRNA的序列特征
运行中的sgRNA序列设计工具
确定待敲除位点后,选择23-至250bp的外显子序列输入到在线免费设计sgRNA 的软件中(例如http://crispr.mit.edu/ ),然后进行设计运算,软件会自动输出sgRNA序列。根据设计的 sgRNA 靶点序列,NNNNNNNNNNNNNNNNNNNNNGG合成一对序列互补的DNA Oligos。
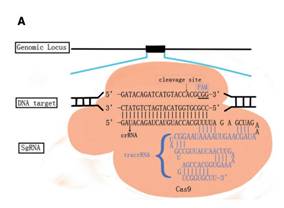
SgRNA脱靶问题一直备受关注,如何设计特异性好的靶点成为大家关注的焦点,大体原则如下:
(1)对于sgRNA的长度,一般应为20 nt左右;
(2)对于sgRNA序列的碱基组成,可选3'末端含GG的sgRNA,同时sgRNA种子序列尽量避免以4个以上的T结尾,GC%含量最佳为40%~60%;
(3)sgRNA在设计网站上的评分越高,越容易脱靶;
(4)如需造成基因knockout,需尽量选择包含有ATG的外显子或靠近ATG的外显子;
(5)检查sgRNA靶向结合位点基因组序列是否存在SNPs;
(6)如采用Cas9单切口酶,设计paired-gRNA需考虑sgRNA之间的间距;
(7)全基因脱靶效应分析,需考虑脱靶位点最大允许几个错配碱基数,建议最少5个碱基。重点考察种子序列和非种子序列碱基错配数。
--请持续关注Crispr专题后续序列
“高分文章和基金是如何用CRISPR/Cas9的?”
“Crispr/Cas9高通量筛选的应用”
“Crispr/Cas9疾病治疗的应用”
长按二维码识别关注“小张聊科研”

关注后获取《科研修炼手册》1.0、2.0、3.0、4.0、基金篇精华合集















