
时间来到2020年,科研热点层出不穷,但是我们发现,ceRNA依然是大家申请课题和发文章的热点方向,这与ceRNA的整体思路比较清晰,后期验证也相对容易实现有很大的关系。
审时度势,与时俱进,本次培训将带你了解ceRNA是何物,各种RNA的相互作用网络如何构建,这些非编码RNA如何产生,如何行使生物学功能,如何相互作用构成生物网络。
同行是如何研究这些热点的,哪些方法可以为我所用。我们将和你分享一些成功案例,梳理研(fa)究(wen)思(tao)路(lu)。
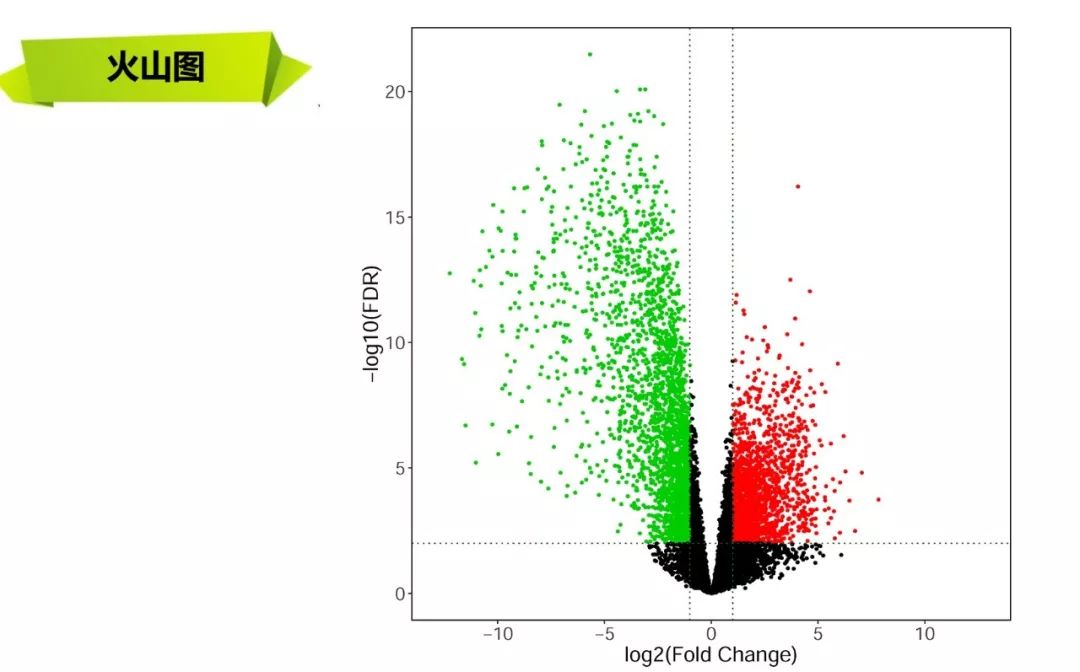
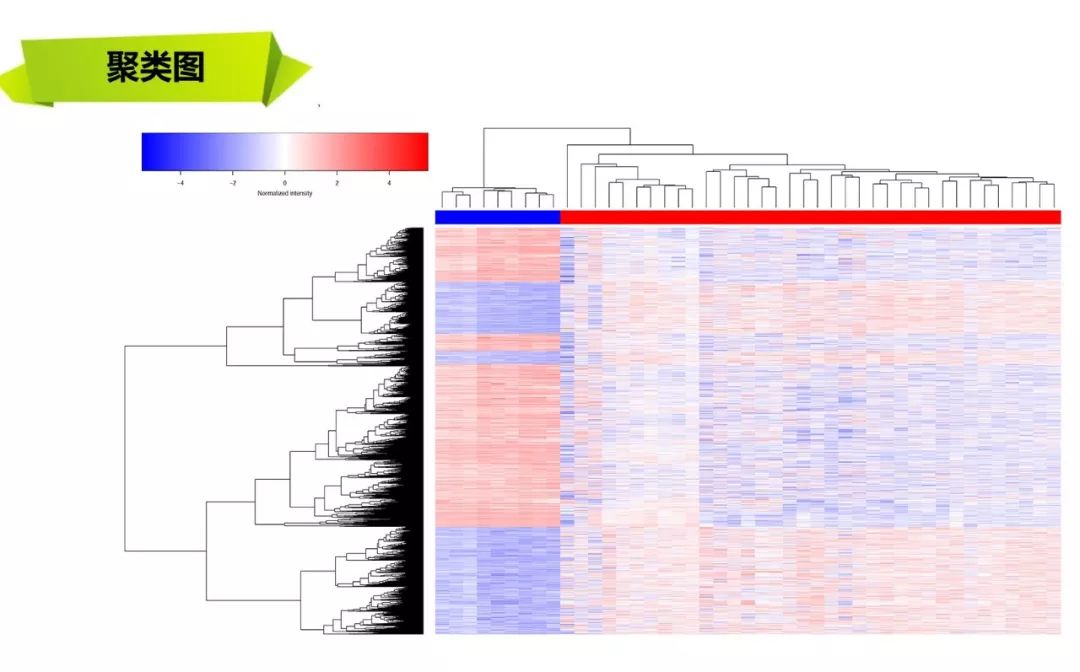
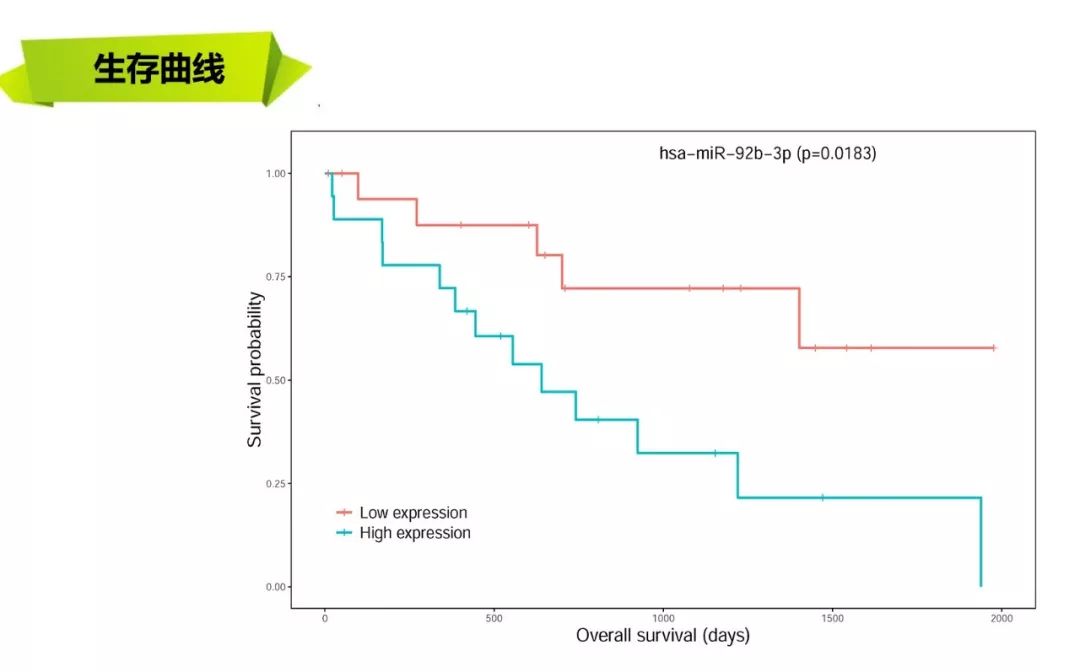
看着人家发文章似乎很容易,自己做起来却无从下手。本次培训将手把手教你如何从RNAseq数据(mRNA, lncRNA, miRNA)得到差异表达基因,基因功能富集,生存分析,ceRNA网络构建。教你画出火山图,柱状图,聚类图,热图,气泡图,KM生存曲线,ceRNA网络图。
学员评价
课程亮点
1、展示
不做实验纯生信挖掘
的paper,阐述非编码RNA发文套路;
2、讲解从mRNA, lncRNA, miRNA数据筛选差异表达基因的方法,
直通关键分子
;
3、传授非编码RNA靶点的预测方法,
构建网络机制
;
4、介绍miRNA,circRNA,lncRNA各种数据库、实用工具及其使用方法,
充实科研武器库
;
5、
TCGA数据库提供
创新性
附
加
代码
,可适用于各种表型分组比较(如肿瘤分期、性别);
6、
对代码进行精心改造,可以用于
综合性数据库
GEO
;
6、
高效速成
,
代码
“傻瓜式”自动化运行
,课后自己修改少量参数便可直接上手,比如这个:
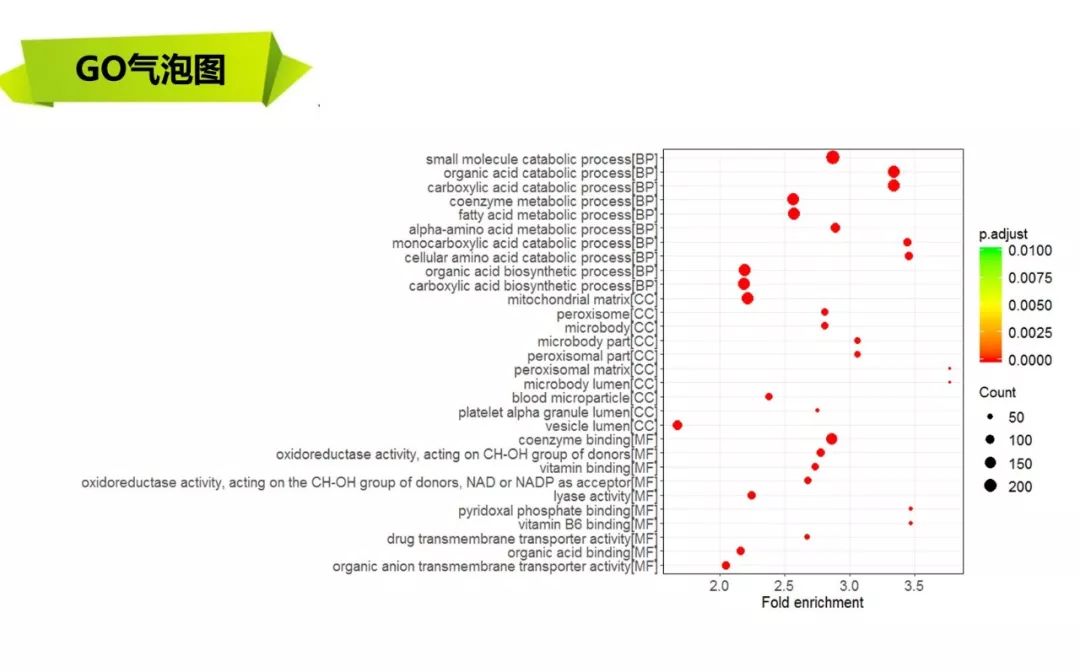
关于GO富集,我们还会提供环状图
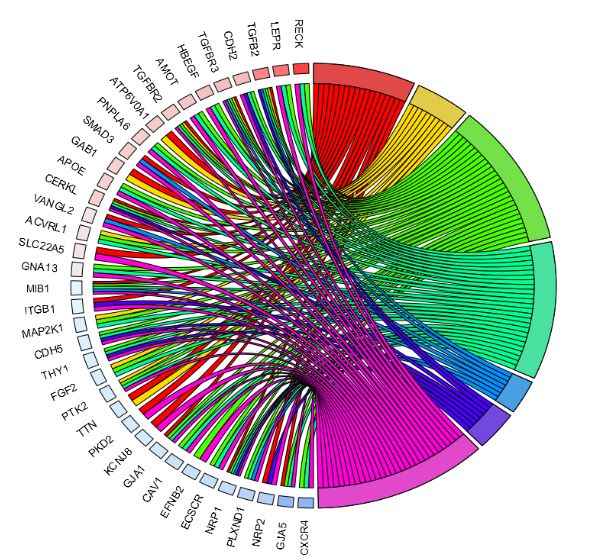
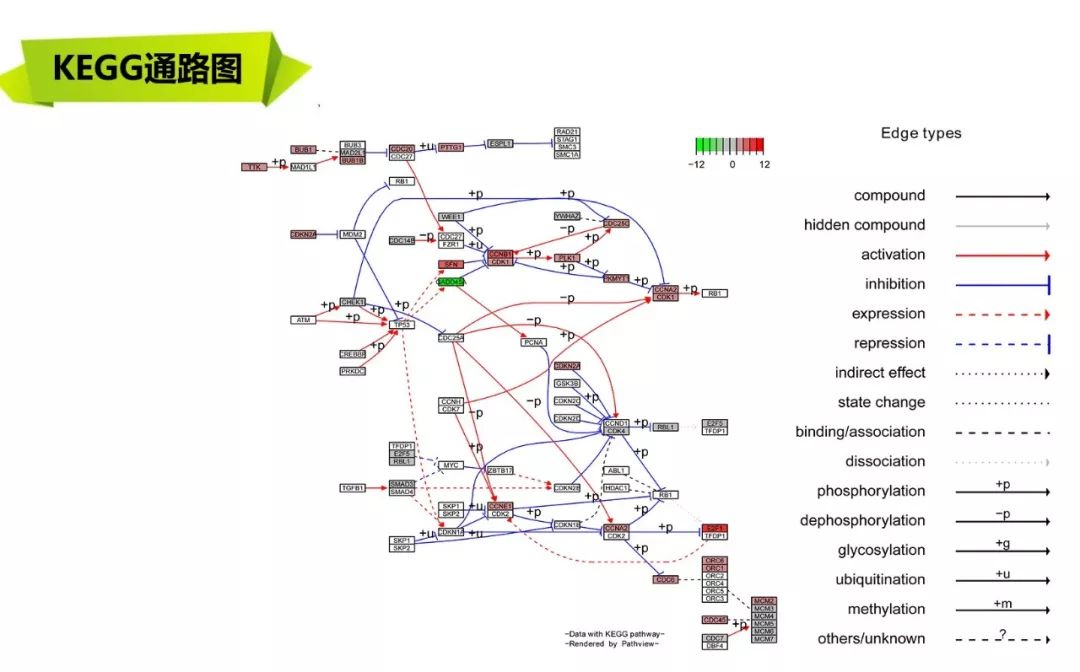
还有这个,
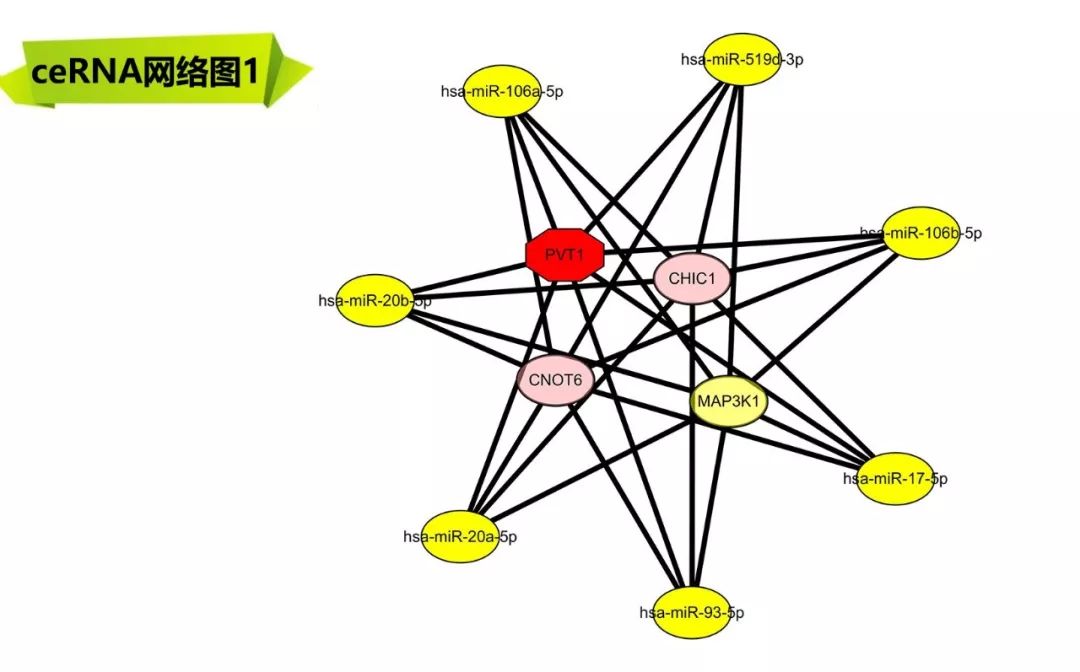
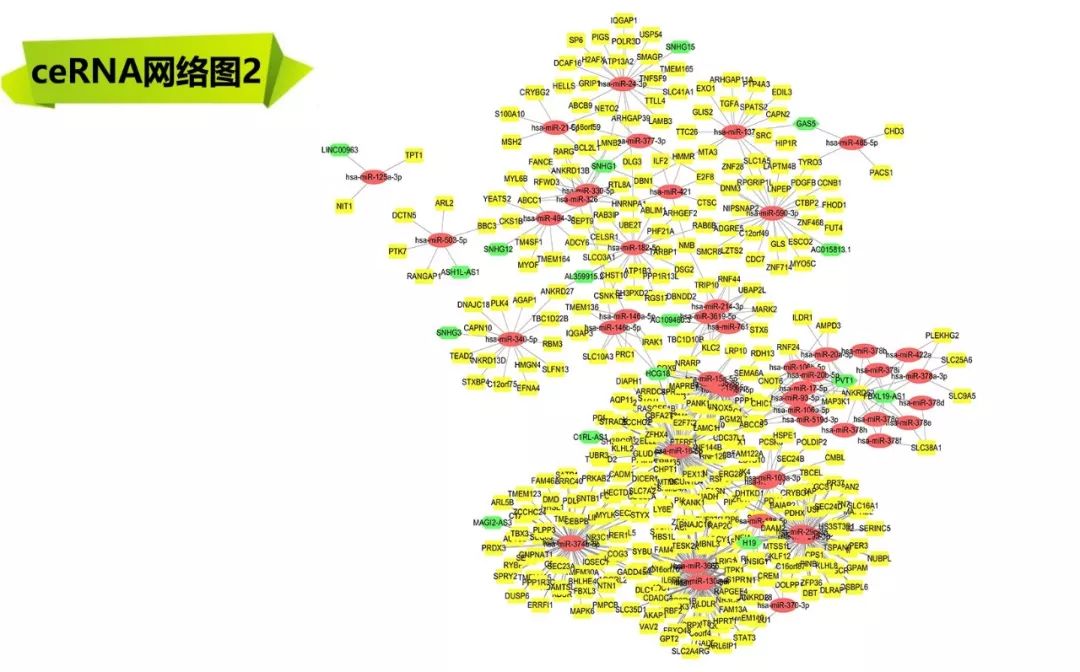
另外,ceRNA还有高大上的桑基图可以表示:
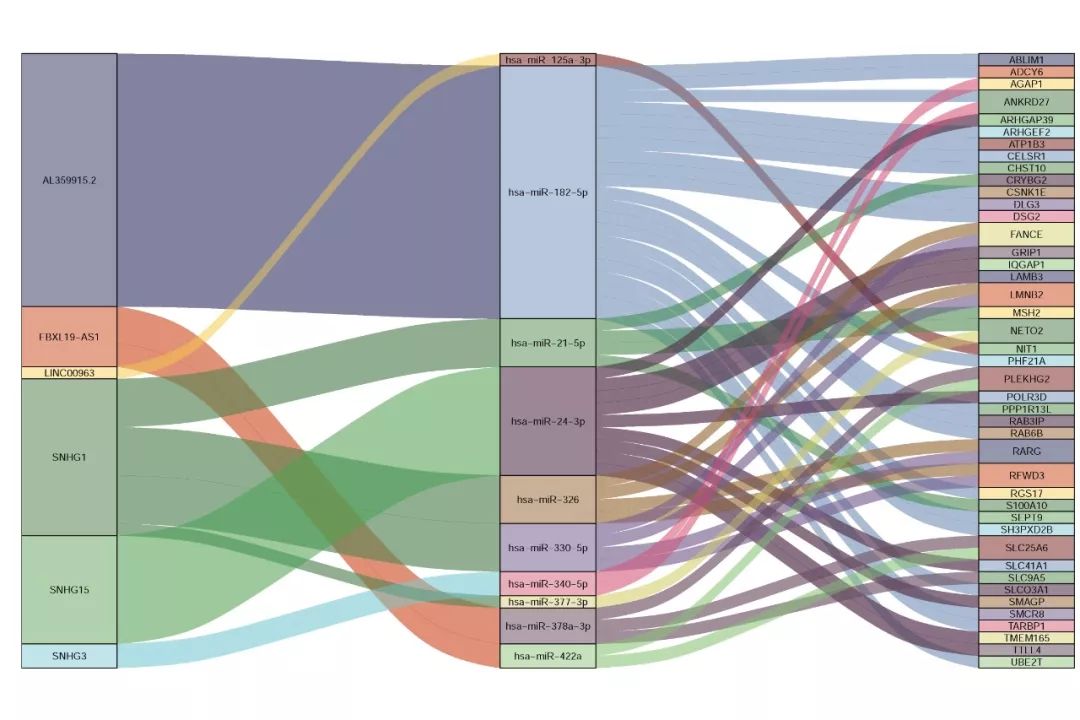
不仅如此,
课后,我们还会给大家设置一个配套的线上课程,作为线下课程的补充和拓展。线上课程详细介绍了多种非编码RNA数据库的使用以及如何利用这些数据库构建ceRNA网络,每一节都是
干货满满
。

李老师
,生物信息学博士,有八年的测序数据分析经验。博士期间参与多个课题项目,涉及机器学习,芯片数据分析,核酸及蛋白序列分析,全基因组甲基化测序数据分析,全转录组测序数据分析,miRNA及靶基因分析,癌症相关基因预测及预后分析等,发表SCI论文32篇,其中一作及并列一作15篇。
工作期间积累了丰富的DNA,甲基化,RNA捕获测序,Amplicon捕获测序实验流程和数据分析经验。具有ctDNA数据分析经验,对变异数据进行注释, 指导临床用药。自行搭建Linux集群,开发测序数据全自动分析平台。
适合人群
广大临床/科研工作者,研究生,
有无生信基础均可参加,
有R语言基础者更佳。
另外,本课程如果想上课更轻松,
建议课前先补一下R语言编程基础,
上课会更轻松,强烈推荐李老师的“
R语言基础与绘图
”,课程链接如下:
https://ke.qq.com/course/1184328?tuin=831272e

课程表
|
Day1
|
|
8:00-9:00
|
报道,领取资料
|
|
9:00-12:00
|
背景知识介绍
|
|
miRNA,circRNA,lncRNA的产生,作用机制,功能
|
|
miRNA,circRNA,lncRNA相关数据库及工具介绍,使用及数据下载
|
|
ceRNA案例分享 (包括mRNA-miRNA-lncRNA网络和mRNA-miRNA-circRNA网络)
|
|
12:00-13:00
|
午休
|
|
13:00-17:00
|
R语言简介
|
|
R语言概述
|
|
R软件及R包安装
|
|
R语言语法及数据类型
|
|
条件语句
|
|
循环
|
|
函数
|
|
17:00-17:30
|
学员提问及讨论
|
|
Day 2
|
|
9:00-12:00
|
利用TCGA数据构建ceRNA网络(实操,基于R)
|
|
TCGA简介及数据下载
|
|
寻找差异表达基因
|
|
火山图,热图,聚类图,柱状图
|
|
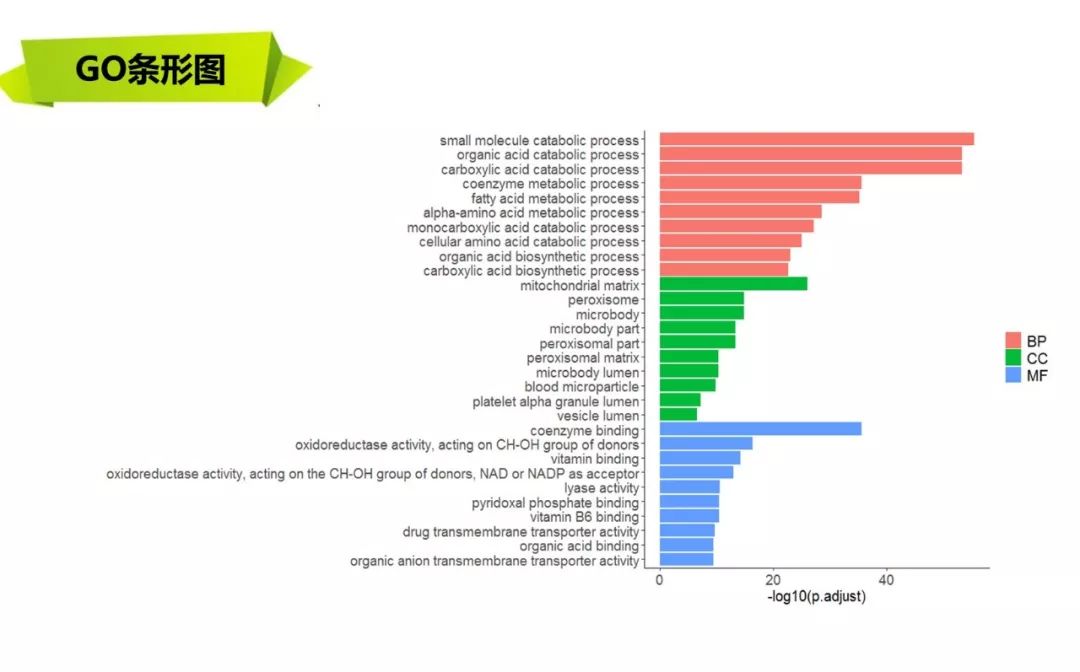
差异表达基因GO,KEGG富集分析,气泡图,柱状图,KEGG通路图展示,GO富集环状图
|
|
mRNA,lncRNA表达相关性分析,点状图
|
|
mRNA, lncRNA, miRNA网络构建,cytoscape展示,hub基因筛选,ceRNA网络桑基图
|
|
12:00-13:00
|
午休
|
|
13:00-17:00
|
利用GEO数据构建ceRNA网络(实操,基于R)
|
|
GEO简介及数据下载
|
|
寻找差异表达基因
|
|
火山图,热图,聚类图,柱状图
|
|
差异表达基因GO,KEGG富集分析,气泡图,柱状图,KEGG通路图展示,
GO富集环状图
|
|
mRNA,lncRNA表达相关性分析,点状图
|
|
mRNA, lncRNA, miRNA网络构建,cytoscape展示,hub基因筛选,
ceRNA网络桑基图
|














