SNP引物和探针的原理在于探针退火温度:
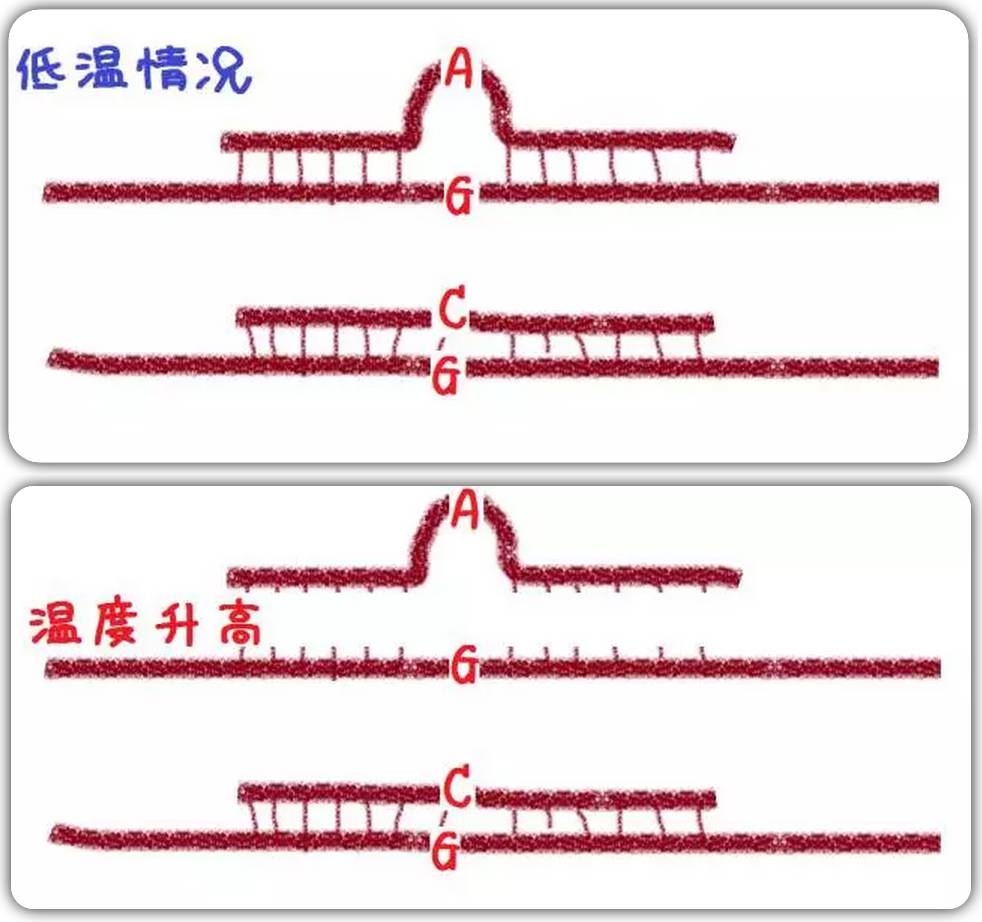
SNP的引物探针还是比较讲究的,首先探针的Tm值要比引物高5℃左右,保证引物延伸的时候探针正在模板上卧轨,等待被水解。
SNP检测尽量用MGB探针,由于MGB这种淬灭集团会结合到DNA的小沟,提高Tm值,也就是说普通探针要二三十个碱基才能达到的Tm值,MGB探针十多个就可以达到了。

比如MGB探针如果15个碱基就能达到对应的Tm值,普通探针需要25个,那探针中单个碱基的变化,在MGB探针中就是1/15的温度差,而普通探针则是1/25。MGB探针对于SNP检测的敏感度就更高了……
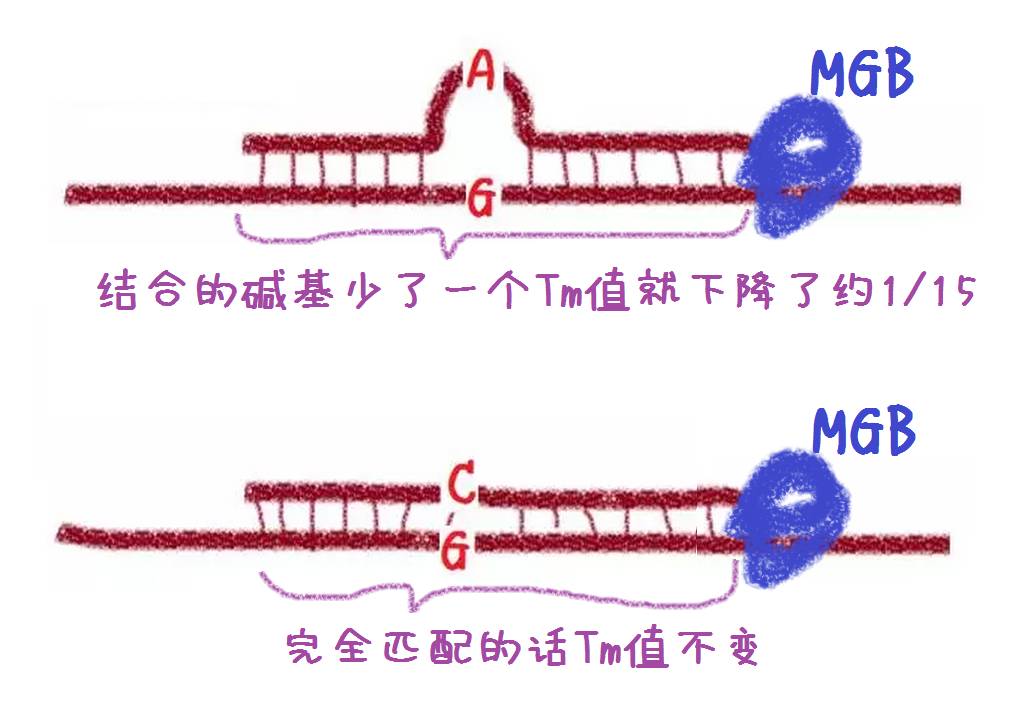
举个栗子,探针设计时候温度都是65℃,探针C能完全匹配,那么65℃的时候,探针还有一半结合在模板上。而探针A不能完全匹配到模板,Tm值就变成了65℃-1/15×65℃=60.67℃,也就是说当65℃延伸的时候,A探针就已经不能结合到模板上了,就是这样来分型的。
设计的话,SNP位点需要尽量设计在探针的中间位置,尽量不要靠近3’末端。那怎么设计这样探针呢?
有个简单的网站,直接输入SNP位点的序列,当然SNP位点需要用其他字母来替代,记得么?R=A/G,Y=C/T,M=A/C,K=G/T,S=C/G,W= A/T,H= A/C/T,B= C/G/T,V=A/C/G, D=A/G/T,N=A/C/G/T。
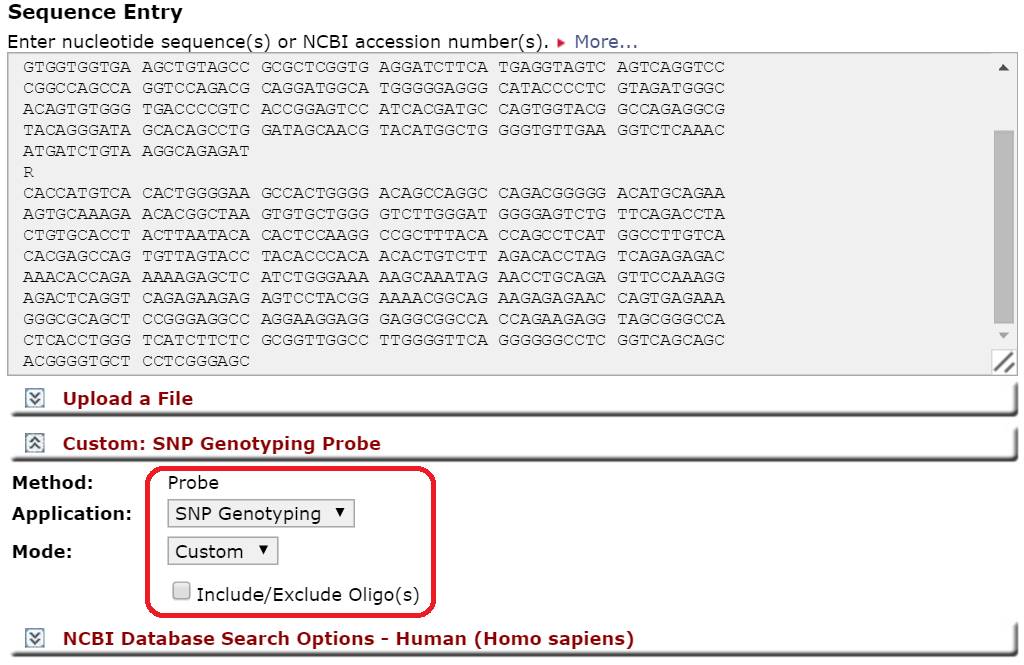
选择一下qPCR仪:
当然会自动识别楚SNP位点:

选一下荧光,由于MGB是ABI的专利,所以这里用的是一个叫BHQ plus的淬灭集团,其实效果基本一样。
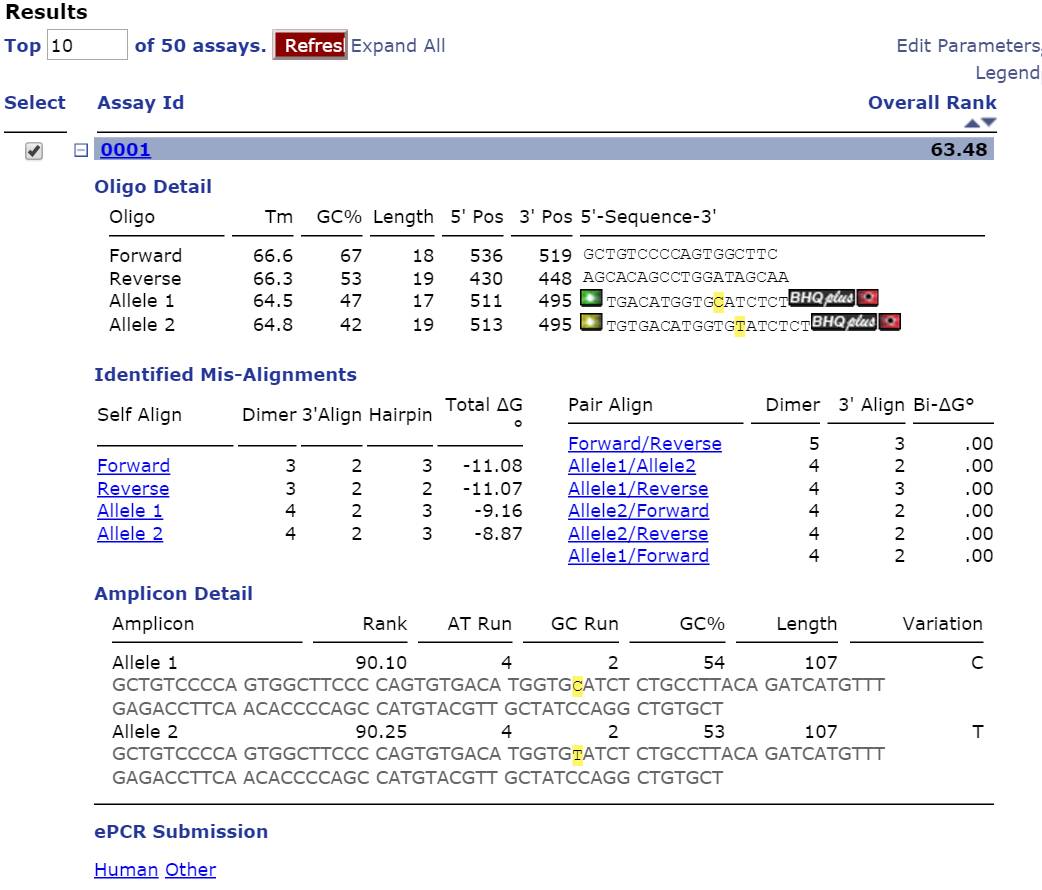
最后,就获得探针和引物的组合了。
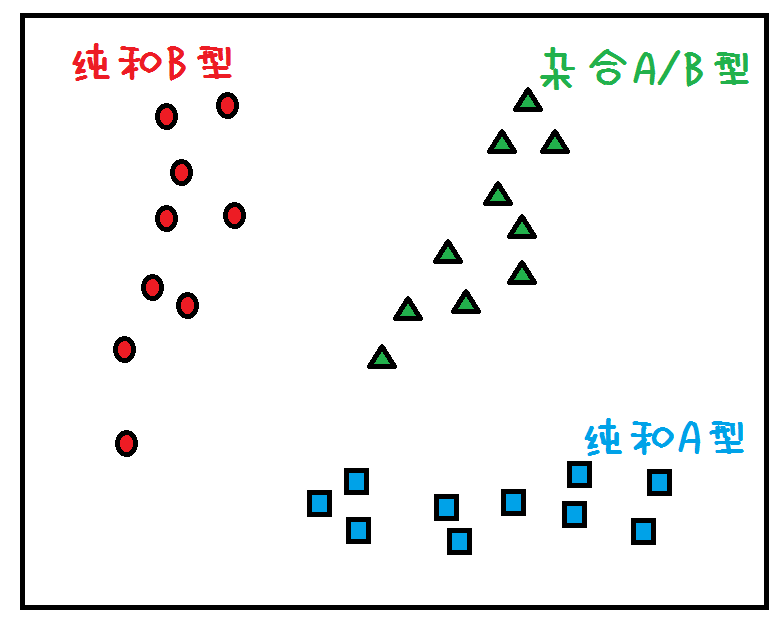
到时候合成的时候,把BHQ plus改成MGB去合成就行了。虽然有专利限制,但其实很多公司都是可以合成MGB的。到时候结果就会是这样的:
李莫愁博士:
用Taqman检测SNP的话,其实就是相对每个位点测一次序便宜些。缺点就是一次只能测一个位点,当然这些方法主要都是检验科在使用啦,真有钱的大金主,不会在意测多少序列的应该……好了,今天就先策到这里吧……要设计探针的这个网址,就回复“
BA
”就行了,记得别在评论区回复哈(后果自负)!
我的论文重复率这么高,到底要如何整?
PCR有问题,其实还是吃了文化的亏……
SNP位点是啥?为啥总是C变T,或者A变G?
如何暗中观察竞争对手的科研动态?
引物设计,那些你大概不知道的秘密
我的论文重复率这么高,到底要如何整?
一篇文献要怎么读?
会做实验就会做饭?你逗我?!






