当我们在分析时需要用
SQL数据库
时,尤其是需要
快速大量的操纵数据
的时候怎么办,很明显直接学习和使用SQL不是特别的方便。
ORM
就是来解决这个问题的,将复杂的SQL语言转化为开发者用的顺手的语种.
关于ORM,摘抄自维基百科
对象关系映射(英语:Object Relational Mapping,简称ORM,或O/RM,或O/R mapping),是一种程序设计技术,用于实现面向对象编程语言里不同类型系统的数据之间的转换。从效果上说,它其实是创建了一个可在>编程语言里使用的“虚拟对象数据库”。如今已有很多免费和收费的ORM产品,而有些程序员更倾向于创建自己的ORM工具。
面向对象是从软件工程基本原则(如耦合、聚合、封装)的基础上发展起来的,而关系数据库则是从数学理论发展而来的,两套理论存在显著的区别。为了解决这个不匹配的现象,对象关系映射技术应运而生。
简单的说:ORM相当于中继数据。具体到产品上,例如下边的ADO.NET Entity Framework。DLINQ中实体类的属性[Table]就算是一种中继数据。
对象关系映射成功运用在不同的面向对象持久层产品中,如:Torque,OJB,Hibernate,TopLink,Castor JDO,TJDO,Active Record,NHibernate,ADO.NET Entity Framework 等。
以Python的Peewee为例,这个ORM模块将每个SQL中的表都转化为一个类,对类进行操作,对于Python写手来说,一点也不复杂了吧。
Peewee轻量级ORM:
支持 MySQL、PostgreSQL、Sqlite、BerkeleyDB等常见数据库,相信能够满足大多数需求了.官方文档十分详尽,虽然都是英文的,这个模块有太多可以挖掘的东西,目前发现基本涵盖了所有需要的数据库的功能
官方文档:

安装模块
-
最基本的安装方法,不推荐,Linux还好,依赖问题好解决。Windows的话,八成会要求安装一个什么C++的解释器,总之,我没折腾懂怎么解决
pip install peewee
-
从源码装 github(https://github.com/coleifer/peewee),我觉着吧,应该也会有依赖问题难以解决
git clone https://github.com/coleifer/peewee.git
cd peewee
python setup.py install
-
官方没有提到,但是我最推荐,最方便的安装方法,anaconda,没听说过的请移步
conda install -c conda-forge peewee
-
官方文档中还有其他的安装方法,比如cpython等,由于不常用,就不说了
基础知识
peewee将数据库的不同部分映射为了类(官方文档称之为Model)的不同成分

peewee将数据库中对应的各种类别,转化为了模块中的各种Filed,比如
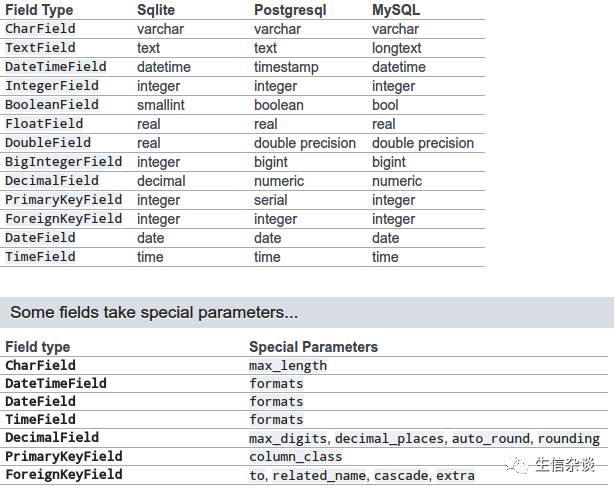
还有一个需要提一下的是ForeignKeyFiled, 方便将两个table union到一起的
from peewee import Model, CharField, ForeignKeyField
class Category(Model):
name = CharField()
parent = ForeignKeyField('self',
related_name='children',
null=True)
每个Filed都有一些参数可以设置:
更多
关于Sqlite多说几句(其他数据库可不可以这样用,不清楚,没试过)
一下是关于commit的问题,默认Sqlite是自动commit的,问题就是巨慢,因此连接数据库时可以指定autocommit为False,但是就需要用一下三种方式手动进行commit,后边还有一个atomic()功能与transaction()一致
db = SqliteDatabase('my_app.db', autocommit=False)
db.begin()
User.create(username='charlie')
db.commit()
with db.transaction():
User.create(username='huey')
@db.transaction()
def create_user(username):
User.create(username=username)
言归正传,开始举例
基本使用方法
我使用Sqlite数据库来做展示,Sqlite可以直接导入导出CSV文件,官方也给出了解决方法 【详情】
然而,我还是决定自己处理,毕竟有很多数据需要修饰什么的
第一次接触这个模块是从《Python高效开发实战Django Tornado Flask Twisted》(刘长龙著),对它的第一印象就是貌似很简单(?),因此,就直接沿着这个模块用下来了。
以Human protein atlas下载到的细胞系表达量数据为例,简单说明一些用法
下载到一个CSV文件,总大小51.3M
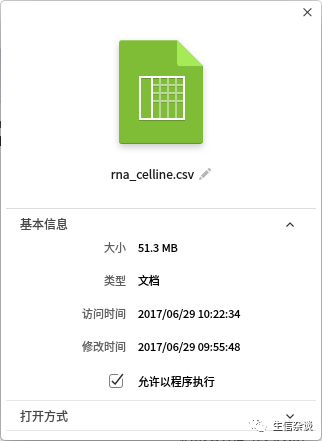
一共包含五列数据,分别是ensembl_id,gene_name, tissue, value,value_type
1.
连接数据库
第一步,先指定一个数据库,新建各种所需的model
import os
from peewee import Model
from peewee import SqliteDatabase
from peewee import CharField, FloatField
db = SqliteDatabase(os.path.join(
os.path.dirname(__file__),
'expression_value.db'),
autocommit=True)
class BaseModel(Model):
u"""基础类,指定数据库和基本数据类型.s"""
tissue = CharField()
ensembl_id = CharField()
gene_name = CharField()
value = FloatField()
class Meta:
u"""指定数据库."""
database = db
class HPA(BaseModel):
u"""存储HPA表达量数据的."""
value_type = CharField(default='TPM')
class Meta:
u"""指定表."""
db_table = 'HPA'
order_by = ('tissue', 'ensembl_id', )
以上一个数据库和一张表就搞定了
2.
插入数据的基础方法
首先自然是读取文件,根据自己的文件格式,自己调整代码
我读取csv的方式如open_csv()所示
class Bulk(object):
u"""测试bulk."""
def __init__(self, csv):
u"""csv."""
self.csv = csv
def open_csv(self):
u"""打开csv文件,然后返回信息."""
with open(self.csv) as reader:
for line in reader:
line = line.rstrip()
#
yield [x.replace('"', '')
for x in line.split(',')]
@staticmethod
def creat_new_table(table):
u"""用来创建table的."""
if not table.table_exists():
table.create_table()
def normal_insert(self):
u"""最普通的insert方式."""
self.creat_new_table(HPA)
for line in self.open_csv():
try:
HPA.create(ensembl_id=line[0],
gene_name=line[1],
tissue=line[2],
value=float(line[4]))
except ValueError:
continue
create()是peewee中最基础的数据插入方式,但同时,也是最慢的,尤其是autocommit=True的情况下
有多慢呢?
这个50+M的csv,我用了两天多才搞定,这两天反正电脑一直在用,就这么浪吧。
3.
插入数据的进阶方法
peewee是支持直接将字典导入数据库的,因此,有dict_to_model和model_to_dict两个类方法,能够实现SQL与Python内置类型以及json的交互,然而,这次我需要用字典,但是用不上这两个方法,感兴趣可以自己查
首先需要对所有的数据进行修饰,将其转化成字典
u"""接上回,想要加快导入的速度,必须使用字典导入的方式,用法如下"""
def construct_dict(self):
u"""按照field: value构成字典"""
keys = ['ensembl_id', 'gene_name',
'tissue', 'value', 'value_type']
for line in self.open_csv():
try:
line[3] = float(line[3])
except ValueError:
continue
yield {key: value for key, value in zip(keys, line)}
constract_dict()返回的结果如下,每一行的数据都转化成一个对应的字典
{
‘ensembl_id’: 'EN*******',
'gene_name': 'TP53',
'tissue': 'liver',
'value': 5,
'value_type': 'TPM'
}
然后,将其导入数据库,根据官方文档的示例修改而来,确实有用,速度快多了















