话说到了基因组时代,通过正向遗传学,找到一个基因,基本就是三个方法:GWAS、bin-map、BSA。无论何种方法,用的基本原理还是我们高中生物课本上学的:
-
基因在染色体上直线排列,
-
染色体会发生重组和交换。
基因组很大,几万个基因,我们很难直接找到影响某个表型的cause基因。今天要讲的就是BSA。
先给大家讲一个故事
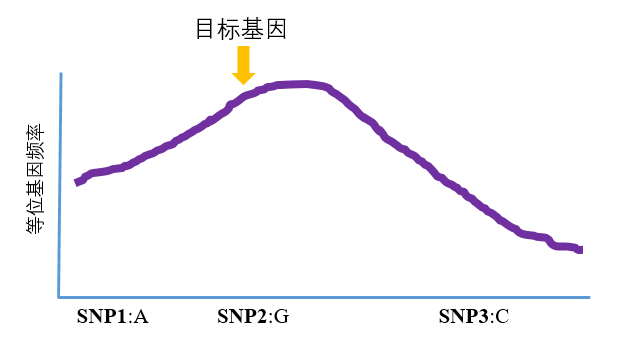
假如我们有两个主角“金角大王”和“银角大王”。金色还是银色,只有一个基因控制。两位大王基因组上,除了我们目标基因不同造成颜色的差异,还有三个SNP,分别为SNP1、2、3(如图所示)。
注意
:原来金色allele和A,G,C在一条染色体上,银色和T,C,G在同一条染色体上。

下面故事发生了:
金角大王和银角大王结婚了,然后生了一群小大王,小大王自由婚配生了一群小小大王…….一群小妖,有金色的也有银色的。有一天,我们抓了一群金色小妖,测个序,看了下每个位点上等位基因频率。发现这个金色小妖群里SNP2上G的等位基因频率很高,SNP1上A次之,SNP3上的C的最低(注意到:开始时候SNP2G,SNP1A,SNP3C都是和金色相随相伴的,即起始等位基因频率都是1)。
那么,如果我们只知道SNP1、2、3的等位基因频率,而不知道金色银色基因在哪的话,我们可以基本判断目标基因在SNP2附近,而不在SNP3附近。
为什么呢?因为越近、越连锁,关联性越强。我们选择这些极端表型的时候,把这个控制表型目标基因的邻居顺便都捞出来了,然后我们可以通过邻居找到当家的。就是这么简单粗暴的大道理。
也就是说,我们可以通过表型选择,把金角小妖混一块,银角小妖混一块,然后扫描全基因组所有位点等位基因频率差异,找到颜色基因所处的位置。
这就是BSA的基本原理:
闲话少说,最近我崇拜的大神,冷泉港的lippman在Cell上灌了一篇封面,两个主要的基因都是通过BSA进行定位,然后用同源基因方法找到的,具体信息自己看文章。文章链接:http://www.cell.com/cell/fulltext/S0092-8674(17)30486-5
为了表示对大神的崇拜之情,我第一时间把文章的信息down下来,进行了重演,以下就是详细过程。
所用软件版本:
-
bwa:0.7.9a-r78
-
samtools:0.1.19-44428cd
-
bcftools:0.1.19-44428cd

实际流程
下载数据和软件安装
根据文章 NCBI SRA的索取号 下载
wget -c ftp://ftp-trace.ncbi.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR527/SRR5274882/SRR5274882.sra
wget -c ftp://ftp-trace.ncbi.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR527/SRR5274881/SRR5274881.sra
wget -c ftp://ftp-trace.ncbi.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR527/SRR5274880/SRR5274880.sra
需要安装软件 SRA tools
wget https://ftp-trace.ncbi.nlm.nih.gov/sra/sdk/2.8.2-1/sratoolkit.2.8.2-1-centos_linux64.tar.gz
tar zxvf sratoolkit.2.8.2-1-centos_linux64.tar.gz
##直接解压缩即可得到可执行文件
### test [ligw@node13 sratoolkit.2.8.2-1-centos_linux64]$ ./bin/fastq-dump -h
NCBI下载的SRA格式转换成fastq格式
/home/ligw/tools/sratoolkit.2.8.2-1-centos_linux64/bin/fastq-dump -F --skip-technical --split-files --defline-qual '+' --defline-seq '@$ac-$si/$ri' --split-3 SRR5274880.sra&
/home/ligw/tools/sratoolkit.2.8.2-1-centos_linux64/bin/fastq-dump -F --skip-technical --split-files --defline-qual '+' --defline-seq '@$ac-$si/$ri' --split-3 SRR5274881.sra&
/home/ligw/tools/sratoolkit.2.8.2-1-centos_linux64/bin/fastq-dump -F --skip-technical --split-files --defline-qual '+' --defline-seq '@$ac-$si/$ri' --split-3 SRR5274882.sra&
参考基因组:
wget ftp://ftp.ensemblgenomes.org/pub/plants/release-35/fasta/solanum_lycopersicum/dna/Solanum_lycopersicum.SL2.50.dna.toplevel.fa.gz
SNP calling pipeline :
### index
bwa index tomato.SL2.50.fa
### mapping
bwa mem -M -t 20 -R "@RG\tID:ejmt\tSM:ejmtpool\tPL:Illumina" tomato.SL2.50.fa SRR5274882_1.fastq SRR5274882_2.fastq > ejmt.sam
### convert sort index remove_duplicate index
samtools view -bS -o ejmt.bam ejmt.sam
samtools sort -m 2G -@ 20 ejmt.bam ejmt.sorted
samtools index ejmt.sorted.bam
### MarkDuplicates
java -Xmx20G -jar /home/ligw/tools/picard.jar MarkDuplicates I=ejmt.sorted.bam O=ejmt.sorted.redup.bam \
METRICS_FILE=ejmt.sort.metrics REMOVE_DUPLICATES=true \
ASSUME_SORTED=true VALIDATION_STRINGENCY=LENIENT
samtools index ejmt.sorted.redup.bam
## vaiant calling
samtools mpileup -DSug -C 50 -Q 20 -q 40 -f tomato.SL2.50.fa ejmt.sorted.redup.bam | bcftools view -cvg - > ejmt.vcf
PS: 野生型池就不重复了
Mapping 基因
shell部分:
### 过滤掉indel
grep -v "##" ejmt.vcf | grep -v "INDEL" > ejmt.snp.vcf
grep -v "##" jmt.vcf | grep -v "INDEL" > jmt.snp.vcf
grep -v "##" wt.vcf | grep -v "INDEL" > wt.snp.vcf
R语言代码:
e.snp 3&e.snp.index$all.reads<80,]
得到染色体window的结果,1Mb大小,100kb step步长
max.pos
得到SNP frequency ratio
wind.bin$E.index=x[2]&e.snp.index$POS<=x[3],]
if(dim(wind.bin.index.E)[1] < 10){return(NA)}
else{return(sum(wind.bin.index.E$alt.no,na.rm=T)/sum(wind.bin.index.E$ref.no,na.rm=T))}})
PS:野生型表型池,代码类似,就不重复了
plot(wind.bin$E.index/wind.bin$W.index~c(1:8146),pch=20,xaxt="n")
##### 主要结果,到此结束。下面的代码是为了画出染色体的边界并进行标注 #######
chr.win.no

原文结果:

注意:
1.细心的同学会发现我的结果虽然与原文类似,但是还是有很大差别的。原因在于原文中定位用到的两个亲本,都不是参考基因组的品种材料,但是野生型和参考基因组类似,我这里为了方便,在统计野生型等位基因频率和突变型等位基因频率时候,用ref reads 代替了wild reads ;用alt reads 代替了mutation reads,仅为演示。
正确的做法
应该是,把P1和P2(就是突变型和野生型)分别测序,call出来SNP,用两个亲本间的SNP做后续分析。至于等位基因频率的计算,在下不才,只用了R的代码,R处理起来还是很慢的,大家也可以用perl python等文本处理能力更强大的代码提取出来有效信息,然后再用R做定位分析和画图。当然语言只是工具,只要明白了原理,知道怎么去抽取有效信息,具体怎么出来相信对大家都不是难事。
2.本文用到的samtools bcftools版本相对比较低,更新到最新版本后,bcftools有了质量过滤选项,根据文章后面提供的信息给出如下代码供参考(具体参数含义,请参考官方文档):
samtools mpileup -R -d 1000000 -t DP,AN -Q 0 -Bug -f tomato.SL2.50.fa ejmt.sorted.redup.bam | bcftools call -mv -O z > ejmt.samtools.raw.vcf
bcftools filter -g 100 -e "MIN(MQ<50)" ejmt.samtools.raw.vcf | bcftools view -m 2 -M 2 -v snps -o ejmt.samtools.filtered.vcf
欢迎大家拍砖,提意见。如果大家觉得写的还可以,也请多鼓励,下次聊聊BSA课题设计中需要注意的问题。



