常见的数据格式
genbank
genbank是美国国立卫生研究院维护的基因序列数据库,汇集并注释了所有公开的核酸以及蛋白质序列。每个记录代表了一个单独的、连续、带有注释的DNA或RNA片段。
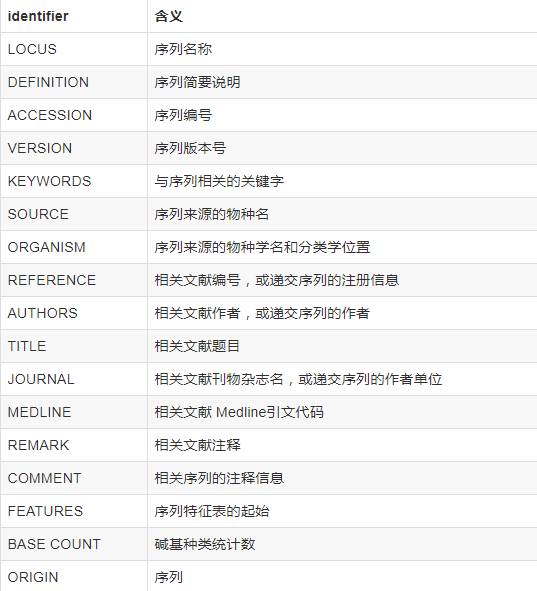
文中关于埃博拉病毒的例子
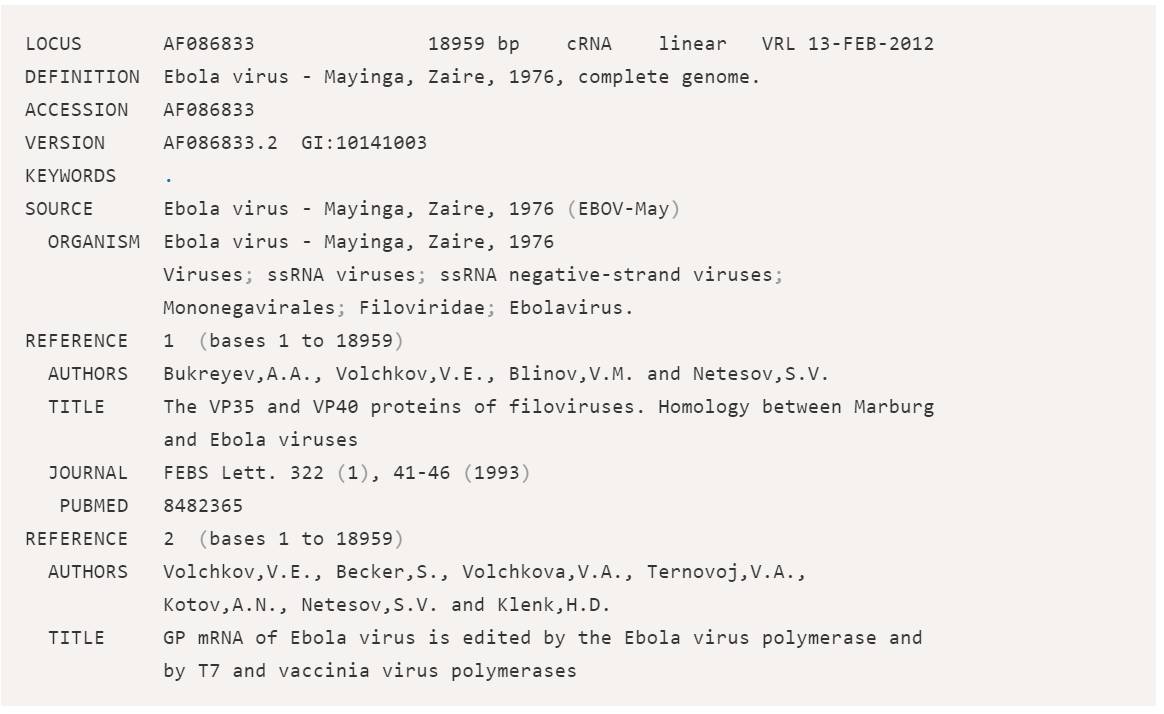
特点:
易读性,便于信息交换
不利于计算机进行数据分析
可通过RefSeq进行格式转化
FASTA格式
在生物信息学中,FASTA格式是一种用于记录核酸序列或肽序列的文本格式,其中的核酸或氨基酸均以单个字母编码呈现。该格式同时还允许在序列之前定义名称和编写注释。这一格式最初由FASTA软件包定义,但现今已是生物信息学领域的一项标准。(来源维基)
格式
FASTA格式中的一条完整序列,包含开头的单行描述行和多行序列数据。描述行行首前置半角大于号(“>”)以和数据行区分。“>”后紧接的内容为该序列的标识符,该行剩余部分则为序列的描述(标识符与描述均非必须)。“>”和标识符之间不应有空格,且建议将单行内容限制在80字符以内。序列的结束以下一条序列的“>”出现为标识。如下为FASTA格式一条序列的示例:
例子
>
gi|31563518|ref|NP_852610.1| microtubule-associated proteins 1A/1B light chain 3A isoform b [Homo sapiens]
MKMRFFSSPCGKAAVDPADRCKEVQQIRDQHPSKIPVIIERYKGEKQLPVLDKTKFLVPDHVNMSELVKI
IRRRLQLNPTQAFFLLVNQHSMVSVSTPIADIYEQEKDEDGFLYMVYASQETFGFIRENE
FASTQ格式
FASTQ格式是一种保存生物序列(通常为核酸序列)及其测序质量得分信息的文本格式。序列与质量得分皆由单个ASCII字符表示。
该格式最初由维尔康姆基金会桑格研究所开发,旨在将FASTA格式序列及其质量数据集成在一起。而目前,FASTQ格式已经成为了保存高通量测序结果的事实标准。(维基百科)
格式
FASTQ文件中,一个序列通常由四行组成:
-第一行以@开头,之后为序列的标识符以及描述信息(与FASTA格式的描述行类似)
-第二行为序列信息
-第三行以+开头,之后可以再次加上序列的标识及描述信息(可选)
-第四行为质量得分信息,与第二行的序列相对应,长度必须与第二行相同
第四行质量分数目前有两种版本,为phred33以及phred64
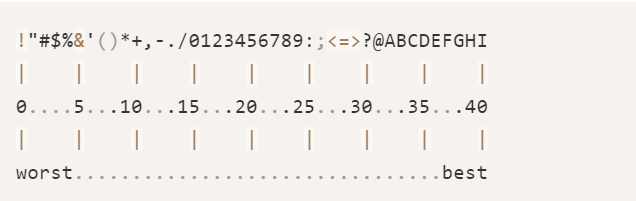 phred33
phred33
 phred64
phred64
常用的数据库
-
NCBI: National Center for Biotechnology Information
-
EMBL: European Molecular Biology Laboratory
-
DDBJ: DNA Data Bank of Japan
-
GenBank
contains all annotated and identified DNA sequence information
-
SRA: Short Read Archive
contains measurements from high throughput sequencing experiments
UniProt: Universal Protein Resource
is the most authoritative repository of protein sequence data.
Protein Data Bank (PDB)
is the major repository of 3D structural information about biological macromolecules (proteins and nucleic acids). PDB contains structures for a spectrum of biomolecules - from small bits of proteins/nucleic acids all the way to complex molecular structures like ribosomes.
数据的获取
Entrez Direct简介
Entrez Direct
(EDirect) provides access to the NCBI's suite of interconnected databases (publication, sequence, structure, gene, variation, expression, etc.) from a UNIX terminal window. Functions take search terms from command-line arguments. Individual operations are combined to build multi-step queries. Record retrieval and formatting normally complete the process.
EDirect also includes an argument-driven function that simplifies the extraction of data from document summaries or other results that are returned in structured XML format. This can eliminate the need for writing custom software to answer ad hoc questions. Queries can move seamlessly between EDirect commands and UNIX utilities or scripts to perform actions that cannot be accomplished entirely within Entrez.(from NCBI)
EDirect工具介绍
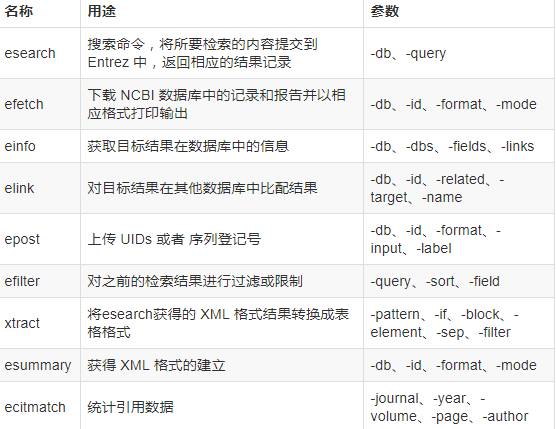
例子:
efetch -db=nuccore -format=gb -id=AF086833 |head
LOCUS AF086833 18959 bp cRNA linear VRL 13-FEB-2012
DEFINITION Ebola virus - Mayinga, Zaire, 1976, complete genome.
ACCESSION AF086833
VERSION AF086833.2
KEYWORDS .
SOURCE Ebola virus - Mayinga, Zaire, 1976 (EBOV-May)
ORGANISM Ebola virus - Mayinga, Zaire, 1976
Viruses; ssRNA viruses; ssRNA negative-strand viruses;
Mononegavirales; Filoviridae; Ebolavirus.
REFERENCE 1 (bases 1 to 18959)
将相同序列存储为genbank格式
efetch -db=nuccore -format=gb -id=AF086833 > AF083833.gb
head AF083833.gb
LOCUS AF086833 18959 bp cRNA linear VRL 13-FEB-2012
DEFINITION Ebola virus - Mayinga, Zaire, 1976, complete genome.
ACCESSION AF086833
VERSION AF086833.2
KEYWORDS .
SOURCE Ebola virus - Mayinga, Zaire, 1976 (EBOV-May)
ORGANISM Ebola virus - Mayinga, Zaire, 1976
Viruses; ssRNA viruses; ssRNA negative-strand viruses;
Mononegavirales; Filoviridae; Ebolavirus.
REFERENCE 1 (bases 1 to 18959)
将相同序列存储为FASTA格式
efetch -db=nuccore -format=fasta -id=AF086833 > AF083833.fa
head AF083833.fa
>
AF086833.2 Ebola virus - Mayinga, Zaire, 1976, complete genome
CGGACACACAAAAAGAAAGAAGAATTTTTAGGATCTTTTGTGTGCGAATAACTATGAGGAAGATTAATAA
TTTTCCTCTCATTGAAATTTATATCGGAATTTAAATTGAAATTGTTACTGTAATCACACCTGGTTTGTTT
CAGAGCCACATCACAAAGATAGAGAACAACCTAGGTCTCCGAAGGGAGCAAGGGCATCAGTGTGCTCAGT
TGAAAATCCCTTGTCAACACCTAGGTCTTATCACATCACAAGTTCCACCTCAGACTCTGCAGGGTGATCC
AACAACCTTAATAGAAACATTATTGTTAAAGGACAGCATTAGTTCACAGTCAAACAAGCAAGATTGAGAA
TTAACCTTGGTTTTGAACTTGAACACTTAGGGGATTGAAGATTCAACAACCCTAAAGCTTGGGGTAAAAC
ATTGGAAATAGTTAAAAGACAAATTGCTCGGAATCACAAAATTCCGAGTATGGATTCTCGTCCTCAGAAA
ATCTGGATGGCGCCGAGTCTCACTGAATCTGACATGGATTACCACAAGATCTTGACAGCAGGTCTGTCCG
TTCAACAGGGGATTGTTCGGCAAAGAGTCATCCCAGTGTATCAAGTAAACAATCTTGAAGAAATTTGCCA
其余例子不一一搬运
Aspera工具介绍
Aspera
提供了大文件高速传输方案,适合于大数据的传输。客服端的使用是免费的。
来源
http://www.chenlianfu.com/?p=2319
安装
wget https://download.asperasoft.com/download/sw/connect/3.7.4/aspera-connect-3.7.4.147727-linux-64.tar.gz
tar -zxvf aspera-connect-3.7.4.147727-linux-64.tar.gz
bash aspera-connect-3.7.4.147727-linux-64.sh
Aspera的用法:
$ ascp [参数] 目标文件 目的地址
Aspera的常用参数:
-T 不进行加密。若不添加此参数,可能会下载不了。
-i string
输入私钥,安装 aspera 后有在目录 ~/.aspera/connect/etc/ 下有几个私钥,使用 linux 服务器的时候一般使用 asperaweb_id_dsa.openssh 文件作为私钥。
--host=string
ftp的host名,NCBI的为ftp-private.ncbi.nlm.nih.gov;EBI的为fasp.sra.ebi.ac.uk。
--user=string
用户名,NCBI的为anonftp,EBI的为era-fasp。
--mode=string
选择模式,上传为 send,下载为 recv。
-l string
设置最大传输速度,比如设置为 200M 则表示最大传输速度为 200m/s。若不设置该参数,则一般可达到10m/s的速度,而设置了,传输速度可以更高。
命令行下载SRA数据(SRR3589956)
ascp -T -i /home/.aspera/connect/etc/asperaweb_id_dsa.openssh [email protected]:sra/sra-instant/reads/ByRun/sra/SRR/SRR358/SRR3589956/SRR3589956.sra ./
浏览器插件下载SRA数据





